Zastosowanie metod PCR i FISH w szybkiej diagnostyce bakteryjnych zakażeń krwi
|
|
|
- Weronika Nowak
- 8 lat temu
- Przeglądów:
Transkrypt
1 PRACA ORYGINALNA Zastosowanie metod PCR i FISH w szybkiej diagnostyce bakteryjnych zakażeń krwi Use of PCR and FISH methods for rapid identification of bacterial bloodstream infections Tomasz Gosiewski, Agata Pietrzyk, Monika Brzychczy-Włoch, Piotr B. Heczko STRESZCZENIE Katedra Mikrobiologii Collegium Medicum Uniwersytetu Jagiellońskiego w Krakowie WSTĘP Celem pracy była ocena możliwości zastosowania metod PCR i FISH w szybkiej diagnostyce bakteryjnych zakażeń krwi. ADRES DO KORESPONDENCJI: Dr n. med. Tomasz Gosiewski Katedra Mikrobiologii Collegium Medicum Uniwersytetu Jagiellońskiego ul. Czysta Kraków tel w 225; fax tgosiews@cm-uj.krakow.pl Ann. Acad. Med. Siles. 2011, 65, 5 6, Copyright Śląski Uniwersytet Medyczny w Katowicach ISSN MATERIAŁ I METODY Analizie poddano 56 próbek: 28 próbek krwi żylnej i 28 próbek podłoży hodowlanych. Wszystkie próbki krwi pochodziły od pacjentów z klinicznymi objawami sepsy, diagnozowanych w Pracowni Diagnostyki Mikrobiologicznej Katedry Mikrobiologii Collegium Medicum Uniwersytetu Jagiellońskiego w Krakowie. Pobrane próbki krwi badano na obecność bakterii, równolegle w systemie hodowli monitorowanej BACTEC oraz za pomocą metod PCR (polymerase chain reaction) i FISH (fluorescent in situ hydridization). Dodatkowo, każdą butelkę z podłożem poddawano analizie za pomocą metody FISH. Izolację DNA prowadzono przy użyciu zestawu QIAamp DNA Blood Mini. W celu usunięcia inhibitorów PCR wprowadzono etap oczyszczania próbek chlorkiem amonu. Do reakcji PCR wykorzystano parę starterów 16SrRNA(+) i 16SrRNA(-) umożliwiającą wykrycie wszystkich gatunków bakterii. W metodzie FISH wykorzystano sondę EUB338 swoistą dla wszystkich gatunków bakterii oraz sondy swoiste dla Enterobacteriaceae (ENT183) i rodzaju Staphylococcus (STA). WYNIKI Odsetek wyników dodatnich dla próbek krwi żylnej wynosił: 71,4%, 28,6% i 10,7% odpowiednio dla PCR, FISH i BACTEC. Różnice między wynikami uzyskanymi za pomocą metod PCR i FISH oraz PCR i BACTEC były statystycznie istotne. W przypadku analizy próbek podłóż hodowlanych, odsetek wyników dodatnich dla metody FISH wynosił 58,3%, natomiast dla metody hodowlanej 33,3%. Różnica ta nie była statystycznie istotna. Czas potrzebny do uzyskania wyników badań prowadzonych za pomocą metod PCR i FISH wynosił około 4 5 godzin. Amplifikacja DNA była możliwa jedynie dla próbek krwi poddanych dodatkowo wstępnej 14
2 PCR I FISH W DIAGNOSTYCE BAKTERIEMII procedurze oczyszczania i przepłukiwania osadu komórkowego. W pozostałych przypadkach obserwowano efekt inhibicji. WNIOSKI Uzyskane wyniki wykazały, że metody PCR i FISH pozwalają na wykrycie obecności bakterii w próbkach pełnej krwi, są bardziej czułe od metody hodowlanej, skracają czas oczekiwania na wynik do kilku godzin, zaś reakcja amplifikacji wymaga zastosowania dodatkowej procedury oczyszczania próbek krwi. SŁOWA KLUCZOWE diagnostyka sepsy, hodowla krwi, PCR, inhibicja PCR, FISH ABSTRACT INTRODUCTION The aim of this study was to evaluate the possibility of applying PCR and FISH methods in rapid diagnostics of bacterial bloodstream infections. MATERIAL AND METHODS 56 samples: 28 venous blood samples and 28 samples of culture media from BACTEC machine after incubation cycle were tested. All blood samples originated from patients with clinical symptoms of sepsis who were diagnosed in the Laboratory of Microbiological Diagnostics at the Chair of Microbiology Medical College Jagiellonian University in Krakow. The blood samples were tested to presence of bacteria in parallel, using PCR, FISH and culture monitored BACTEC system. Additionally, each bottle with medium was analyzed by FISH method. DNA was extracted with QIAamp DNA Blood Mini (Qiagen). To remove PCR inhibitors the step of samples purification with ammonium chloride was added. For PCR reaction a pair of 16SrRNA(+) and 16SrRNA(-) starters which enables detection of all bacteria species was applied. In FISH method a probe specific to all bacteria species EUB338 and probes specific to Enterobacteriaceae (ENT183) and Staphylococcus genus (STA) were used. RESULTS Percentage of positive venous blood samples for PCR, FISH and BACTEC was: 71.4%, 28.6% and 10.7%, respectively. The differences between results obtained by PCR and FISH methods and by PCR and BACTEC were statistically significant. In case of culture media samples analysis a percentage of positive results for FISH was 58.3%, while for culture method 33.3%. This difference was not statistically significant. The time needed to receive a results of samples examination using PCR and FISH methods was about 4 5 hours. DNA amplification was obtainable only for these blood samples which were in addition initially purified, and special cellular sediment washing procedure was used. For unpurified samples inhibition effect was noted. Conclusions Results obtained by us indicated that PCR and FISH methods: allow to detect bacteria in whole blood samples, are much more sensitive than culture method, shorten waiting time for results to few hours and that the use of additional procedure of blood samples purification is needed to receive a positive result of amplification. KEY WORDS diagnostics of sepsis, blood culture, PCR, PCR inhibition, FISH 15
3 WSTĘP Zakażenia wywoływanie przez bakterie i grzyby od zawsze stanowiły poważny problem medyczny. Najgroźniejszym z nich jest zakażenie ogólnoustrojowe, czyli sepsa, określane także mianem posocznicy. Mimo postępów w jej leczeniu, osiągniętych głównie dzięki zastosowaniu antybiotykoterapii oraz wprowadzeniu do praktyki medycznej technologii pozwalających na długotrwałe podtrzymywanie czynności życiowych u pacjentów znajdujących się w stanie krytycznym, nadal nie udaje się utrzymać przy życiu wielu chorych. Paradoksalnie, w miarę rozwoju wiedzy medycznej i wprowadzania do lecznictwa coraz nowszych procedur terapeutycznych, zapadalność na sepsę się zwiększa. Lever i wsp. donoszą, że w USA w ciągu roku zapada na nią 750 tys. osób i jest ona przyczyną ponad 215 tys. zgonów [1]. W Unii Europejskiej z powodu ciężkiej sepsy umiera rocznie 146 tys. chorych. W samej tylko Wielkiej Brytanii śmiertelność z tego powodu waha się w przedziale 30 50/100 tys. w ciągu roku, co plasuje sepsę w czołówce dziesięciu najczęstszych przyczyn zgonów [2]. W krajach rozwiniętych sepsa rozwija się u 2 4/1000 żywo urodzonych dzieci, stając się główną przyczyną ich śmierci [3,4]. W Polsce brak dokładnych danych epidemiologicznych, ale Zieliński i wsp. podają, że w 2005 r. z powodu sepsy nastąpiło 967 zgonów, w tym 43 zgony dzieci [5]. Zwiększająca się śmiertelność z powodu posocznicy jest skutkiem narastającej oporności na antybiotyki, stosowania inwazyjnych metod leczenia oraz starzenia się społeczeństwa. Sepsa jest największym zagrożeniem dla osób z obniżoną odpornością, zwłaszcza kiedy są one długotrwale hospitalizowane na oddziałach intensywnej opieki medycznej. Dotyka ona przede wszystkim pacjentów ze schorzeniami nowotworowymi, poddawanych immunosupresji, chorych oparzonych, osób w podeszłym wieku oraz dzieci [3,6]. W leczeniu zakażeń krwi najważniejszym i najtrudniejszym problemem decydującym o skuteczności terapii, a w konsekwencji o kosztach i czasie hospitalizacji jest skuteczna diagnostyka czynników wywołujących ogólnoustrojową odpowiedź zapalną w przebiegu sepsy. Oznaczenie czynnika etiologicznego pozwala na zastosowanie skutecznej, celowanej antybiotykoterapii. Materiałem poddawanym badaniu diagnostycznemu jest krew pobrana od pacjenta manifestującego objawy kliniczne sepsy, takie jak: tachykardia, bradykardia, podwyższona lub obniżona ciepłota ciała, spadek ciśnienia tętniczego krwi i inne. Do tej pory tzw. złotym standardem diagnostycznym są hodowle krwi prowadzone na specjalnych podłożach, w systemach hodowli automatycznej, np. BACTEC (BectonDickinson). Do zalet tych metod należy ich prostota oraz względnie niski koszt wykonania badania. Słabą stroną metody opartej na hodowli krwi jest natomiast jej czasochłonność, sięgająca nawet 5 dni (do czasu wydania wyniku badania) oraz niska czułość, która powoduje, że jedynie w około 15 20% hodowli udaje się uzyskać wzrost mikroorganizmów [5,7]. Wynika z tego, że w zdecydowanej większości przypadków lekarz może stosować jedynie antybiotykoterapię empiryczną z powodu braku uzyskania wzrostu mikroorganizmów odpowiedzialnych za infekcję. Sytuację dodatkowo pogarsza fakt poddawania pacjentów antybiotykoterapii przed pobraniem próbek krwi na posiew. Chorzy są często leczeni antybiotykami, zanim dochodzi do manifestacji objawów sepsy. Hodowle krwi w takim wypadku są bardzo utrudnione, z uwagi na to, iż znajdują się w niej antybiotyki, które hamują wzrost mikroorganizmów. Aby zwiększyć szansę na wykrycie czynników mikrobiologicznych we krwi, podejmowane są próby oparcia ich detekcji na metodach serologicznych, np. na wykrywaniu lipopolisacharydu (LPS) bakterii Gram-ujemnych czy galaktomannanu grzybów [8,9]. Innym celem molekularnym, który rokuje o wiele większe nadzieje na skuteczną, precyzyjną i szybką diagnostykę zakażeń krwi są kwasy nukleinowe drobnoustrojów będących czynnikami etiologicznymi infekcji. Zarówno DNA, jak i RNA każdego organizmu zawierają unikalne dla niego sekwencje, stanowiące swoisty odcisk palca. Dzięki znajomości tych sekwencji możliwe jest zastosowanie metod biologii molekularnej, takich jak PCR czy hybrydyzacja do oznaczania obecności mikroorganizmów we krwi. W literaturze można znaleźć doniesienia na ten temat, jednak jak dotąd metody hodowlane (mimo ich ograniczeń) są nadal powszechnie stosowane jako podstawowe i najczęściej jedyne badanie mikrobiologiczne krwi. Krew jako materiał do badań mikrobiologicznych stawia największe wyzwania spośród wszystkich materiałów biologicznych. Największą trudność sprawia to, że mikroorganizmy odpowiedzialne za zaka- 16
4 PCR I FISH W DIAGNOSTYCE BAKTERIEMII żenie znajdują się we krwi w bardzo małych ilościach lub też następuje tylko ich okresowy wysiew do krwi. Czułość metod molekularnych znacznie przewyższa czułość metody hodowlanej. Poza tym wcześniejsze zastosowanie antybiotykoterapii nie wpływa na wynik badania, z uwagi na to, że nie ma potrzeby uzyskania wzrostu bakterii czy grzybów na podłożu hodowlanym, a konieczne jest jedynie wykrycie ich sekwencji DNA czy RNA [10]. Jednakże metody biologii molekularnej również napotykają trudności podczas prowadzenia diagnostyki mikrobiologicznej krwi. Krew w swoim składzie zawiera m.in. hem i laktoferynę, które są bardzo silnymi inhibitorami enzymów polimeraz DNA używanych w metodzie PCR [11]. Hem powoduje oddysocjowywanie DNA od polimerazy (rozpad kompleksu enzym substrat), a także blokuje kieszeń katalityczną enzymu. Ponadto krew zawiera w swoim składzie różnego rodzaju enzymy proteolityczne mogące degradować polimerazę DNA oraz nukleolityczne, które degradują łańcuchy DNA i RNA będące właściwym materiałem diagnostycznym. Bardzo ważnym inhibitorem jest także EDTA dodawane do krwi jako antykoagulant, który ma zdolność helatowania jonów magnezu, niezbędnych do optymalnego działania enzymu polimerazy. Większość dostępnych procedur preparatyki krwi nie pozwala na pozbycie się efektu inhibicji polimeraz, co w konsekwencji prowadzi do uzyskania wyniku fałszywie negatywnego [12]. Celem pracy była ocena możliwości wykorzystania procedur biologii molekularnej, opartych na metodzie PCR (polymerase chain reaction) i fluorescencyjnej hybrydyzacji in situ, w skrócie FISH (fluorescent in situ hybridization), które mogłyby znaleźć zastosowanie w szybkiej (trwającej kilka godzin) mikrobiologicznej diagnostyce molekularnej próbek krwi pobranej od pacjentów z klinicznymi objawami sepsy. MATERIAŁ I METODY Analizie poddano 56 próbek: 28 krwi żylnej i 28 podłoży hodowlanych. Wszystkie próbki krwi pochodziły od pacjentów z klinicznymi objawami sepsy (ale jeszcze przed potwierdzeniem mikrobiologicznym), diagnozowanych w Pracowni Diagnostyki Mikrobiologicznej Katedry Mikrobiologii CM UJ w Krakowie. Próbki krwi pobierano natychmiast po wystąpieniu objawów klinicznych sugerujących sepsę, takich jak: wzrost albo obniżenie temperatury ciała (> 38 C lub < 36 C), tachykardia ( 90/ min), przyśpieszenie oddechu ( 20/min) oraz zmiana liczby leukocytów we krwi obwodowej (< 4000/μl albo > /μl lub > 10% niedojrzałych form granulocytów), zgodnie z zaleceniami dotyczącymi diagnostyki sepsy, czyli przede wszystkim w okolicach szczytu gorączki. Badania uzyskały akceptację Komisji Bioetycznej UJ (KBET/94/B/2009). HODOWLA BAKTERII Próbki krwi były podawane rutynowej diagnostyce mikrobiologicznej w systemie hodowli monitorowanej BACTEC 9050 (BectonDickinson) oraz za pomocą metod PCR i FISH. Krew pobraną od pacjentów posiewano do odpowiednich podłoży przeznaczonych do hodowli bakterii tlenowych (Plus Aerobic/F+, BectonDickinson) i beztlenowych (Anaerobic/F, BectonDickinson). W przypadku wyhodowania bakterii próbkę poddawano klasycznej diagnostyce mikrobiologicznej wraz z oznaczeniem antybiotykooporności. Dodatkowo każdą butelkę z podłożem hodowlanym poddawano analizie metodą FISH, niezależnie od tego, czy wynik hodowli w aparacie BACTEC był pozytywny, czy negatywny. Równolegle w celu wykonania badań molekularnych pobierano 2 ml krwi do probówek Vacutainer K 3 E (BectonDickinson). Aby wyznaczyć czułość metod PCR i FISH, pobierano próbki krwi od ochotników, bez klinicznych objawów sepsy lub innej infekcji, do których dodano bakterie E. coli ATCC 25922, uzyskując szereg o malejącym gradiencie gęstości, obejmujący wartości od 10 6 do 10 1 cfu/ /ml (colony forming unit jednostki tworzące kolonie). Następnie każdą próbkę z szeregu rozcieńczeń poddawano badaniu za pomocą metod PCR i FISH, aby wyznaczyć graniczną czułość obu metod. IZOLACJA DNA Z KRWI Całość izolacji DNA przeprowadzano pod nawiewem laminarnym, aby uniknąć kontaminacji. Metoda PCR wymagała wykorzystania izolatu DNA uzyskanego z krwi. Korek probówki Vacutainer opalano nad płomieniem palnika gazowego. Następnie strzykawką za- 17
5 opatrzoną w igłę pobierano 200 μl krwi i izolowano DNA za pomocą zestawu QIAamp DNA Blood Mini Kit (Qiagen). Zastosowano także procedurę izolacji rozszerzoną o etap lizy erytrocytów i usuwania hemoglobiny z próbek. W tym celu pobierano z probówki Vacutainer 200 μl krwi i wlewano ją do 800 μl 0,17 M świeżo przygotowanego lub mrożonego chlorku amonu (ICN Biomedicals). Mieszaninę inkubowano w 37 C w termobloku przez 20 min w celu rozbicia erytrocytów. Następnie próbkę wirowano przez 5 min przy 8500 x g w wirówce. Pipetą odrzucano nadsącz, osad zaś zawieszano w 1 ml jałowej wody i ponownie wirowano w tych samych warunkach. Płukanie i wirowanie powtarzano jeszcze dwukrotnie lub do momentu, aż pelet przybrał barwę bladoróżową. Z otrzymanego osadu, zawierającego leukocyty i ewentualnie komórki bakteryjne, izolowano DNA, wykorzystując zestaw do izolacji DNA QIAamp DNA Blood Mini Kit. Izolację przeprowadzano zgodnie z protokołem załączonym przez producenta zestawu. Analogicznie postępowano z próbkami płynu z butelek po zakończonej inkubacji w aparacie BACTEC Każdy izolat DNA poddawano pomiarowi stężenia oraz czystości uzyskanego kwasu deoksyrybonukleinowego. Zastosowano metodę spektrofotometryczną oraz aparat NanoDrop (Thermo Scientific), za pomocą którego dokonywano pomiaru absorbancji próbki DNA w objętości 2 μl przy długości fali 260 nm i 280 nm. Oprogramowanie komputerowe samodzielnie wyliczało stężenie DNA wyrażone w [ng/μl] oraz czystość próbki jako stosunek wartości absorbancji 260 nm do 280 nm. AMPLIFIKACJA METODĄ PCR Mieszaninę reakcyjną sporządzano w objętości 25 μl. W jej skład wchodziły (wszystkie składniki z firmy DNA Gdańsk): 3,0 mm MgCl 2 ; 1x bufor do polimerazy Delta 2; woda; 0,5U polimerazy Delta 2; 100 μm każdego dntp; po 10 μm starterów [13]: 16S rrna(-)5 GGA CTA CCA GGG TAT CTA AT 3 i 16S rrna(+) 5 AGT TTG ATC CTG GCT CAG 3 ; 1,5 μl matrycy DNA. Przygotowane próbki umieszczono w termocyklerze PTC-200 (BioRad) i amplifikowano zgodnie z profilem termicznym [14]: 95 C przez 3 min; (95 C przez 45 sek; 59,3 C przez 45 sek; 72 C przez 1 min) x 30; 72 C przez 5 min. Następnie próbki poddawano rozdziałowi elektroforetycznemu w 1% agarozie (Prona) przy 5V/cm. Zastosowane startery pozwalały na uzyskanie produktu PCR o długości 798 pz. Kontrolę pozytywną reakcji PCR stanowiło DNA wyizolowane z krwi, do której uprzednio dodano bakterie E. coli ATCC 25922, natomiast kontrolą negatywną było DNA wyizolowane z próbki krwi od pacjenta bez klinicznych objawów sepsy lub innej infekcji. HYBRYDYZACJA IN SITU FISH Z probówki Vacutainer pobierano w komorze laminarnej 200 μl krwi. Następnie próbkę poddawano preparatyce jak do izolacji DNA, do momentu uzyskania bladoróżowego osadu. Analogicznie postępowano z próbkami płynu z butelek po zakończonej inkubacji w aparacie BACTEC Osad zawieszano w 20 μl jałowej, dejonizowanej wody, z czego 10 μl nanoszono pipetą na powierzchnię szkiełka mikroskopowego do hybrydyzacji SuperFrost Plus (Menzel Glaser), aby uzyskać rozmaz o średnicy około 10 mm. Preparat suszono pod nawiewem laminarnym, a następnie zalewano 500 μl roztworu 4% paraformaldehydu (Sigma) i umieszczano na okres 20 min w temperaturze 4 C. Następnie preparat przepłukiwano PBS i zalewano 1 2 ml 96% metanolu (POCh). Całość przetrzymywano pod przykryciem przez 15 min w temperaturze 20 C. Po zakończonym procesie utrwalania metanol odpłukiwano ciepłym (37 C) PBS i nanoszono na preparat 20 μl rozcieńczonego roztworu lizozymu (1 mg/ml) (Sigma) z lizostafiną (0,05 mg/ml) (Sigma). Całość inkubowano przez 5 min w temperaturze 37 C, a następnie dwukrotnie płukano jałową wodą destylowaną. Hybrydyzację przeprowadzano przy użyciu znakowanych fluorochromami na 5` końcach, jednołańcuchowych sond oligonukleotydowych (Genomed) komplementarnych do konserwatywnego fragmentu 16S rrna typowego dla danego rodzaju bakterii. Dla rodzaju Staphylococcus stosowano sondę STA: (CY3-5 TCC TCC ATA TCT CTG CGC 3 ) [15], dla Enterobacteriaceae sondę ENT183: (CY3-5 CTC TTT GGT CTT GCG ACG- 3 ) [16], zaś dla wykrycia wszystkich gatunków bakterii łącznie, sondę EUB338: (FITC-5 GCT GCC TCC CGT AGG AGT 3 -FITC) (17). Ponadto użyto barwnik DAPI (Sigma) wykazujący powinowactwo do DNA komórkowego. 18
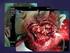
6 PCR I FISH W DIAGNOSTYCE BAKTERIEMII W celu przeprowadzenia hybrydyzacji zmieszano po 5 μl roztworu (50 ng/μl) sond EUB338 oraz STA lub ENT183 z 40 μl buforu do hybrydyzacji: 20 mm Tris HCl ph 7,2 (Sigma); 0,9 M NaCl (Serva); 0,1% SDS (Serva) ogrzanego do temperatury około 50 C. Gotowy roztwór (50 μl) nanoszono na preparat za pomocą pipety automatycznej i całość umieszczano w wilgotnej komorze osłoniętej folią aluminiową na 2 h w temperaturze 50 C. Następnie odpłukiwano sondę ciepłym buforem płuczącym, o takim samym składzie jak bufor do hybrydyzacji, ale bez dodatku 0,1% SDS i dobarwiano preparat roztworem DAPI; 50 μl (5 μl DAPI i 45 μl wody jałowej, destylowanej) przez 3 min w temperaturze pokojowej. Wybarwiony preparat dokładnie płukano jałową, destylowaną wodą i suszono w ciemności. Preparat analizowano za pomocą mikroskopu fluorescencyjnego BX51 (Olympus) wyposażonego w obiektyw immersyjny 100x (Olympus) z lampą UV (Olympus) oraz kamerę F Viev (Olympus). Uzyskany obraz analizowano za pomocą programu komputerowego AnalySYS (Soft Imaging). METODY STATYSTYCZNE Do analizy statystycznej wykorzystano test dokładny Fishera, dwustronny. Metoda PCR pozwoliła wykryć we krwi już 6 x 10 1 cfu/ml bakterii z rodzaju E. coli, natomiast metoda FISH 6 x 10 3 cfu/ml komórek bakteryjnych. Wyniki te uznano za czułość obu metod molekularnych. Zaobserwowano, że w przypadku badania próbek krwi żylnej pobranych od pacjentów odsetek wyników pozytywnych wynosił: 71,4%, 28,6% i 10,7% odpowiednio dla PCR, FISH i BACTEC (ryc. 1). Odsetek dodatnich próbek 100% 80% 60% 40% 20% 0% Odsetek wyników dodatnich w badaniu pe³nej krwi trzema metodami 71,40% 28,50% 10,70% PCR FISH BACTEC Ryc. 1. Zestawienie wyników badania pełnej krwi pochodzącej od pacjentów z klinicznymi objawami sepsy za pomocą trzech metod badawczych. Fig. 1. Comparison of the results of whole blood samples originated from patients with clinical sepsis symptoms by three searching methods. WYNIKI Wszystkie wyizolowane próbki DNA poddano pomiarowi stężenia i czystości. Otrzymane stężenia zawierały się w przedziale 95,5 151,8 ng/μl, zaś czystość izolatów w przedziale 1,65 2,05. Z badanych próbek krwi izolowano DNA metodą bez wstępnego oczyszczania z hemoglobiny oraz równolegle metodą z wstępną lizą erytrocytów w chlorku amonu. Reakcje PCR udało się przeprowadzić jedynie na próbkach DNA wyizolowanych z krwi poddanej wstępnej procedurze oczyszczania chlorkiem amonu i przepłukiwaniem osadu komórkowego. Podobnie nie udało się uzyskać żadnych dodatnich wyników w reakcji PCR przeprowadzonej na izolatach DNA uzyskanych z podłoży po hodowli w aparacie BACTEC. Dzięki oczyszczaniu próbek z hemoglobiny otrzymano dobrej jakości, wyraźny obraz mikroskopowy bez tła autofluorescencji (ryc. 2). Ryc. 2. Zdjęcie z mikroskopu fluorescencyjnego uzyskane metodą FISH na pełnej krwi żylnej. Widoczne bakterie z rodziny Enterobacteriaceae zastosowano sondę ENT183; powiększenie 1000 x. Fig. 2. Fluorescent microscopy photography in FISH method from venous whole blood. Bacteria from Enterobacteriaceae family are visible ENT183 probe was used; magnification 1000 x. 19
7 Stwierdzono statystycznie istotne różnice między wynikami uzyskanymi metodami PCR i FISH oraz PCR i BACTEC (p = 0,003 i p < 0,001). Różnica między FISH i BACTEC nie była statystycznie istotna (p = 0,177). W przypadku próbek podłoży hodowlanych pochodzących z aparatu BACTEC wykazano, że odsetek wyników pozytywnych dla FISH wynosił 58,3%, natomiast dla BACTEC 33,3%, jednak różnica ta także nie była statystycznie istotna (p = 0,414) (ryc. 3). Odsetek dodatnich próbek 100% 80% 60% 40% 20% Odsetek wyników dodatnich w badaniu pod³o a po hodowli krwi za pomoca metody FISH i hodowlanej 0% 58,30% FISH 33,30% BACTEC Ryc. 3. Zestawienie wyników badania podłoża hodowlanego z butelek z aparatu BACTEC po zakończonej inkubacji za pomocą metody FISH i hodowlanej. Fig. 3. Comparison of the results of the BACTEC medium samples after incubation by FISH and culture methods. Czas potrzebny do uzyskania wyniku badania przy użyciu metod PCR i FISH wynosił około 4 5 godzin, zaś dla metody posiewu nawet 72 godziny. DYSKUSJA Współczesna diagnostyka mikrobiologiczna bakteryjnych zakażeń krwi sprowadza się właściwie tylko do hodowli krwi i dalszej diagnostyki wyhodowanego drobnoustroju. Taki sposób postępowania jest podyktowany z jednej strony ograniczonymi środkami finansowymi przeznaczanymi na badania laboratoryjne, zwłaszcza w krajach rozwijających się, z drugiej zaś brakiem alternatywy stanowiącej odpowiednią przeciwwagę dla metod hodowlanych. Do zalet tych metod należą prostota oraz względnie niski koszt wykonania badania. Słabą stroną metody opartej na hodowli krwi jest natomiast jej czasochłonność, sięgająca nawet 5 dni (do czasu wydania wyniku badania), oraz niska czułość, która powoduje, że jedynie w około 15 20% przypadków udaje się uzyskać wzrost mikroorganizmów [5,7]. W naszych badaniach odsetek pozytywnych wyników hodowli krwi wynosił 10,7%. Wydaje się, że dobre rozwiązanie stanowią metody diagnostyczne bazujące na biologii molekularnej, które są z powodzeniem stosowane w laboratoryjnej diagnostyce medycznej, z wyjątkiem molekularnej diagnostyki mikrobiologicznej krwi. Na czoło metod biologii molekularnej użytecznych w mikrobiologicznej diagnostyce medycznej wysuwają się PCR i różnego rodzaju hybrydyzacje kwasów nukleinowych, takie jak FISH. Krew jako materiał diagnostyczny stanowi bardzo duże wyzwanie dla laboratoriów mikrobiologicznych na całym świecie. Wykrywanie drobnoustrojów we krwi jest bardzo trudne z uwagi na ich zwykle niewielką liczbę, ograniczaną dodatkowo przez zastosowane leczenie antybiotykami. Krew jest tkanką bogatą we wszelkiego rodzaju substancje, które mogą skutecznie uniemożliwiać przeprowadzenie badania przy użyciu metod biologii molekularnej, zwłaszcza PCR jest bardzo wrażliwy na wpływ czynników destabilizujących. Najbardziej istotne są substancje blokujące enzym polimerazy DNA, który jest motorem reakcji amplifikacji DNA. Najważniejszy z nich jest hem, ale także immunoglobuliny czy trombocyty [18,19]. Hem powoduje oddysocjowywanie DNA od polimerazy (rozpad kompleksu enzym substrat), a ponadto blokuje kieszeń katalityczną enzymu. Krew zawiera także enzymy proteolityczne mogące degradować polimerazę DNA oraz nukleolityczne, które degradują łańcuchy DNA i RNA będące właściwym materiałem diagnostycznym. Bardzo ważnym inhibitorem jest także EDTA dodawane do krwi jako antykoagulant, mający zdolność helatowania jonów magnezu niezbędnych do preparatyki próbek krwi. W celu wyeliminowania efektu inhibicji reakcji PCR stosuje się różne metody. Zazwyczaj polegają one na bardzo dokładnym przepłukiwaniu próbek czy też ich rozcieńczaniu lub dodawaniu do mieszaniny reakcyjnej albuminy wołowej (BSA), glicerolu czy soli nieorganicznych, jak np. FeCl 3, które stanowią dla inhibitorów cel alternatywny i zmniejszają ich oddziaływanie na polimerazę DNA [20,21,22]. W przypadku metod hybrydyzacji in situ, a zwłaszcza jej fluorescencyjnej odmiany FISH, preparatyka krwi powoduje powstanie bardzo silnego tła autofluorescencji, które jest generowane przez obecną w próbce hemoglobinę. 20
8 PCR I FISH W DIAGNOSTYCE BAKTERIEMII Silne świecenie tła, jak i obecność różnego rodzaju białek powoduje z jednej strony trudności w odróżnieniu fluoryzujących komórek bakteryjnych od tła, a z drugiej niespecyficzną hybrydyzację sond do sekwencji 16S rrna, w konsekwencji zaś zafałszowanie wyników. W niniejszej pracy zastosowano preparatykę próbek krwi, polegającą na bezpośredniej izolacji DNA z krwi. Mimo uzyskania izolatów DNA, nie udało się przeprowadzić reakcji amplifikacji DNA, najprawdopodobniej na skutek inhibicji enzymu polimerazy przez hem. Dopiero rozszerzona metoda izolacji, polegająca na wstępnej lizie erytrocytów za pomocą chlorku amonu, a następnie na usuwaniu hemoglobiny przez dokładne płukanie próbki jałową wodą destylowaną, pozwoliła na zminimalizowanie ilości hemu w próbkach, co w połączeniu z zastosowaniem specjalistycznego zestawu do izolacji DNA skutkowało uzyskaniem czystych izolatów DNA, niepowodujących zahamowania reakcji amplifikacji. Prawdopodobnie z analogicznych powodów nie udało się uzyskać czystych preparatów DNA izolowanego z butelek po hodowli krwi. Stosowane podłoża były syntetyczne i zawierały substancje hamujące działanie antybiotyków, co zwiększało prawdopodobieństwo wyhodowania drobnoustrojów z krwi, jednak przy okazji najprawdopodobniej mogły one zablokować reakcję PCR. Wielu autorów wskazuje na możliwość wykorzystania metody FISH do wykrywania bakterii i grzybów w butelkach z podłożami hodowlanymi po zakończeniu cyklu inkubacji w aparatach hodowli automatycznej, choć nie przyśpiesza to diagnostyki mikrobiologicznej krwi [23,24]. Wydaje się, że dodatkowe badanie metodą FISH butelek uznanych przez system automatycznej hodowli za jałowe w aparacie BACTEC lub w innym systemie do hodowli krwi powinno podnosić poziom wykrywalności bakterii, jednak w naszych badaniach nie wykazano statystycznie istotnej różnicy między liczbą pozytywnych wyników uzyskanych za pomocą FISH (58,3%) i hodowli (33,3%). W przypadku badania próbek pełnej krwi stwierdzono statystycznie znamienną różnicę między liczbą pozytywnych wyników uzyskanych metodą FISH i PCR, choć różnica między metodą hodowlaną a FISH nie była już statystycznie istotna (p = 0,177) (ryc. 1). Najwyższy odsetek wyników pozytywnych badania pełnej krwi uzyskano za pomocą PCR (71,4%), następnie za pomocą FISH (28,6%), a najniższy za pomocą hodowli w aparacie BACTEC (10,7%). Wszystkie dodatnie wyniki z hodowli uzyskały potwierdzenie w metodzie FISH i w PCR, podobnie jak wszystkie dodatnie wyniki z FISH znalazły potwierdzenie w metodzie PCR. Wysoki odsetek wyników pozytywnych uzyskanych w metodzie PCR świadczy o wysokiej jej czułości, istotnie przewyższającej czułość hodowli, ale także czułość metody FISH. Warto podkreślić, że nasze badania koncentrowały się wyłącznie na detekcji bakteryjnych czynników zakażeń krwi i nie uwzględniały grzybów, które też, chociaż rzadziej, mogą być przyczyną sepsy. Możliwe więc, że odsetek wyników dodatnich uzyskanych za pomocą PCR i FISH byłby jeszcze wyższy, gdyby zastosowano dodatkowo startery i sondy ukierunkowane na detekcję tych właśnie drobnoustrojów. Nasza praca potwierdza możliwość wykorzystania metod molekularnych w szybkiej diagnostyce bakteryjnych zakażeń krwi. Uzyskane wyniki są na tyle obiecujące, że skłaniają do rozszerzenia badań i opracowania bardziej precyzyjnych narzędzi umożliwiających równoczesną identyfikację różnych gatunków bakterii we krwi oraz grzybów, a także detekcję mechanizmów oporności na antybiotyki. Dopiero to pozwoli na skuteczną diagnostykę molekularną sepsy. WNIOSKI 1. Metody molekularne PCR i FISH pozwalają na wykrycie komórek bakteryjnych w pełnej krwi. 2. Zastosowanie metod molekularnych skraca czas oczekiwania na wynik do kilku godzin, natomiast w przypadku zautomatyzowanego systemu do hodowli krwi typu BACTEC czas ten sięga nawet kilku dni. 3. Wykorzystanie metody FISH do detekcji bakterii w próbkach podłoży uzyskanych po hodowli krwi zwiększyło odsetek wyników pozytywnych w porównaniu z metodą hodowlaną, ale różnica ta nie była statystycznie istotna. 4. Reakcja amplifikacji (PCR) jest bardzo czuła, lecz jednocześnie wrażliwa na inhibitory utrudniające jej przebieg, co w przypadku diagnostyki sepsy może oznaczać konieczność wprowadzenia dodatkowego etapu oczyszczania próbek. 21
9 Publikacja naukowa finansowana ze środków budżetowych w latach jako projekt badawczy (N N ). PIŚMIENNICTWO 1. Lever A., Mackenzie I., Sepsis: definition, epidemiology and diagnosis. Clinic. Rev. 2008; 335(27): Martin G.S., Mannino D.M., Moss M. The epidemiology of sepsis in the United States from 1979 through N. Engl. J. Med. 2003; 348: Baltimore R.S. Neonatal sepsis: epidemiology and managment. Paediatr. Drugs 2003; 5: Watson R.S., Carcillo J.A. Scope and epidemiology of pediatric sepsis. Pediatr. Crit. Care Med. 2005; 6: Zieliński A., Czarkowski M.P. Choroby zakaźne w Polsce w 2005 roku. Przegl. Epidemiol. 2007; 61: Williams M.D., Braun L.A., Cooper L.M., i wsp. Hospitalized cancer patients with severe sepsis: analysis of incidence, mortality and associated costs of care. Crit. Care 2004; 8: Jamal W., Tamaray G., Pazhoor A., Rotimi V.O. Comparative evaluation of BacT/ALERT 3D and BACTEC systems for the recovery of pathogens causing bloodstream infections. Med. Princ. Pract. 2006; 15: Alexander B.D. Diagnosis of fungal infection: new technologies for the mycology laboratory. Transpl. Infect. Dis. 2004; 4: Romaschin A.D., Harris D.M., Ribeiro M.B. i wsp. A rapid assay of endotoxin in whole blood using autologous neutrophil dependent chemiluminescence. J. Immunol. Methods 1998; 121: Klouche M., Schroder U. Rapid methods for diagnosis of bloodstream infections. Clin. Chem. Lab. Med. 2008; 46: Abu Al-Soud P., Randstrom P. Purification and Characterization of PCR-Inhibitory Components in Blood Cells. J. Clinic. Microbiol. 2001; 39: Akane A., Matsubara K., Nakamura H. Identification of the heme compound copurified with deoxyribonucleic acid (DNA) from bloodstains a major inhibitor of polymerase chain reaction amplification. J. Forensic. Sci. 1994; 37: Hryniewiecki T., Gzyl A., Augustynowicz E., Rawaczyńska I. Reakcja amplifikacji bakteryjnego DNA w diagnostyce infekcyjnego zapalenia wsierdzia. Med. Dosw. Mikrobiol. 2002; 54: Kane T., Johannigman J. The Detection of Microbial DNA in the Blood. Ann. Surg. 1998; 1: Urakawa H., Noble P.A., Fantroussi S.E. Kelly J.J., Stahl D.A. Single-base-pair discrimination of terminal mismatches by using oligonucleotide microarrays and neural network analyses. Appl. Envir. Microbiol. 2002; 68: Friedrich U., Van Langenhove H., Altendorf K. Lipski A. Microbial community and physicochemical analysis of an industrial waste gas biofilter and design of 16S rrna-targeting oligonucleotide probes. Environ. Microbiol. 2003; 5: Amann R.I., Binder B.J., Olson R.J., Chisholm S.W., Devereux R., Stahl D.A. Combination of 16S rrna-targeted oligonucleotide probes with flow cytometry for analyzing mixed microbial populations. Appl. Environ. Microbiol. 1990; 56: Abu Al-Soud W., Rĺdström P. Capacity of Nine Thermostable DNA Polymerases To Mediate DNA Amplification in the Presence of PCR Inhibiting Samples. Appl. Environ. Microbiol. 1998; 64: Banas B., Kost B.P., Goebel F.D. Platelets, a typical source of error in real-time PCR quantification of mitochondrial DNA content in human peripheral blood cells. Eur. J. Med. Res. 2004; 9: Kreader C. Relief of Amplification Inhibition in PCR with Bovine Serum Albumin or T4 Gene 32 Protein. Appl. Environ. Microbiol. 1996; 62: Rĺdström P., Abu Al-Soud P., Lantz P. A sample preparation method which facilitates detection of bacteria in blood cultures by the polymerase chain reaction. J. Microbiol. Methods 1998; 21: Michael D., Braida., Laura M., i wsp. Removal of PCR inhibitors from soil DNA by chemical flocculation. J. Microbiol. Methods 2003; 52: Gescher D.M., Kovacevic D., Schmiedel D. i wsp. Fluorescence in situ hybridisation (FISH) accelerates identification of Gram-positive cocci in positive blood cultures. Int. J. Antimicrob. Agents 2008; 19: Forrest G.N., Mehta S., Weekes E. i wsp. Impact of rapid in situ hybridization testing on coagulase-negative staphylococci positive blood cultures. J. Antimicrob. Chemother. 2006; 58:
Diagnostyka molekularna w OIT
 Diagnostyka molekularna w OIT B A R B A R A A D A M I K K A T E D R A I K L I N I K A A N E S T E Z J O L O G I I I I N T E N S Y W N E J T E R A P I I U N I W E R S Y T E T M E D Y C Z N Y W E W R O C
Diagnostyka molekularna w OIT B A R B A R A A D A M I K K A T E D R A I K L I N I K A A N E S T E Z J O L O G I I I I N T E N S Y W N E J T E R A P I I U N I W E R S Y T E T M E D Y C Z N Y W E W R O C
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA
 Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa IZOLACJA DNA Z HODOWLI KOMÓRKOWEJ.
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa IZOLACJA DNA Z HODOWLI KOMÓRKOWEJ.
Diagnostyka mikrobiologiczna zakażeń krwi Paweł Zwierzewicz
 Diagnostyka mikrobiologiczna zakażeń krwi Paweł Zwierzewicz Diagnostyka mikrobiologiczna sepsy oferta firmy biomerieux Automatyczne analizatory do posiewów krwi Automatyczne analizatory do identyfikacji
Diagnostyka mikrobiologiczna zakażeń krwi Paweł Zwierzewicz Diagnostyka mikrobiologiczna sepsy oferta firmy biomerieux Automatyczne analizatory do posiewów krwi Automatyczne analizatory do identyfikacji
AmpliTest Babesia spp. (PCR)
 AmpliTest Babesia spp. (PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla pierwotniaków z rodzaju Babesia techniką PCR Nr kat.: BAC21-100 Wielkość zestawu: 100 oznaczeń Objętość pojedynczej reakcji:
AmpliTest Babesia spp. (PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla pierwotniaków z rodzaju Babesia techniką PCR Nr kat.: BAC21-100 Wielkość zestawu: 100 oznaczeń Objętość pojedynczej reakcji:
AmpliTest Salmonella spp. (Real Time PCR)
 AmpliTest Salmonella spp. (Real Time PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla bakterii z rodzaju Salmonella techniką Real Time PCR Nr kat.: BAC01-50 Wielkość zestawu: 50 oznaczeń Objętość
AmpliTest Salmonella spp. (Real Time PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla bakterii z rodzaju Salmonella techniką Real Time PCR Nr kat.: BAC01-50 Wielkość zestawu: 50 oznaczeń Objętość
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA
 Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa tel. 22 572 0735, 606448502
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa tel. 22 572 0735, 606448502
PL B1. UNIWERSYTET JAGIELLOŃSKI, Kraków, PL BUP 05/14. TOMASZ GOSIEWSKI, Kraków, PL MONIKA BRZYCHCZY-WŁOCH, Kraków, PL
 PL 219490 B1 RZECZPOSPOLITA POLSKA (12) OPIS PATENTOWY (19) PL (11) 219490 (13) B1 Urząd Patentowy Rzeczypospolitej Polskiej (21) Numer zgłoszenia: 400501 (22) Data zgłoszenia: 24.08.2012 (51) Int.Cl.
PL 219490 B1 RZECZPOSPOLITA POLSKA (12) OPIS PATENTOWY (19) PL (11) 219490 (13) B1 Urząd Patentowy Rzeczypospolitej Polskiej (21) Numer zgłoszenia: 400501 (22) Data zgłoszenia: 24.08.2012 (51) Int.Cl.
PCR bez izolacji testujemy Direct PCR Kits od ThermoFisher Scientific
 PCR bez izolacji testujemy Direct PCR Kits od ThermoFisher Scientific Specjalnie dla Was przetestowaliśmy w naszym laboratorium odczynniki firmy Thermo Scientific umożliwiające przeprowadzanie reakcji
PCR bez izolacji testujemy Direct PCR Kits od ThermoFisher Scientific Specjalnie dla Was przetestowaliśmy w naszym laboratorium odczynniki firmy Thermo Scientific umożliwiające przeprowadzanie reakcji
Zestaw do wykrywania Babesia spp. i Theileria spp. w kleszczach, krwi i hodowlach komórkowych
 Nr kat. PK25N Wersja zestawu: 1.2012 Zestaw do wykrywania spp. i Theileria spp. w kleszczach, krwi i hodowlach komórkowych dwie oddzielne reakcje PCR 2x50 reakcji PCR (50 µl), włączając w to kontrole Zestaw
Nr kat. PK25N Wersja zestawu: 1.2012 Zestaw do wykrywania spp. i Theileria spp. w kleszczach, krwi i hodowlach komórkowych dwie oddzielne reakcje PCR 2x50 reakcji PCR (50 µl), włączając w to kontrole Zestaw
AmpliTest GMO screening-nos (Real Time PCR)
 AmpliTest GMO screening-nos (Real Time PCR) Zestaw do wykrywania sekwencji DNA terminatora NOS techniką Real Time PCR Nr kat.: GMO03-50 GMO03-100 Wielkość zestawu: 50 reakcji 100 reakcji Objętość pojedynczej
AmpliTest GMO screening-nos (Real Time PCR) Zestaw do wykrywania sekwencji DNA terminatora NOS techniką Real Time PCR Nr kat.: GMO03-50 GMO03-100 Wielkość zestawu: 50 reakcji 100 reakcji Objętość pojedynczej
Zastosowanie metody RAPD do różnicowania szczepów bakteryjnych
 Zastosowanie metody RAPD do różnicowania szczepów bakteryjnych Wstęp teoretyczny Technika RAPD (ang. Random Amplification of Polymorphic DNA) opiera się na prostej reakcji PCR, przeprowadzanej na genomowym
Zastosowanie metody RAPD do różnicowania szczepów bakteryjnych Wstęp teoretyczny Technika RAPD (ang. Random Amplification of Polymorphic DNA) opiera się na prostej reakcji PCR, przeprowadzanej na genomowym
PROKALCYTONINA infekcje bakteryjne i sepsa. wprowadzenie
 PROKALCYTONINA infekcje bakteryjne i sepsa wprowadzenie CZĘŚĆ PIERWSZA: Czym jest prokalcytonina? PCT w diagnostyce i monitowaniu sepsy PCT w diagnostyce zapalenia dolnych dróg oddechowych Interpretacje
PROKALCYTONINA infekcje bakteryjne i sepsa wprowadzenie CZĘŚĆ PIERWSZA: Czym jest prokalcytonina? PCT w diagnostyce i monitowaniu sepsy PCT w diagnostyce zapalenia dolnych dróg oddechowych Interpretacje
fix RNA Roztwór do przechowywania i ochrony przed degradacją próbek przeznaczonych do izolacji RNA kat. nr. E0280 Sierpień 2018
 Sierpień 2018 fix RNA Roztwór do przechowywania i ochrony przed degradacją próbek przeznaczonych do izolacji RNA kat. nr. E0280 EURx Ltd. 80-297 Gdansk Poland ul. Przyrodnikow 3, NIP 957-07-05-191 KRS
Sierpień 2018 fix RNA Roztwór do przechowywania i ochrony przed degradacją próbek przeznaczonych do izolacji RNA kat. nr. E0280 EURx Ltd. 80-297 Gdansk Poland ul. Przyrodnikow 3, NIP 957-07-05-191 KRS
Ćwiczenie 2. Identyfikacja płci z wykorzystaniem genu amelogeniny (AMGXY)
 Ćwiczenie 2. Identyfikacja płci z wykorzystaniem genu amelogeniny (AMGXY) Cel ćwiczenia Amplifikacja fragmentu genu amelogeniny, znajdującego się na chromosomach X i Y, jako celu molekularnego przydatnego
Ćwiczenie 2. Identyfikacja płci z wykorzystaniem genu amelogeniny (AMGXY) Cel ćwiczenia Amplifikacja fragmentu genu amelogeniny, znajdującego się na chromosomach X i Y, jako celu molekularnego przydatnego
Zestaw do wykrywania Chlamydia trachomatis w moczu lub w kulturach komórkowych
 Nr kat. PK15 Wersja zestawu: 1.2016 Zestaw do wykrywania w moczu lub w kulturach komórkowych na 50 reakcji PCR (50µl), włączając w to kontrole Detekcja oparta jest na amplifikacji fragmentu genu crp (cysteine
Nr kat. PK15 Wersja zestawu: 1.2016 Zestaw do wykrywania w moczu lub w kulturach komórkowych na 50 reakcji PCR (50µl), włączając w to kontrole Detekcja oparta jest na amplifikacji fragmentu genu crp (cysteine
Zestaw do wykrywania Anaplasma phagocytophilum w kleszczach, krwi i hodowlach komórkowych
 Nr kat. PK24N Wersja zestawu: 1.2016 Zestaw do wykrywania phagocytophilum w kleszczach, krwi i hodowlach komórkowych dwie oddzielne reakcje PCR 2x50 reakcji PCR (50 µl), włączając w to kontrole Detekcja
Nr kat. PK24N Wersja zestawu: 1.2016 Zestaw do wykrywania phagocytophilum w kleszczach, krwi i hodowlach komórkowych dwie oddzielne reakcje PCR 2x50 reakcji PCR (50 µl), włączając w to kontrole Detekcja
AmpliTest Chlamydia/Chlamydophila (Real Time PCR)
 AmpliTest Chlamydia/Chlamydophila (Real Time PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla bakterii z rodzajów Chlamydia i Chlamydophila techniką Real Time PCR Nr kat.: BAC18-50 BAC18-100 Wielkość
AmpliTest Chlamydia/Chlamydophila (Real Time PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla bakterii z rodzajów Chlamydia i Chlamydophila techniką Real Time PCR Nr kat.: BAC18-50 BAC18-100 Wielkość
TaqNovaHS. Polimeraza DNA RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925
 TaqNovaHS RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 TaqNovaHS Polimeraza TaqNovaHS jest mieszaniną termostabilnej polimerazy DNA
TaqNovaHS RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 TaqNovaHS Polimeraza TaqNovaHS jest mieszaniną termostabilnej polimerazy DNA
OFERTA TEMATÓW PRAC DYPLOMOWYCH (MAGISTERSKICH) do zrealizowania w Katedrze MIKROBIOLOGII
 OFERTA TEMATÓW PRAC DYPLOMOWYCH (MAGISTERSKICH) do zrealizowania w Katedrze MIKROBIOLOGII Opracowanie zestawu do wykrywania DNA Aspergillus flavus za pomocą specjalistycznego sprzętu medycznego. Jednym
OFERTA TEMATÓW PRAC DYPLOMOWYCH (MAGISTERSKICH) do zrealizowania w Katedrze MIKROBIOLOGII Opracowanie zestawu do wykrywania DNA Aspergillus flavus za pomocą specjalistycznego sprzętu medycznego. Jednym
Ampli-LAMP Babesia canis
 Novazym Products Zestaw do identyfikacji materiału genetycznego pierwotniaka Babesia canis canis techniką Loop-mediated Isothermal AMPlification (LAMP) Numery katalogowe produktu: AML-Bc-200 AML-Bc-400
Novazym Products Zestaw do identyfikacji materiału genetycznego pierwotniaka Babesia canis canis techniką Loop-mediated Isothermal AMPlification (LAMP) Numery katalogowe produktu: AML-Bc-200 AML-Bc-400
Nowoczesna diagnostyka mikrobiologiczna
 Nowoczesna diagnostyka mikrobiologiczna 1 2 Nowoczesne laboratorium mikrobiologiczne połączenie metod manualnych i automatyzacji Nowoczesne laboratorium mikrobiologiczne To nie tylko sprzęt diagnostyczny,
Nowoczesna diagnostyka mikrobiologiczna 1 2 Nowoczesne laboratorium mikrobiologiczne połączenie metod manualnych i automatyzacji Nowoczesne laboratorium mikrobiologiczne To nie tylko sprzęt diagnostyczny,
Laboratorium Wirusologiczne
 Laboratorium Wirusologiczne Krzysztof Pyrd Pracownia wirusologiczna: Powstanie i wyposażenie całkowicie nowego laboratorium w ramach Zakładu Mikrobiologii WBBiB Profil: laboratorium molekularno-komórkowe
Laboratorium Wirusologiczne Krzysztof Pyrd Pracownia wirusologiczna: Powstanie i wyposażenie całkowicie nowego laboratorium w ramach Zakładu Mikrobiologii WBBiB Profil: laboratorium molekularno-komórkowe
Nazwa i typ aparatu:.. Lp. Opis parametru Kryterium Parametr wymagany
 Załącznik nr 3. Aparat do detekcji wzrostu drobnoustrojów z krwi i płynów ustrojowych Parametry graniczne. Nazwa i typ aparatu:.. Lp. Opis parametru Kryterium Parametr wymagany Parametr oferowany 1 Hodowla
Załącznik nr 3. Aparat do detekcji wzrostu drobnoustrojów z krwi i płynów ustrojowych Parametry graniczne. Nazwa i typ aparatu:.. Lp. Opis parametru Kryterium Parametr wymagany Parametr oferowany 1 Hodowla
PL B1. UNIWERSYTET PRZYRODNICZY W LUBLINIE, Lublin, PL BUP 26/11
 PL 214501 B1 RZECZPOSPOLITA POLSKA (12) OPIS PATENTOWY (19) PL (11) 214501 (13) B1 (21) Numer zgłoszenia: 391458 (51) Int.Cl. C12Q 1/68 (2006.01) C12N 15/29 (2006.01) Urząd Patentowy Rzeczypospolitej Polskiej
PL 214501 B1 RZECZPOSPOLITA POLSKA (12) OPIS PATENTOWY (19) PL (11) 214501 (13) B1 (21) Numer zgłoszenia: 391458 (51) Int.Cl. C12Q 1/68 (2006.01) C12N 15/29 (2006.01) Urząd Patentowy Rzeczypospolitej Polskiej
TaqNova-RED. Polimeraza DNA RP20R, RP100R
 TaqNova-RED Polimeraza DNA RP20R, RP100R RP20R, RP100R TaqNova-RED Polimeraza DNA Rekombinowana termostabilna polimeraza DNA Taq zawierająca czerwony barwnik, izolowana z Thermus aquaticus, o przybliżonej
TaqNova-RED Polimeraza DNA RP20R, RP100R RP20R, RP100R TaqNova-RED Polimeraza DNA Rekombinowana termostabilna polimeraza DNA Taq zawierająca czerwony barwnik, izolowana z Thermus aquaticus, o przybliżonej
Opis przedmiotu zamówienia wraz z wymaganiami technicznymi i zestawieniem parametrów
 Załącznik nr 1 do SIWZ Nazwa i adres Wykonawcy Opis przedmiotu zamówienia wraz z wymaganiami technicznymi i zestawieniem parametrów Przedmiot zamówienia; automatyczny system do diagnostyki molekularnej:
Załącznik nr 1 do SIWZ Nazwa i adres Wykonawcy Opis przedmiotu zamówienia wraz z wymaganiami technicznymi i zestawieniem parametrów Przedmiot zamówienia; automatyczny system do diagnostyki molekularnej:
PROBLEMY TERAPEUTYCZNE WTÓRNYCH ZAKAŻEŃ KRWI POWODOWANE PRZEZ PAŁECZKI Enterobacterales W PRAKTYCE ODDZIAŁÓW ZABIEGOWYCH I ZACHOWAWCZYCH
 PROBLEMY TERAPEUTYCZNE WTÓRNYCH ZAKAŻEŃ KRWI POWODOWANE PRZEZ PAŁECZKI Enterobacterales W PRAKTYCE ODDZIAŁÓW ZABIEGOWYCH I ZACHOWAWCZYCH DOROTA ROMANISZYN KATEDRA MIKROBIOLOGII UJCM KRAKÓW Zakażenie krwi
PROBLEMY TERAPEUTYCZNE WTÓRNYCH ZAKAŻEŃ KRWI POWODOWANE PRZEZ PAŁECZKI Enterobacterales W PRAKTYCE ODDZIAŁÓW ZABIEGOWYCH I ZACHOWAWCZYCH DOROTA ROMANISZYN KATEDRA MIKROBIOLOGII UJCM KRAKÓW Zakażenie krwi
Genomic Maxi AX zestaw do izolacji genomowego DNA wersja 0616
 Genomic Maxi AX zestaw do izolacji genomowego DNA wersja 0616 10 izolacji Nr kat. 995-10 Pojemność kolumny do oczyszczania DNA wynosi 500 µg 1 Skład zestawu Składnik Ilość Temp. Przechowywania Kolumny
Genomic Maxi AX zestaw do izolacji genomowego DNA wersja 0616 10 izolacji Nr kat. 995-10 Pojemność kolumny do oczyszczania DNA wynosi 500 µg 1 Skład zestawu Składnik Ilość Temp. Przechowywania Kolumny
RT31-020, RT , MgCl 2. , random heksamerów X 6
 RT31-020, RT31-100 RT31-020, RT31-100 Zestaw TRANSCRIPTME RNA zawiera wszystkie niezbędne składniki do przeprowadzenia syntezy pierwszej nici cdna na matrycy mrna lub całkowitego RNA. Uzyskany jednoniciowy
RT31-020, RT31-100 RT31-020, RT31-100 Zestaw TRANSCRIPTME RNA zawiera wszystkie niezbędne składniki do przeprowadzenia syntezy pierwszej nici cdna na matrycy mrna lub całkowitego RNA. Uzyskany jednoniciowy
Elżbieta Arłukowicz Streszczenie rozprawy doktorskiej
 Elżbieta Arłukowicz Streszczenie rozprawy doktorskiej Analiza zmienności ilościowej i jakościowej tlenowej flory bakteryjnej izolowanej z ran przewlekłych kończyn dolnych w trakcie leczenia tlenem hiperbarycznym
Elżbieta Arłukowicz Streszczenie rozprawy doktorskiej Analiza zmienności ilościowej i jakościowej tlenowej flory bakteryjnej izolowanej z ran przewlekłych kończyn dolnych w trakcie leczenia tlenem hiperbarycznym
ĆWICZENIE 1 i 2 Modyfikacja geu wołowej beta-laktoglobuliny przy użyciu metody Overlap Extension PCR (wydłużania nakładających się odcinków)
 ĆWICZENIE 1 i 2 Modyfikacja geu wołowej beta-laktoglobuliny przy użyciu metody Overlap Extension PCR (wydłużania nakładających się odcinków) Celem ćwiczenia jest wprowadzenie mutacji punktowej do genu
ĆWICZENIE 1 i 2 Modyfikacja geu wołowej beta-laktoglobuliny przy użyciu metody Overlap Extension PCR (wydłużania nakładających się odcinków) Celem ćwiczenia jest wprowadzenie mutacji punktowej do genu
Ćwiczenie numer 6. Analiza próbek spożywczych na obecność markerów GMO
 Ćwiczenie numer 6 Analiza próbek spożywczych na obecność markerów GMO 1. Informacje wstępne -screening GMO -metoda CTAB -qpcr 2. Izolacja DNA z soi metodą CTAB 3. Oznaczenie ilościowe i jakościowe DNA
Ćwiczenie numer 6 Analiza próbek spożywczych na obecność markerów GMO 1. Informacje wstępne -screening GMO -metoda CTAB -qpcr 2. Izolacja DNA z soi metodą CTAB 3. Oznaczenie ilościowe i jakościowe DNA
AmpliTest Panel odkleszczowy (Real Time PCR)
 AmpliTest Panel odkleszczowy (Real Time PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla bakterii Borrelia burgdorferi, Anaplasma, Ehrlichia oraz pierwotniaków rodzaju Babesia (B. canis, B. gibsoni,
AmpliTest Panel odkleszczowy (Real Time PCR) Zestaw do wykrywania sekwencji DNA specyficznych dla bakterii Borrelia burgdorferi, Anaplasma, Ehrlichia oraz pierwotniaków rodzaju Babesia (B. canis, B. gibsoni,
Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV
 Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV Cel ćwiczenia Określenie podatności na zakażenie wirusem HIV poprzez detekcję homo lub heterozygotyczności
Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV Cel ćwiczenia Określenie podatności na zakażenie wirusem HIV poprzez detekcję homo lub heterozygotyczności
PathogenFree DNA Isolation Kit Zestaw do izolacji DNA Instrukcja użytkownika
 PathogenFree DNA Isolation Kit Zestaw do izolacji DNA Instrukcja użytkownika Spis treści 1. Zawartość 2 1.1 Składniki zestawu 2 2. Opis produktu 2 2.1 Założenia metody 2 2.2 Instrukcja 2 2.3 Specyfikacja
PathogenFree DNA Isolation Kit Zestaw do izolacji DNA Instrukcja użytkownika Spis treści 1. Zawartość 2 1.1 Składniki zestawu 2 2. Opis produktu 2 2.1 Założenia metody 2 2.2 Instrukcja 2 2.3 Specyfikacja
Genomic Mini AX Bacteria+ Spin
 Genomic Mini AX Bacteria+ Spin Zestaw o zwiększonej wydajności do izolacji genomowego DNA z bakterii Gram-dodatnich. wersja 1017 100 izolacji Nr kat. 060-100MS Pojemność kolumny do oczyszczania DNA wynosi
Genomic Mini AX Bacteria+ Spin Zestaw o zwiększonej wydajności do izolacji genomowego DNA z bakterii Gram-dodatnich. wersja 1017 100 izolacji Nr kat. 060-100MS Pojemność kolumny do oczyszczania DNA wynosi
Instrukcje do ćwiczeń oraz zakres materiału realizowanego na wykładach z przedmiotu Mikrobiologia na kierunku chemia kosmetyczna
 1 Zakład Mikrobiologii UJK Instrukcje do ćwiczeń oraz zakres materiału realizowanego na wykładach z przedmiotu Mikrobiologia na kierunku chemia kosmetyczna 2 Zakład Mikrobiologii UJK Zakres materiału (zagadnienia)
1 Zakład Mikrobiologii UJK Instrukcje do ćwiczeń oraz zakres materiału realizowanego na wykładach z przedmiotu Mikrobiologia na kierunku chemia kosmetyczna 2 Zakład Mikrobiologii UJK Zakres materiału (zagadnienia)
Novabeads Food DNA Kit
 Novabeads Food DNA Kit Novabeads Food DNA Kit jest nowej generacji narzędziem w technikach biologii molekularnej, umożliwiającym izolację DNA z produktów spożywczych wysoko przetworzonych. Metoda oparta
Novabeads Food DNA Kit Novabeads Food DNA Kit jest nowej generacji narzędziem w technikach biologii molekularnej, umożliwiającym izolację DNA z produktów spożywczych wysoko przetworzonych. Metoda oparta
Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu
 Ćwiczenie 4 Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu Wstęp CYP2D6 kodowany przez gen występujący w co najmniej w 78 allelicznych formach związanych ze zmniejszoną
Ćwiczenie 4 Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu Wstęp CYP2D6 kodowany przez gen występujący w co najmniej w 78 allelicznych formach związanych ze zmniejszoną
Anna Durka. Opiekun pracy: Dr n. med. Waldemar Machała
 Anna Durka Zastosowanie aktywowanego białka C (Xigris) u pacjentów leczonych z powodu ciężkiej sepsy w II Zakladzie Anestezjologii i Intensywnej Terapii USK nr 2 im. WAM w Łodzi. Opiekun pracy: Dr n. med.
Anna Durka Zastosowanie aktywowanego białka C (Xigris) u pacjentów leczonych z powodu ciężkiej sepsy w II Zakladzie Anestezjologii i Intensywnej Terapii USK nr 2 im. WAM w Łodzi. Opiekun pracy: Dr n. med.
Ampli-LAMP Salmonella species
 Novazym Products Novazym Polska ul. Żywokostowa 23, 61-680 Poznań, tel. +48 0 61 610 39 10, fax +48 0 61 610 39 11, email info@novazym.com Zestaw do identyfikacji materiału genetycznego bakterii z rodzaju
Novazym Products Novazym Polska ul. Żywokostowa 23, 61-680 Poznań, tel. +48 0 61 610 39 10, fax +48 0 61 610 39 11, email info@novazym.com Zestaw do identyfikacji materiału genetycznego bakterii z rodzaju
Zestaw do izolacji genomowego DNA z bakterii
 Nr kat. EM02 Wersja: 1.2018 Zestaw do izolacji genomowego DNA z bakterii EXTRACTME jest zarejestrowanym znakiem towarowym BLIRT S.A. www.blirt.eu Nr kat. EM02 I. PRZEZNACZENIE ZESTAWU Zestaw EXTRACTME
Nr kat. EM02 Wersja: 1.2018 Zestaw do izolacji genomowego DNA z bakterii EXTRACTME jest zarejestrowanym znakiem towarowym BLIRT S.A. www.blirt.eu Nr kat. EM02 I. PRZEZNACZENIE ZESTAWU Zestaw EXTRACTME
Rekomendacje diagnostyczne inwazyjnych zakażeń bakteryjnych nabytych poza szpitalem M. Kadłubowski, A. Skoczyńska, W. Hryniewicz, KOROUN, NIL, 2009
 Opracowanie: Marcin Kadłubowski, Anna Skoczyńska, Waleria Hryniewicz Krajowy Ośrodek Referencyjny ds. Diagnostyki Bakteryjnych Zakażeń Ośrodkowego Układu Nerwowego (KOROUN) Zakład Epidemiologii i Mikrobiologii
Opracowanie: Marcin Kadłubowski, Anna Skoczyńska, Waleria Hryniewicz Krajowy Ośrodek Referencyjny ds. Diagnostyki Bakteryjnych Zakażeń Ośrodkowego Układu Nerwowego (KOROUN) Zakład Epidemiologii i Mikrobiologii
1. Zamawiający: 2. Opis przedmiotu zamówienia:
 Zapytanie ofertowe na dostawę analizatora wraz z oprogramowaniem do automatycznej izolacji, amplifikacji i detekcji sekwencji DNA wirusów brodawczaka ludzkiego typu wysokiego ryzyka (hr-hpv) metodą PCR
Zapytanie ofertowe na dostawę analizatora wraz z oprogramowaniem do automatycznej izolacji, amplifikacji i detekcji sekwencji DNA wirusów brodawczaka ludzkiego typu wysokiego ryzyka (hr-hpv) metodą PCR
Diagnostyka wirusologiczna w praktyce klinicznej
 Diagnostyka wirusologiczna w praktyce klinicznej Ponad 60% zakażeń w praktyce klinicznej jest wywołana przez wirusy. Rodzaj i jakość materiału diagnostycznego (transport!) oraz interpretacja wyników badań
Diagnostyka wirusologiczna w praktyce klinicznej Ponad 60% zakażeń w praktyce klinicznej jest wywołana przez wirusy. Rodzaj i jakość materiału diagnostycznego (transport!) oraz interpretacja wyników badań
MATERIAŁY SZKOLENIOWE DLA ZAKŁADÓW HIGIENY WETERYNARYJNEJ W ZAKRESIE LABORATORYJNEJ DIAGNOSTYKI AFRYKAŃSKIEGO POMORU ŚWIŃ
 MATERIAŁY SZKOLENIOWE DLA ZAKŁADÓW HIGIENY WETERYNARYJNEJ W ZAKRESIE LABORATORYJNEJ DIAGNOSTYKI AFRYKAŃSKIEGO POMORU ŚWIŃ Puławy 2013 Opracowanie: Prof. dr hab. Iwona Markowska-Daniel, mgr inż. Kinga Urbaniak,
MATERIAŁY SZKOLENIOWE DLA ZAKŁADÓW HIGIENY WETERYNARYJNEJ W ZAKRESIE LABORATORYJNEJ DIAGNOSTYKI AFRYKAŃSKIEGO POMORU ŚWIŃ Puławy 2013 Opracowanie: Prof. dr hab. Iwona Markowska-Daniel, mgr inż. Kinga Urbaniak,
Genomic Midi AX. 20 izolacji
 Genomic Midi AX Uniwersalny zestaw o zwiększonej wydajności do izolacji genomowego DNA z różnych materiałów. Procedura z precypitacją DNA. wersja 0517 20 izolacji Nr kat. 895-20 Pojemność kolumny do oczyszczania
Genomic Midi AX Uniwersalny zestaw o zwiększonej wydajności do izolacji genomowego DNA z różnych materiałów. Procedura z precypitacją DNA. wersja 0517 20 izolacji Nr kat. 895-20 Pojemność kolumny do oczyszczania
HARMONOGRAM ZAJĘĆ dla studentów Uniwersyteckiego Centrum Medycyny Weterynaryjnej UJ-UR Mikrobiologia weterynaryjna II rok 2016/2017 semestr letni
 HARMONOGRAM ZAJĘĆ dla studentów Uniwersyteckiego Centrum Medycyny Weterynaryjnej UJ-UR Mikrobiologia weterynaryjna II rok 2016/2017 semestr letni Seminaria sala wykładowa Katedry Mikrobiologii (II piętro),
HARMONOGRAM ZAJĘĆ dla studentów Uniwersyteckiego Centrum Medycyny Weterynaryjnej UJ-UR Mikrobiologia weterynaryjna II rok 2016/2017 semestr letni Seminaria sala wykładowa Katedry Mikrobiologii (II piętro),
Instrukcje do ćwiczeń oraz zakres materiału realizowanego na wykładach z przedmiotu Inżynieria bioprocesowa na kierunku biotechnologia
 1 Zakład Mikrobiologii UJK Instrukcje do ćwiczeń oraz zakres materiału realizowanego na wykładach z przedmiotu Inżynieria bioprocesowa na kierunku biotechnologia 2 Zakład Mikrobiologii UJK Zakres materiału
1 Zakład Mikrobiologii UJK Instrukcje do ćwiczeń oraz zakres materiału realizowanego na wykładach z przedmiotu Inżynieria bioprocesowa na kierunku biotechnologia 2 Zakład Mikrobiologii UJK Zakres materiału
Zestawy do izolacji DNA i RNA
 Syngen kolumienki.pl Gotowe zestawy do izolacji i oczyszczania kwasów nukleinowych Zestawy do izolacji DNA i RNA Katalog produktów 2012-13 kolumienki.pl Syngen Biotech Sp. z o.o., 54-116 Wrocław, ul. Ostródzka
Syngen kolumienki.pl Gotowe zestawy do izolacji i oczyszczania kwasów nukleinowych Zestawy do izolacji DNA i RNA Katalog produktów 2012-13 kolumienki.pl Syngen Biotech Sp. z o.o., 54-116 Wrocław, ul. Ostródzka
Prowadzący: dr Lidia Boss znacznik fluorescencyjny (np. SYBR Green II)
 Uniwersytet Gdański, Wydział Biologii Biologia i Biologia Medyczna II rok Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią ============================================================================
Uniwersytet Gdański, Wydział Biologii Biologia i Biologia Medyczna II rok Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią ============================================================================
ROCZN. PZH, 1998, 49, 73-86
 ROCZN. PZH, 1998, 49, 73-86 74 wane narzędzia i sprzęt medyczny. Przeniesienie grzybów Candida m oże nastąpić przez ręce personelu, bieliznę, pościel, sprzęt do pielęgnacji i leczenia. C elem pracy było
ROCZN. PZH, 1998, 49, 73-86 74 wane narzędzia i sprzęt medyczny. Przeniesienie grzybów Candida m oże nastąpić przez ręce personelu, bieliznę, pościel, sprzęt do pielęgnacji i leczenia. C elem pracy było
10) istotne kliniczne dane pacjenta, w szczególności: rozpoznanie, występujące czynniki ryzyka zakażenia, w tym wcześniejsza antybiotykoterapia,
 Załącznik nr 2 Standardy jakości w zakresie mikrobiologicznych badań laboratoryjnych, w tym badań technikami biologii molekularnej, oceny ich jakości i wartości diagnostycznej oraz laboratoryjnej interpretacji
Załącznik nr 2 Standardy jakości w zakresie mikrobiologicznych badań laboratoryjnych, w tym badań technikami biologii molekularnej, oceny ich jakości i wartości diagnostycznej oraz laboratoryjnej interpretacji
Genomic Maxi AX Direct
 Genomic Maxi AX Direct Uniwersalny zestaw o zwiększonej wydajności do izolacji genomowego DNA z różnych materiałów. Procedura bez etapu precypitacji. wersja 0517 10 izolacji Nr kat. 995-10D Pojemność kolumny
Genomic Maxi AX Direct Uniwersalny zestaw o zwiększonej wydajności do izolacji genomowego DNA z różnych materiałów. Procedura bez etapu precypitacji. wersja 0517 10 izolacji Nr kat. 995-10D Pojemność kolumny
Powodzenie reakcji PCR wymaga właściwego doboru szeregu parametrów:
 Powodzenie reakcji PCR wymaga właściwego doboru szeregu parametrów: dobór warunków samej reakcji PCR (temperatury, czas trwania cykli, ilości cykli itp.) dobór odpowiednich starterów do reakcji amplifikacji
Powodzenie reakcji PCR wymaga właściwego doboru szeregu parametrów: dobór warunków samej reakcji PCR (temperatury, czas trwania cykli, ilości cykli itp.) dobór odpowiednich starterów do reakcji amplifikacji
FLUORESCENCYJNA HYBRYDYZACJA IN SITU W DETEKCJI INFEKCYJNYCH POWIKŁAŃ PRZEWLEKŁEJ STYMULACJI SERCA BADANIA PILOTAŻOWE
 PRACA ORYGINALNA FLUORESCENCYJNA HYBRYDYZACJA IN SITU W DETEKCJI INFEKCYJNYCH POWIKŁAŃ PRZEWLEKŁEJ STYMULACJI SERCA BADANIA PILOTAŻOWE FLUORESCENT IN SITU HYBRIDIZATION IN THE DETECTION OF INFECTIOUSCOMPLICATIONS
PRACA ORYGINALNA FLUORESCENCYJNA HYBRYDYZACJA IN SITU W DETEKCJI INFEKCYJNYCH POWIKŁAŃ PRZEWLEKŁEJ STYMULACJI SERCA BADANIA PILOTAŻOWE FLUORESCENT IN SITU HYBRIDIZATION IN THE DETECTION OF INFECTIOUSCOMPLICATIONS
Techniki molekularne ćw. 1 1 z 6
 Techniki molekularne ćw. 1 1 z 6 Instrukcja do ćwiczeń Nr 1. Temat: Izolacja całkowitego DNA z tkanki ssaczej metodą wiązania DNA do kolumny krzemionkowej oraz spektrofotometryczna ocena jego czystości
Techniki molekularne ćw. 1 1 z 6 Instrukcja do ćwiczeń Nr 1. Temat: Izolacja całkowitego DNA z tkanki ssaczej metodą wiązania DNA do kolumny krzemionkowej oraz spektrofotometryczna ocena jego czystości
ZRÓŻNICOW ANIE GENETYCZNE SZCZEPÓW DROŻDŻY FERM ENTUJĄCYCH KSYLOZĘ
 ŻYW NO ŚĆ 3(20)Supl 1999 JOANNA CHMIELEWSKA, JOANNA KAWA-RYGIELSKA ZRÓŻNICOW ANIE GENETYCZNE SZCZEPÓW DROŻDŻY FERM ENTUJĄCYCH KSYLOZĘ S t r e s z c z e n i e W pracy podjęto próbę wykorzystania metody
ŻYW NO ŚĆ 3(20)Supl 1999 JOANNA CHMIELEWSKA, JOANNA KAWA-RYGIELSKA ZRÓŻNICOW ANIE GENETYCZNE SZCZEPÓW DROŻDŻY FERM ENTUJĄCYCH KSYLOZĘ S t r e s z c z e n i e W pracy podjęto próbę wykorzystania metody
Zakład Mikrobiologii Klinicznej [1]
![Zakład Mikrobiologii Klinicznej [1] Zakład Mikrobiologii Klinicznej [1]](/thumbs/73/69154378.jpg) Zakład Mikrobiologii Klinicznej [1] Dane kontaktowe: Tel. 41 36 74 710, 41 36 74 712 Kierownik: dr n. med. Bonita Durnaś, specjalista mikrobiologii Personel/Kadra: Diagności laboratoryjni: mgr Dorota Żółcińska
Zakład Mikrobiologii Klinicznej [1] Dane kontaktowe: Tel. 41 36 74 710, 41 36 74 712 Kierownik: dr n. med. Bonita Durnaś, specjalista mikrobiologii Personel/Kadra: Diagności laboratoryjni: mgr Dorota Żółcińska
Genomic Midi AX Direct zestaw do izolacji genomowego DNA (procedura bez precypitacji) wersja 1215
 Genomic Midi AX Direct zestaw do izolacji genomowego DNA (procedura bez precypitacji) wersja 1215 20 izolacji Nr kat. 895-20D Pojemność kolumny do oczyszczania DNA wynosi 100 µg 1 Skład zestawu Składnik
Genomic Midi AX Direct zestaw do izolacji genomowego DNA (procedura bez precypitacji) wersja 1215 20 izolacji Nr kat. 895-20D Pojemność kolumny do oczyszczania DNA wynosi 100 µg 1 Skład zestawu Składnik
RAPORT Z BADAŃ REALIZOWANYCH W RAMACH OCENY STĘŻENIA BIOAEROZOLU ZANIECZYSZCZAJĄCEGO POWIETRZE NA PODSTAWIE LICZEBNOŚCI WYBRANYCH GRUP DROBNOUSTROJÓW
 Mysłowice, 22.03.2016 r. RAPORT Z BADAŃ REALIZOWANYCH W RAMACH OCENY STĘŻENIA BIOAEROZOLU ZANIECZYSZCZAJĄCEGO POWIETRZE NA PODSTAWIE LICZEBNOŚCI WYBRANYCH GRUP DROBNOUSTROJÓW Zleceniodawca: Stowarzyszenie
Mysłowice, 22.03.2016 r. RAPORT Z BADAŃ REALIZOWANYCH W RAMACH OCENY STĘŻENIA BIOAEROZOLU ZANIECZYSZCZAJĄCEGO POWIETRZE NA PODSTAWIE LICZEBNOŚCI WYBRANYCH GRUP DROBNOUSTROJÓW Zleceniodawca: Stowarzyszenie
Zestaw dydaktyczny. genotypowanie szczepów 5taphy/ococcus aureus metodą PCR-RFLP
 2014-07-17 Zestaw dydaktyczny EasyGenotyping PCR-RFLP - S. aureus genotypowanie szczepów 5taphy/ococcus aureus metodą PCR-RFLP Celem ćwiczenia jest przeprowadzenie typowania genetycznego dostarczonych
2014-07-17 Zestaw dydaktyczny EasyGenotyping PCR-RFLP - S. aureus genotypowanie szczepów 5taphy/ococcus aureus metodą PCR-RFLP Celem ćwiczenia jest przeprowadzenie typowania genetycznego dostarczonych
Analiza genetyczna w niepowodzeniach ciąży i badaniach prenatalnych
 Analiza genetyczna w niepowodzeniach ciąży i badaniach prenatalnych Dr n. med. Joanna Walczak- Sztulpa Katedra i Zakład Genetyki Medycznej Uniwersytet Medyczny im. Karola Marcinkowskiego w Poznaniu Diagnostyka
Analiza genetyczna w niepowodzeniach ciąży i badaniach prenatalnych Dr n. med. Joanna Walczak- Sztulpa Katedra i Zakład Genetyki Medycznej Uniwersytet Medyczny im. Karola Marcinkowskiego w Poznaniu Diagnostyka
Zalecenia rekomendowane przez Ministra Zdrowia. KPC - ang: Klebsiella pneumoniae carbapenemase
 Zalecenia dotyczące postępowania w przypadku identyfikacji w zakładach opieki zdrowotnej szczepów bakteryjnych Enterobacteriaceae wytwarzających karbapenemazy typu KPC * * KPC - ang: Klebsiella pneumoniae
Zalecenia dotyczące postępowania w przypadku identyfikacji w zakładach opieki zdrowotnej szczepów bakteryjnych Enterobacteriaceae wytwarzających karbapenemazy typu KPC * * KPC - ang: Klebsiella pneumoniae
Laboratorium 5. Wpływ temperatury na aktywność enzymów. Inaktywacja termiczna
 Laboratorium 5 Wpływ temperatury na aktywność enzymów. Inaktywacja termiczna Prowadzący: dr inż. Karolina Labus 1. CZĘŚĆ TEORETYCZNA Szybkość reakcji enzymatycznej zależy przede wszystkim od stężenia substratu
Laboratorium 5 Wpływ temperatury na aktywność enzymów. Inaktywacja termiczna Prowadzący: dr inż. Karolina Labus 1. CZĘŚĆ TEORETYCZNA Szybkość reakcji enzymatycznej zależy przede wszystkim od stężenia substratu
Biologia medyczna, materiały dla studentów
 Zasada reakcji PCR Reakcja PCR (replikacja in vitro) obejmuje denaturację DNA, przyłączanie starterów (annealing) i syntezę nowych nici DNA (elongacja). 1. Denaturacja: rozplecenie nici DNA, temp. 94 o
Zasada reakcji PCR Reakcja PCR (replikacja in vitro) obejmuje denaturację DNA, przyłączanie starterów (annealing) i syntezę nowych nici DNA (elongacja). 1. Denaturacja: rozplecenie nici DNA, temp. 94 o
AKTUALNOÂCI BINET. Nr 10/ Drogie Koleżanki i Koledzy. Inwazyjna choroba meningokokowa w 2015 roku
 www.koroun.edu.pl Drogie Koleżanki i Koledzy Witamy serdecznie, oddajemy w Państwa ręce kolejny numer Aktualności BINet. Jest on poświęcony epidemiologii inwazyjnych zakażeń wywoływanych przez Neisseria
www.koroun.edu.pl Drogie Koleżanki i Koledzy Witamy serdecznie, oddajemy w Państwa ręce kolejny numer Aktualności BINet. Jest on poświęcony epidemiologii inwazyjnych zakażeń wywoływanych przez Neisseria
Zestaw do izolacji DNA z krwi świeżej i mrożonej
 Nr kat. EM05 Wersja zestawu: 1.2014 Zestaw do izolacji DNA z krwi świeżej i mrożonej EXTRACTME jest zastrzeżonym znakiem towarowym firmy BLIRT S.A. www.dnagdansk.com Nr kat. EM05 I. PRZEZNACZENIE ZESTAWU
Nr kat. EM05 Wersja zestawu: 1.2014 Zestaw do izolacji DNA z krwi świeżej i mrożonej EXTRACTME jest zastrzeżonym znakiem towarowym firmy BLIRT S.A. www.dnagdansk.com Nr kat. EM05 I. PRZEZNACZENIE ZESTAWU
Ampli-LAMP Goose Parvovirus
 Novazym Products Zestaw do identyfikacji materiału genetycznego parwowirusa GPV u gęsi techniką Loop-mediated Isothermal AMPlification (LAMP) Numery katalogowe produktu: AML-GPV-200 AML-GPV-400 Wydanie
Novazym Products Zestaw do identyfikacji materiału genetycznego parwowirusa GPV u gęsi techniką Loop-mediated Isothermal AMPlification (LAMP) Numery katalogowe produktu: AML-GPV-200 AML-GPV-400 Wydanie
Autor: dr Mirosława Staniaszek
 Zakład Hodowli Roślin Ogrodniczych Pracownia Genetyki i Hodowli Roślin Warzywnych Fot. J. Sobolewski Procedura identyfikacji genu Frl warunkującego odporność pomidora na Fusarium oxysporum f.sp. radicis-lycopersici
Zakład Hodowli Roślin Ogrodniczych Pracownia Genetyki i Hodowli Roślin Warzywnych Fot. J. Sobolewski Procedura identyfikacji genu Frl warunkującego odporność pomidora na Fusarium oxysporum f.sp. radicis-lycopersici
BIOLOGIA MOLEKULARNA
 BIOLOGIA MOLEKULARNA oferta asortymentowa do diagnostyki molekularnej dla przemysłu i weterynarii Strona 1 Ogólne warunki sprzedaży 1. Zamówienia można składać za pomocą poczty e-mail na adres: zamowienia@argenta.com.pl
BIOLOGIA MOLEKULARNA oferta asortymentowa do diagnostyki molekularnej dla przemysłu i weterynarii Strona 1 Ogólne warunki sprzedaży 1. Zamówienia można składać za pomocą poczty e-mail na adres: zamowienia@argenta.com.pl
Prezentacja Pracowni Ekologii Drobnoustrojów w Katedry Mikrobiologii UJCM
 Prezentacja Pracowni Ekologii Drobnoustrojów w Katedry Mikrobiologii UJCM Informacja o Katedrze Rozwój j naukowy młodej kadry naukowców w w kontekście priorytetów badawczych: W 2009 roku 1 pracownik Katedry
Prezentacja Pracowni Ekologii Drobnoustrojów w Katedry Mikrobiologii UJCM Informacja o Katedrze Rozwój j naukowy młodej kadry naukowców w w kontekście priorytetów badawczych: W 2009 roku 1 pracownik Katedry
SPRAWOZDANIE Z BADAŃ MIKROBIOLOGICZNYCH Nr 20006/11859/09
 SPRAWOZDANIE MOŻE BYĆ POWIELANE TYLKO W CAŁOŚCI. INNA FORMA KOPIOWANIA WYMAGA PISEMNEJ ZGODY LABORATORIUM. SPRAWOZDANIE Z BADAŃ MIKROBIOLOGICZNYCH Nr 20006/11859/09 BADANIA WŁASNOŚCI PRZECIWDROBNOUSTROJOWYCH
SPRAWOZDANIE MOŻE BYĆ POWIELANE TYLKO W CAŁOŚCI. INNA FORMA KOPIOWANIA WYMAGA PISEMNEJ ZGODY LABORATORIUM. SPRAWOZDANIE Z BADAŃ MIKROBIOLOGICZNYCH Nr 20006/11859/09 BADANIA WŁASNOŚCI PRZECIWDROBNOUSTROJOWYCH
Analysis of infectious complications inf children with acute lymphoblastic leukemia treated in Voivodship Children's Hospital in Olsztyn
 Analiza powikłań infekcyjnych u dzieci z ostrą białaczką limfoblastyczną leczonych w Wojewódzkim Specjalistycznym Szpitalu Dziecięcym w Olsztynie Analysis of infectious complications inf children with
Analiza powikłań infekcyjnych u dzieci z ostrą białaczką limfoblastyczną leczonych w Wojewódzkim Specjalistycznym Szpitalu Dziecięcym w Olsztynie Analysis of infectious complications inf children with
[D.24] FORMULARZ ASORTYMENTOWO - CENOWY
![[D.24] FORMULARZ ASORTYMENTOWO - CENOWY [D.24] FORMULARZ ASORTYMENTOWO - CENOWY](/thumbs/71/64225236.jpg) [D.24] FORMULARZ ASORTYMENTOWO - CENOWY Pakiet Nr 24 - Dzierżawa aparatu do posiewu krwi i płynów ustrojowych wraz z dostawą podłoży w automa- tycznym systemie do wczesnego wykrywania bakterii i grzybów
[D.24] FORMULARZ ASORTYMENTOWO - CENOWY Pakiet Nr 24 - Dzierżawa aparatu do posiewu krwi i płynów ustrojowych wraz z dostawą podłoży w automa- tycznym systemie do wczesnego wykrywania bakterii i grzybów
Inżynieria Genetyczna ćw. 3
 Materiały do ćwiczeń z przedmiotu Genetyka z inżynierią genetyczną D - blok Inżynieria Genetyczna ćw. 3 Instytut Genetyki i Biotechnologii, Wydział Biologii, Uniwersytet Warszawski, rok akad. 2018/2019
Materiały do ćwiczeń z przedmiotu Genetyka z inżynierią genetyczną D - blok Inżynieria Genetyczna ćw. 3 Instytut Genetyki i Biotechnologii, Wydział Biologii, Uniwersytet Warszawski, rok akad. 2018/2019
BIOLOGIA KOMÓRKI. Mikroskopia fluorescencyjna -2 Przyżyciowe barwienia organelli wewnątrzkomórkowych
 BIOLOGIA KOMÓRKI Mikroskopia fluorescencyjna -2 Przyżyciowe barwienia organelli Wstęp Komórki eukariotyczne, w odróżnieniu od komórek prokariotycznych (bakterie, archeony) posiadają wysoce skomplikowaną
BIOLOGIA KOMÓRKI Mikroskopia fluorescencyjna -2 Przyżyciowe barwienia organelli Wstęp Komórki eukariotyczne, w odróżnieniu od komórek prokariotycznych (bakterie, archeony) posiadają wysoce skomplikowaną
Wirus HPV w ciąży. 1. Co to jest HPV?
 Wirus HPV w ciąży Czy zdajesz sobie sprawę z tego, że rak szyjki macicy jest drugą, najczęstszą chorobą nowotworową u kobiet na świecie a piąta wśród kobiet i mężczyzn łącznie? W samej Polsce, jak donosi
Wirus HPV w ciąży Czy zdajesz sobie sprawę z tego, że rak szyjki macicy jest drugą, najczęstszą chorobą nowotworową u kobiet na świecie a piąta wśród kobiet i mężczyzn łącznie? W samej Polsce, jak donosi
SPRAWOZDANIE Z BADAŃ MIKROBIOLOGICZNYCH Nr 20005/11858/09
 SPRAWOZDANIE MOŻE BYĆ POWIELANE TYLKO W CAŁOŚCI. INNA FORMA KOPIOWANIA WYMAGA PISEMNEJ ZGODY LABORATORIUM. SPRAWOZDANIE Z BADAŃ MIKROBIOLOGICZNYCH Nr 20005/11858/09 BADANIA WŁASNOŚCI PRZECIWDROBNOUSTROJOWYCH
SPRAWOZDANIE MOŻE BYĆ POWIELANE TYLKO W CAŁOŚCI. INNA FORMA KOPIOWANIA WYMAGA PISEMNEJ ZGODY LABORATORIUM. SPRAWOZDANIE Z BADAŃ MIKROBIOLOGICZNYCH Nr 20005/11858/09 BADANIA WŁASNOŚCI PRZECIWDROBNOUSTROJOWYCH
Czas w medycynie laboratoryjnej. Bogdan Solnica Katedra Biochemii Klinicznej Uniwersytet Jagielloński Collegium Medicum Kraków
 Czas w medycynie laboratoryjnej Bogdan Solnica Katedra Biochemii Klinicznej Uniwersytet Jagielloński Collegium Medicum Kraków Czas w medycynie laboratoryjnej w procesie diagnostycznym pojedynczego pacjenta...
Czas w medycynie laboratoryjnej Bogdan Solnica Katedra Biochemii Klinicznej Uniwersytet Jagielloński Collegium Medicum Kraków Czas w medycynie laboratoryjnej w procesie diagnostycznym pojedynczego pacjenta...
Platforma Genie II. innowacyjne narzędzie do identyfikacji materiału genetycznego patogenów techniką LAMP Loop-mediated Isothermal AMPlification
 1 Platforma Genie II innowacyjne narzędzie do identyfikacji materiału genetycznego patogenów techniką LAMP Loop-mediated Isothermal AMPlification 1 2 Charakterystyka platformy Genie II Genie II jest innowacyjnym
1 Platforma Genie II innowacyjne narzędzie do identyfikacji materiału genetycznego patogenów techniką LAMP Loop-mediated Isothermal AMPlification 1 2 Charakterystyka platformy Genie II Genie II jest innowacyjnym
prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie
 prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie Sekwencyjność występowania zaburzeń molekularnych w niedrobnokomórkowym raku płuca
prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie Sekwencyjność występowania zaburzeń molekularnych w niedrobnokomórkowym raku płuca
Przykłady analizy płynów z jam ciała na analizatorze XE-5000
 Przykłady analizy płynów z jam ciała na analizatorze XE-5000 Jeśli pacjent ma być leczony szybko i skutecznie, laboratorium musi w krótkim czasie dostarczać wiarygodnych wyników, o ile to możliwe przez
Przykłady analizy płynów z jam ciała na analizatorze XE-5000 Jeśli pacjent ma być leczony szybko i skutecznie, laboratorium musi w krótkim czasie dostarczać wiarygodnych wyników, o ile to możliwe przez
WYKRYWANIE OBECNOŚCI BAKTERII Z RODZAJU LISTERIA W ŻYWNOŚCI
 ĆWICZENIE IV Autor i główny prowadzący dr Dorota Korsak WYKRYWANIE OBECNOŚCI BAKTERII Z RODZAJU LISTERIA W ŻYWNOŚCI Wstęp Rodzaj Listeria należy do typu Firmicutes wspólnie z rodzajem Staphylococcus, Streptococcus,
ĆWICZENIE IV Autor i główny prowadzący dr Dorota Korsak WYKRYWANIE OBECNOŚCI BAKTERII Z RODZAJU LISTERIA W ŻYWNOŚCI Wstęp Rodzaj Listeria należy do typu Firmicutes wspólnie z rodzajem Staphylococcus, Streptococcus,
Genomic Mini AX Milk Spin
 Genomic Mini AX Milk Spin Zestaw o zwiększonej wydajności do izolacji genomowego DNA z próbek mleka. wersja 1017 100 izolacji Nr kat. 059-100S Pojemność kolumny do oczyszczania DNA wynosi 15 μg. Produkt
Genomic Mini AX Milk Spin Zestaw o zwiększonej wydajności do izolacji genomowego DNA z próbek mleka. wersja 1017 100 izolacji Nr kat. 059-100S Pojemność kolumny do oczyszczania DNA wynosi 15 μg. Produkt
RAPORT Z BADAŃ 01369/2015/D/AGST. Blirt S.A Gdańsk, ul. Trzy Lipy 3/1.38. Dział DNA-Gdańsk. Nr zlecenia
 Strona1/7 Blirt S.A. 80-172 Gdańsk, ul. Trzy Lipy 3/1.38 RAPORT Z BADAŃ Dział DNA-Gdańsk Nr zlecenia 01369/2015/D/AGST NAZWA I ADRES KLIENTA Zenon Koszorz Ground-Therm Sp z o.o. Ul. Stepowa 30 44-105 Gliwice
Strona1/7 Blirt S.A. 80-172 Gdańsk, ul. Trzy Lipy 3/1.38 RAPORT Z BADAŃ Dział DNA-Gdańsk Nr zlecenia 01369/2015/D/AGST NAZWA I ADRES KLIENTA Zenon Koszorz Ground-Therm Sp z o.o. Ul. Stepowa 30 44-105 Gliwice
bi ~ Zestaw dydal<tyczny umożliwiający przeprowadzenie izolacji genomowego DNA z bakterii EasyLation Genomie DNA INSTRUI<CJA DLA STUDENTÓW
 Zestaw dydaktyczny EasyLation Genomie DNA Nr kat. DY80 Wersja zestawu: 1.2014 Zestaw dydal
Zestaw dydaktyczny EasyLation Genomie DNA Nr kat. DY80 Wersja zestawu: 1.2014 Zestaw dydal
Wojewódzka Stacja Sanitarno-Epidemiologiczna w Poznaniu
 Wojewódzka Stacja Sanitarno-Epidemiologiczna w Poznaniu PROGRAM WHO ELIMINACJI ODRY/RÓŻYCZKI Program eliminacji odry i różyczki został uchwalony przez Światowe Zgromadzenie Zdrowia 28 maja 2003 roku. Realizacja
Wojewódzka Stacja Sanitarno-Epidemiologiczna w Poznaniu PROGRAM WHO ELIMINACJI ODRY/RÓŻYCZKI Program eliminacji odry i różyczki został uchwalony przez Światowe Zgromadzenie Zdrowia 28 maja 2003 roku. Realizacja
Zestaw do izolacji genomowego DNA z bakterii
 Nr kat. EM02 Wersja zestawu: 1.2014 Zestaw do izolacji genomowego DNA z bakterii EXTRACTME jest zastrzeżonym znakiem towarowym firmy BLIRT S.A. www.dnagdansk.com Nr kat. EM02 I. PRZEZNACZENIE ZESTAWU Zestaw
Nr kat. EM02 Wersja zestawu: 1.2014 Zestaw do izolacji genomowego DNA z bakterii EXTRACTME jest zastrzeżonym znakiem towarowym firmy BLIRT S.A. www.dnagdansk.com Nr kat. EM02 I. PRZEZNACZENIE ZESTAWU Zestaw
Polimeraza Taq (1U/ l) 1-2 U 1 polimeraza Taq jako ostatni składniki mieszaniny końcowa objętość
 Ćwiczenie 6 Technika PCR Celem ćwiczenia jest zastosowanie techniki PCR do amplifikacji fragmentu DNA z bakterii R. leguminosarum bv. trifolii TA1 (RtTA1). Studenci przygotowują reakcję PCR wykorzystując
Ćwiczenie 6 Technika PCR Celem ćwiczenia jest zastosowanie techniki PCR do amplifikacji fragmentu DNA z bakterii R. leguminosarum bv. trifolii TA1 (RtTA1). Studenci przygotowują reakcję PCR wykorzystując
Ocena skuteczności preparatów miejscowo znieczulających skórę w redukcji bólu w trakcie pobierania krwi u dzieci badanie z randomizacją
 234 Ocena skuteczności preparatów miejscowo znieczulających skórę w redukcji bólu w trakcie pobierania krwi u dzieci badanie z randomizacją The effectiveness of local anesthetics in the reduction of needle
234 Ocena skuteczności preparatów miejscowo znieczulających skórę w redukcji bólu w trakcie pobierania krwi u dzieci badanie z randomizacją The effectiveness of local anesthetics in the reduction of needle
Wstęp Cele pracy Materiał i metody
 Wstęp. Wirus brodawczaka ludzkiego (HPV, human papillomavirus) ma istotny udział w etiopatogenezie raka płaskonabłonkowego ustnej części gardła. Obecnie częstość występowania raka HPV-zależnego w krajach
Wstęp. Wirus brodawczaka ludzkiego (HPV, human papillomavirus) ma istotny udział w etiopatogenezie raka płaskonabłonkowego ustnej części gardła. Obecnie częstość występowania raka HPV-zależnego w krajach
Zestaw do izolacji totalnego DNA z bakterii. Nr kat. EM02 Wersja zestawu:
 Zestaw do izolacji totalnego DNA z bakterii Nr kat. EM02 Wersja zestawu: 1.2012 Nr kat. EM02 I. PRZEZNACZENIE ZESTAWU Zestaw EXTRACTME DNA BACTERIA przeznaczony jest do szybkiej i wydajnej izolacji bakteryjnego
Zestaw do izolacji totalnego DNA z bakterii Nr kat. EM02 Wersja zestawu: 1.2012 Nr kat. EM02 I. PRZEZNACZENIE ZESTAWU Zestaw EXTRACTME DNA BACTERIA przeznaczony jest do szybkiej i wydajnej izolacji bakteryjnego
XIII. Staphylococcus, Micrococcus ćwiczenia praktyczne
 XIII. Staphylococcus, Micrococcus ćwiczenia praktyczne Ćwiczenie 1. Ocena wzrostu szczepów gronkowców na: a. agarze z dodatkiem 5% odwłóknionej krwi baraniej (AK) typ hemolizy morfologia kolonii.. b. podłożu
XIII. Staphylococcus, Micrococcus ćwiczenia praktyczne Ćwiczenie 1. Ocena wzrostu szczepów gronkowców na: a. agarze z dodatkiem 5% odwłóknionej krwi baraniej (AK) typ hemolizy morfologia kolonii.. b. podłożu
Laboratorium 8. Badanie stresu oksydacyjnego jako efektu działania czynników toksycznych
 Laboratorium 8 Badanie stresu oksydacyjnego jako efektu działania czynników toksycznych Literatura zalecana: Jakubowska A., Ocena toksyczności wybranych cieczy jonowych. Rozprawa doktorska, str. 28 31.
Laboratorium 8 Badanie stresu oksydacyjnego jako efektu działania czynników toksycznych Literatura zalecana: Jakubowska A., Ocena toksyczności wybranych cieczy jonowych. Rozprawa doktorska, str. 28 31.
StayRNA bufor zabezpieczający RNA przed degradacją wersja 0215
 StayRNA bufor zabezpieczający RNA przed degradacją wersja 0215 100 ml, 250 ml, 500 ml Nr kat. 038-100, 038-250, 038-500 Nietosyczny roztwór wodny do przechowywania i zabezpieczania różnego rodzaju tkanek
StayRNA bufor zabezpieczający RNA przed degradacją wersja 0215 100 ml, 250 ml, 500 ml Nr kat. 038-100, 038-250, 038-500 Nietosyczny roztwór wodny do przechowywania i zabezpieczania różnego rodzaju tkanek
XXV. Grzyby cz I. Ćwiczenie 1. Wykonanie i obserwacja preparatów mikroskopowych. a. Candida albicans preparat z hodowli barwiony metoda Grama
 XXV. Grzyby cz I. Ćwiczenie 1. Wykonanie i obserwacja preparatów mikroskopowych a. Candida albicans preparat z hodowli barwiony metoda Grama Opis preparatu: b. Saccharomyces cerevisiae preparat z hodowli
XXV. Grzyby cz I. Ćwiczenie 1. Wykonanie i obserwacja preparatów mikroskopowych a. Candida albicans preparat z hodowli barwiony metoda Grama Opis preparatu: b. Saccharomyces cerevisiae preparat z hodowli
AmpliTest TBEV (Real Time PCR)
 AmpliTest TBEV (Real Time PCR) Zestaw do wykrywania sekwencji RNA specyficznych dla TBEV (Tick-borne encephalitis virus) techniką Real Time PCR Nr kat.: RV03-50 Wielkość zestawu: 50 oznaczeń Objętość pojedynczej
AmpliTest TBEV (Real Time PCR) Zestaw do wykrywania sekwencji RNA specyficznych dla TBEV (Tick-borne encephalitis virus) techniką Real Time PCR Nr kat.: RV03-50 Wielkość zestawu: 50 oznaczeń Objętość pojedynczej
Metody badania ekspresji genów
 Metody badania ekspresji genów dr Katarzyna Knapczyk-Stwora Warunki wstępne: Proszę zapoznać się z tematem Metody badania ekspresji genów zamieszczonym w skrypcie pod reakcją A. Lityńskiej i M. Lewandowskiego
Metody badania ekspresji genów dr Katarzyna Knapczyk-Stwora Warunki wstępne: Proszę zapoznać się z tematem Metody badania ekspresji genów zamieszczonym w skrypcie pod reakcją A. Lityńskiej i M. Lewandowskiego
