WIRUSOLOGIA MOLEKULARNA
|
|
|
- Justyna Rogowska
- 8 lat temu
- Przeglądów:
Transkrypt
1 WIRUSOLOGIA MOLEKULARNA Skrypt do ćwiczeń 2015 Zakład Wirusologii dr Monika Adamczyk-Popławska dr Agnieszka Kwiatek dr Monika Radlińska Instytut Mikrobiologii Wydział Biologii UW
2 nr ćwiczenia infekcyjne bakteriofagów (liza i lizogenia) Techniki namnażania wirusów i oznaczanie ich miana Mechanizmy obronne bakterii przed infekcją bakteriofagami Metody indukcji profagów Otrzymanie biblioteki fragmentów restrykcyjnych faga HP1 w wektorze plazmidowym pbluescript KS II (+) Diagnostyka wirusologiczna Phage Display Transfekcja H.influenzae DNA faga HP1 Elementy bakteriofagowe wykorzystywane w biologii molekularnej liza i lizogenia faga - test kroplowy uzyskanie lizogenów faga HP1 namnażanie faga vir (1) analiza wyników lizy i lizogenii faga izolacja lizogenów faga wt (1) izolacja lizogenów faga HP1 (1) namnażanie faga vir (2) oznaczanie miana faga vir izolacja lizogenów faga wt (2) izolacja lizogenów faga HP1 (2) oznaczanie miana faga vir odczytanie wyników infekcja abortywna (1) restrykcjamodyfikacj a (1) izolacja i uzyskanie lizogenów faga HP1 i wt - odczytanie i analiza wyników infekcja abortywna (2) restrykcjamodyfikacja (2) odczytanie i analiza wyników indukcja faga HP1 indukcja faga CIts857S7 (1) immobilizacj a białka dodanie biblioteki bakteriofago wej indukcja faga CIts857S7 (2) izolacja DNA faga HP1 izolacja DNA chromosoma lnego H. influenzae elucja i namnożenie specyficznie związanych fagów izolacja DNA faga HP1 indukcja faga CIts857S7 - analiza wyników przygotowanie substratów rekombinacji DNA: - trawienie restrykcyjne DNA faga HP1 i wektora pbluescript; - ligacja w/w DNA wytrącanie i mianowanie faga wprowadzen ie zrekombino wanego DNA do komórek E. coli - transformacj a namnożenie fagów wykonanie izolacja i analiza restrykcyjna DNA rekombinant ów wykrywanie materiału genetyczneg o wirusów (technika PCR) analiza wyników izolacja DNA faga M13 wykonanie analiza wyników analiza wyników analiza wyników 2
3 3
4 ĆWICZENIE 1 Część teoretyczna Cykle infekcyjne bakteriofagów: liza i lizogenia (1) Techniki namnażania bakteriofagów (1) Wirusy są bezwzględnymi wewnątrzkomórkowymi pasożytami, o budowie niekomórkowej, pozbawionymi własnego metabolizmu. Materiałem genetycznym wirusów jest DNA lub RNA. Te dwa rodzaje kwasu nukleinowego nigdy nie występują jednocześnie w tej samej cząstce wirusowej. Genom wirusów zawiera informację genetyczną niezbędną do odtwarzania cząstek potomnych wirusa i syntezy enzymów, czyli zawiera geny kodujące informacje o budowie (sekwencji nukleotydowej) rdzenia oraz białkowego kapsydu. Natomiast genom wirusów nie koduje żadnej informacji genetycznej dotyczącej własnych układów enzymatycznych, jakie są potrzebne do przeprowadzania procesów metabolicznych (np. aparatu do wytwarzania i przekształcania energii, ani do syntezy białek). Nukleinowy rdzeń otoczony jest białkowym kapsydem. Kapsyd zbudowany jest z kapsomerów, które są uorganizowanymi oligomerami zbudowanymi najczęściej z 5 6 cząsteczek białka. Kapsyd może zawierać jeden rodzaj białka, ale zazwyczaj zawiera dwa lub więcej rodzajów. Symetria kapsydu może być helikalna (pałeczkowata) lub ikosaedralna (bryłowa, kulista). Kapsydy ikosaedralne są najczęściej ikosaedrami (dwudziestościami) lub dodekaedrami (dwunastościanami). Ponadto występują wirusy posiadające symetrię złożoną. Kapsyd i rdzeń nukleinowy tworzą nukleokapsyd. Nukleokapsyd może być nagi lub okryty dodatkową osłonką. Osłonki zawierają białka, cukry i tłuszcze. Wirion jest to pojedyncza zakaźna cząstka wirusa zdolna do replikacji w żywej komórce gospodarza. Wirusy ze względu na przyjęte kryterium możemy podzielić na różne grupy: 1. kryterium: komórki gospodarza: - Wirusy bakteryjne (bakteriofagi), np. bakteriofag M13,, X174, HP1, Ngo 6, CTX ; - Wirusy roślinne, np. wirus mozaiki tytoniu (TMV), wirus mozaiki kalafiora (CaMV); - Wirusy zwierzęce, np. wirus grypy, ospy, ospy wietrznej, zapalenia wątroby (HCV, HBV, HAV), brodawczaka ludzkiego (HPV), ludzki wirus niedoboru odporności (HIV), SV40; - Wirusy infekujące grzyby, np. ScV L-A (wirus L-A infekujący Saccharomyces cerevisiae); - Wirusy infekujące Protista; 2. kryterium: materiał genetyczny: - Zawierające dwuniciowy DNA (ang. double-stranded DNA, dsdna), dsdna może być w formie liniowej lub kolistej. Do tej grupy wirusów należą m.in. wirus ospy, herpeswirusy, bakteriofag, bakteriofag HP1, HPV, bakulowirusy, bakteriofag T7; - Zawierające jednoniciowy DNA (ang. single-stranded DNA, ssdna), genom może być w postaci jednej cząsteczki lub być podzielony na dwa lub więcej segmentów; DNA może być zarówno o polarności dodatniej jak i ujemnej; np. M13, X174; - Zawierające RNA o dodatniej polarności [(+) RNA], np. HCV, wirus różyczki; - Zawierające RNA o ujemnej polarności [(-) RNA], genom może być w postaci jednego lub kilku segmentów, np. wirus Ebola, wirus odry; - Zawierające dwuniciowy RNA (dsrna), w większości przypadków genom składa się z dwóch lub większej liczby segmentów RNA, np. ScV L-A, rotawirusy; 3
5 3. kryterium: wielkość: - Wirusy małe do 50 nm; - Wirusy średnie do 150 nm; - Wirusy duże powyżej 150 nm; Bakteriofagi Bakteriofagi (fagi) to wirusy infekujące bakterie (bakteriofagi należące do 10 rodzin) i archeony (10 rodzin). Materiałem genetycznym fagów jest: (+) ssrna, dsrna, ssdna (fag M13, X174) lub dsdna (kolisty lub liniowy) (, T7, HP1). Cząstki wirusowe mogą mieć budowę prostą lub złożoną. Cząstki o budowie prostej mogą mieć strukturę helikalną (M13) lub izomeryczną ( X174). W cząstkach bakteriofagowych o budowie złożonej wyróżniamy główkę i ogonek (, T7, HP1). Główka jest ikosaedralna, przy czym ogonek może być krótki (T7), długi kurczliwy (Mu) lub długi niekurczliwy ( ) (Rys. 1). Wirion rzadko otoczony jest osłonką. Rys. 1. Przykład budowy morfologicznej bakteriofagów złożonych. Etapy namnażania się bakteriofagów w komórkach gospodarza przebiegają podobnie jak innych wirusów i obejmują następujące etapy: - Adsorpcja cząstek fagowych do powierzchni wrażliwych komórek gospodarza (białka fagowe, receptory, odpowiednie warunki fizyko-chemiczne, m.o.i.); proces ten polega na utworzeniu wiązań jonowych między receptorem bakteriofagowym a receptorem bakteryjnym; - Penetracja kwasu nukleinowego; - Transkrypcja, translacja, replikacja bakteriofagowego materiału genetycznego; - Składanie i dojrzewanie cząstek bakteriofagowych; - Uwolnienie cząstek wirusowych z komórek gospodarza*; * W określonych warunkach: (1) może nie dojść do adsorpcji cząstek fagowych do komórek gospodarza; (2) zajdzie adsorpcja cząstek fagowych do powierzchni komórek gospodarza i wniknięcie kwasu nukleinowego, ale: (i) DNA zostanie wbudowany w genom gospodarza wystąpi zjawisko lizogenii; (ii) DNA zostanie zdegradowany między innymi przez stanowiące część systemów restrykcji modyfikacji endonukleazy restrykcyjne komórek gospodarza (ćwiczenie 3 i 4); (iii) wystąpi zjawisko infekcji abortywnej (ćwiczenie 3 i 4). Komórka bakteryjna może zostać zakażona fagami, jeśli jest na nie wrażliwa (ang. sensitive). Bakterie mogą być genetycznie oporne na zakażenie fagami. Komórki takie nie posiadają na powierzchni, koniecznych do adsorpcji wirusa receptorów. Natomiast szczepy bakteryjne odporne (and. resistant), to takie, które posiadają w swoim genomie wintegrowanego faga i są niewrażliwe na infekcję takim samych lub pokrewnym fagiem. Szczep bakteryjny, który zapewnia optymalne warunki do namnożenia się bakteriofaga to szczep permisywny, a taki, którego cechy nie pozwalają na namnożenie się fagów to szczep restryktywny. 4
6 Bakteria posiadająca w swoim genomie wbudowany genom infekującego ją faga to lizogen, a bakteriofag wbudowany w genom gospodarza to profag. W genomach wielu gatunków bakterii wykryto sekwencje profagowe. Często występuje więcej niż jedna sekwencja profagowa w jednym genomie bakteryjnym. W skrajnych przypadkach np. w genomie Escherichia coli O157:H7 szczep Sakai występuje 18 sekwencji profagowych, które stanowią 16% genomu gospodarza. Innym przykładem jest Streptococcus pyogenes, który w zależności od szczepu zawiera od 4 do 6 profagów, które stanowią nawet 12% DNA gospodarza (Rys. 2). Rys. 2. Występowanie sekwencji profagowych w genomach wybranych bakterii. Profagi zaznaczono szarymi prostokątami. (A) S. pyogenes szczep M1, M18 i M3 (od środka do zewnątrz); (B) S. aureus szczep Mu50, N315, MW2 i 8325; (C) E. coli o157:h7 Sakai, O157:H7 EDL933, K12 i CFT073; (D) S. enterica serovar Typhimurium LT2 i serovar Typhi CT18 (Canchaya i wsp. 2003). Sekwencje profagowe mają znaczenie nie tylko ilościowe w genetyce i fizjologii bakterii, ale również znaczenie jakościowe. Z występowaniem procesu lizogenii związana jest superinfekcja i konwersja lizogenna. Superinfekcja jest to zjawisko polegające na tym, że bedące lizogenami komórki gospodarza są odporne na ponowne zakażenie takim samym lub pokrewnym bakteriofagiem. Fag adsorbuje się do odpornych komórek i wstrzykuje DNA, ale nie namnaża się w takiej komórce. Lizogen jest odporny na ponowne zakażenie takim samym fagiem, ale jest wrażliwy na infekcję innym fagiem, nawet lizogenizującym ale włączającym swój genom w inne miejsce chromosomu gospodarza. Konwersja lizogenna jest to proces, na skutek którego komórki gospodarza nabywają nowe cechy, w tym cechy związane z ich wirulencją. Corynobacterium diphtheriae wytwarza toksynę, jeżeli występuje w postaci lizogena. Wirulencja Vibrio cholerae jest także związana z występowaniem sekwencji profagowej w genomie tej bakterii. Toksyna cholery jest kodowana przez nitkowatego profaga CTX zawierającego 10 genów, m.in. rstr, rsta, rstc, rstb, cep, orfu, ace, zot. Gen zot koduje enterotoksynę. Innymi bakteriami, u których wykazano związek konwersji lizogennej z chorobotwórczością są uropatogenne E. coli, Salmonella sp., Haemophilus influenzae szczepy Sb., Pseudomonas aeruginosa (lizogenizacja profagiem D3 powoduje zmianę antygenu powierzchniowego) (Tabela 1). 5
7 Tabela 1. Związek sekwencji profagowych z wirulencją niektórych bakterii patogennych. Bakteria Fag Produkt genu Vibrio cholerae CTX cholera toksyny Streptococcus pyogenes T12 toksyna erytrogenna Corynebacterium diphtheriae bakteriofag β toksyna błonicy Clostridium botulinum CEβ toksyna botulinowa Escherichia coli bakteriofagi lambdoidalne toksyna Shiga Shigella dysenteriae bakteriofagi lambdoidalne Stx1 i Stx2 Ponadto znaczenie sekwencji profagowych nie ogranicza się tylko do bakterii patogennych, ale także przystosowanie bakterii niepatogennych do ich niszy ekologicznej może być związane z występowaniem profagów w ich genomie. Bakteriofag lambda ( ) Gospodarzem bakteriofaga lambda ( ) są bakterie z gatunku E. coli. Bakteriofag ten należy do rodziny Siphoviridae. jest bakteriofagiem łagodnym, tzn może wywoływać zarówno lizę jak i lizogenię. Fag ten charakteryzuje się budową złożoną ma główkę ikosaedralną i długi niekurczliwy ogonek. Genom jest w postaci dwuniciowego liniowego DNA o wielkości pz i zakończony jest lepkimi końcami 5 (12 nt sekwencje cos niezbędne w procesie pakowania DNA do główek fagowych). Do wydajnej adsorpcji wirusa na powierzchni komórek gospodarza wymagana jest obecność Mg 2+. W czasie adsorpcji włókno ogonka (23 nm) (zbudowane z białek gpj) łączy się z receptorem bakteryjnym [białko LamB (część systemu transportu maltozy)]. W genomie gospodarza znajduje się miejsce attb, a w genomie wirusa attp. Są to regiony homologiczne, zachodzi wbudowanie DNA bakteriofaga do genomu gospodarza. W procesie tym uczestniczą integrazy tyrozynowe. Jednym z białek odpowiedzialnych za utrzymanie stanu lizogenii jest represor CI. Drogą alternatywą w cyklu życiowym bakteriofaga jest liza. Jest to proces, w którym dochodzi do powstania i uwolnienia cząstek fagowych (Rys. 3). Liza komórek gospodarza zachodzi w bogatym podłożu, optymalnej temperaturze i niskim m.o.i (ang. multiplicity of infection). Widocznym objawem lizy na hodowli bakteryjnej prowadzonej na podłożu stałym jest łysinka. Każda łysinka zawiera ok cząstek wirusowych. Natomiast uboga pożywka, niska temperatura oraz wysokie m.o.i. sprzyjają lizogenii. Indukcja profaga (przejście od stanu lizogenii do lizy) może nastąpić spontanicznie lub być indukowana czynnikami fizycznymi, np. promieniowanie UV, lub chemicznymi, np. mitomycyna C. 6
8 Rys. 3. Schemat dwóch szlaków cyklu życiowego bakteriofaga. Dziki szczep lambdy jest bakteriofagiem łagodnym, czyli takim, który może powodować zarówno lizę jak i lizogenię. Natomiast w laboratoriach skonstruowano kilka zmutowanych szczepów: (i) vir wirulentna forma bakteriofaga lambda; ma zmutowany operator; zawsze wchodzi w cykl lityczny; (ii) CIts857 bakteriofag ze zmutowanym represorem CI; represor ten jest temperaturowrażliwy, w temp. 30 C ma prawidłową konformację, a w temp. 42 C ma nieprawidłową; (iii) CIts857Sam7 bakteriofag, który posiada zarówno temperaturowrażliwy represor CI jak i mutację typu amber w genie kodującym białko S. Mutanty typu amber tworzą łysinki jedynie na szczepach zawierających supresor dla tych mutacji. Bakteriofag HP1 Gospodarzem bakteriofaga HP1 jest Haemophilus influenzae. Fag ten należy do rodziny Myoviridae. Jest to bakteriofag łagodny. HP1 charakteryzuje się budową złożoną. W jego budowie wyróżnia się główkę ikosaedralną i kurczliwy ogonek zakończony kurczliwymi włókienkami. Genom tego wirusa stanowi dwuniciowy liniowy DNA o wielkości pz i zakończony jest 9 nt lepkimi końcami (tzw. sekwencje cos). Na powierzchni komórek gospodarza receptorami dla bakteriofaga HP1 jest LOS (lipooligosacharyd). Bakteriofagi i HP1 są przykładami bakteriofagów łagodnych. Fagi te tworzą łysinki mętne, ponieważ lizują tylko część komórek gospodarza. Obok bakteriofagów łagodnych występują również bakteriofagi zjadliwe (wirulentne). Wywołują one zawsze lizę. Przykładem takiego faga jest np. bakteriofag T7 infekujący E. coli. 7
9 Namnażanie bakteriofagów Bakteriofagi można namnażać w hodowlach stałych lub płynnych. Bakterie i fagi miesza się w proporcjach takich, aby liczba fagów była znacząco mniejsza niż liczba bakterii (m.o.i.) (Rys. 4. pkt 1). Mieszanina jest inkubowana w warunkach pozwalających bakteriom dzielić się i ulegać infekcji poprzez fagi. W pierwszej metodzie, do mieszaniny bakterii i bakteriofagów należy dodać tzw. miękki agar (półpłynny) (Rys. 4 pkt 2). Wszystko wylewamy na powierzchnię płytki agarowej (Rys. 4. pkt 3, 4). Bakterie dzielą się, fagi infekują tylko przyległe bakterie, ponieważ są unieruchomione w agarze. Nie zainfekowane komórki kontynuują podziały, tworząc tzw. murawę bakterii. Zainfekowane bakterie ulegają lizie: w ten sposób powstają przejrzyste obszary na tle zmętniałej murawy bakterii: nazywamy je łysinkami (plaque) (Rys. 4. pkt 5). Po uwzględnieniu współczynnika rozcieńczenia faga możemy policzyć łysinki i obliczyć liczbę infekcyjnych fagów w oryginalnej próbce. Liczbę fagów podaje się jako liczbę jednostek tworzących łysinki (pfu - ang. plaque forming units, jednostki tworzące łysinkę). Rys. 4. Schemat przedstawiający wykonanie płytek dwuwarstwowych. 8
10 Test kroplowy (spot-test) Test polega na nakropleniu na murawkę bakteryjną zawiesiny fagowej (np. odpowiednio rozcieńczonej). Metoda pozwala na szybkie określenie miana faga (na jednej szalce). Murawkę bakteryjną przygotowujemy metodą płytek dwuwarstwowych poprzez zmieszanie szczepu bakteryjnego z podłożem półpłynnym. Test krzyżowy (ang. cross-streaking) fag bakterie Test polega na krzyżowaniu linii z zawiesinami fagowymi oraz linii ze szczepami bakteryjnymi. Zawiesinę fagową nakładamy na szalkę z podłożem stałym ezą. Pozwalamy, aby zawiesina dobrze wsiąkła w podłoże, a następnie nanosimy zawiesiny bakteryjne (również za pomocą ezy). 9
11 Część praktyczna Materiały Szczepy bakteryjne: - E. coli Top10 ( - ) s r - m -, F - mcra (mrr-hsdrms-mcrbc) 80 LacZ M15 lacx74 deor reca1 arad139 (ara-leu) 7697 galu galk lambda^-rpsl enda1 nupg - E. coli ER3102 ( cits857 ) s - E. coli ER1562 ( wt ) s MM294, mcra1272::tn10 (Tet r ), hsdr2 (r k -, m k + ), mcrb1 - Haemophilus influenzae Rd30 (HP1 - ) HP1 s Bakteriofagi (miana fagów zostaną podane przez prowadzącego zajęcia): - zawiesiny fagów lambda: wt, CIts857, vir - zawiesina faga HP1 Podłoża: - LB płynne 1% trypton, 0,5% ekstrakt drożdżowy, 0,5% NaCl - LB półpłynne skład j.w. z dodatkiem 0,7% agaru - LB stałe skład j.w. z dodatkiem 1,5% agaru - pożywka BHI (ang. brain heart infusion - wyciąg mózgowo-sercowy) uzupełniona heminą stężenie końcowe w pożywce: 10 µg/ml i dinukleotydem nikotynoaminoadeninowym (NAD): 2 µg/ml Roztwory: - bufor S : 50 mm TrisCl (ph 7,5), 100 mm NaCl, 8 mm MgSO 4, 2% żelatyna (w/v) - NAD: stężenie początkowe 0,4 mg/ml - hemina: stężenie początkowe 1 mg/ml rozpuszczona w 0,1 N NaOH z dodatkiem L-histydyny (1 mg/ml) Wykonanie 1. Namnażanie bakteriofaga vir (patrz rys. 4. na str. 8) - zawiesinę faga vir rozcieńczyć w buforze SM tak, aby otrzymać ok pfu/ml; - zmieszać w probówce 0,2 ml nocnej hodowli szczepu E. coli Top10 z 0,1 ml odpowiednio rozcieńczonego faga vir; - inkubować 10 min w temp. pokojowej; - dodać 3 ml LB półpłynnego schłodzonego do temp. 45 C, wymieszać i wylać na szalkę z LB stałym; - inkubować h w temp. 37 C; 2. Liza i lizogenia (patrz rysunek str. 9) - wylać sześć szalek ze stałym podłożem LB, każdą szalkę podzielić na trzy części i podpisać: wt, vir, CIts857; - na tak przygotowane szalki wysiać powierzchniowo jeden ze szczepów: Top10, ER1562 lub ER3102 (3 ml półpłynnego LB o temp. 45 C + 0,2 ml nocnej hodowli bakteryjnej) pamiętając o odpowiednim podpisaniu szalek, każdy szczep wysiać w 2 powtórzeniach; - po zakrzepnięciu górnej warstwy agarowej nanieść mikropipetą na powierzchnię po 5 µl każdej zawiesiny fagów na odpowiadający im sektor szalek; - odczekać aż płyn z fagami wsiąknie w agar; - inkubować h, odpowiednio w temp. 30 C lub 42 C i odczytać wynik; 10
12 3. Uzyskanie lizogenów faga HP1 - na szalkę ze stałym podłożem BHI uzupełnionym NAD i heminą wysiać powierzchniowo szczep Haemophilus influenzae Rd30 (3 ml półpłynnego BHI o temp. 45 C + 0,1 ml nocnej hodowli bakteryjnej); - po zakrzepnięciu górnej warstwy agarowej nanieść mikropipetą na powierzchnię po 2 µl zawiesiny faga HP1, odczekać aż płyn z fagami wsiąknie w agar, inkubować h w temp. 37 C i odczytać wynik; Wybrana literatura: 1. Canchaya C, Proux C, Fournous G, Bruttin A, Brüssow H (2003). Prophage genomics. Microbiol. Mol. Biol. Rev. 67 (2): Waldor, M.K. and Mekalanos, J.J (1996) Lysogenic conversion by a filamentous phage encoding cholera toxin. Science, 272 (5270): Eklund, M. W., F. T. Poysky, S. M. Reed, and C. A. Smith. (1971). Bacteriophage and the toxigenicity of Clostridium botulinum type C. Science 172:
13 ĆWICZENIE 2 Cykle infekcyjne bakteriofagów: liza i lizogenia (2) Techniki namnażania wirusów i oznaczanie ich miana (2) Część teoretyczna Namnażanie wirusów prowadzi się w hodowlach zawierających wrażliwe komórki gospodarza. Namnażanie i izolację wirusów zwierzęcych prowadzi się w liniach komórkowych i tkankowych. Linie komórkowe wyprowadza się z różnych tkanek i narządów człowieka. Różnią się one wrażliwością na wirusy zaliczane do różnych grup systematycznych. Do namnażania i izolacji wirusów wybiera się komórki najbardziej wrażliwe na poszczególne wirusy i pozwalające na uzyskanie najwyższego zbioru zakaźnych cząstek wirusa (Tabela 2). Tabela 2. Przykłady linii komórkowych wykorzystywanych do namnażania wirusów zwierzęcych. Nazwa komórek Pochodzenie Tkanka Morfologia Wrażliwość na wirusy JM człowiek limfocyty T limfocyty HIV HeLa człowiek rak szyjki macicy nabłonkowe polio1, adenowirusy, herpeswirusy Chang człowiek wątroba nabłonkowe polio 1-3, adenowirusy, VSV KB człowiek rak jamy ustnej nabłonkowe polio 1, adenowirusy WI-38 człowiek płuca fibroblastyczne polio 1, VSV, adenowirusy MA104 małpa nerka nabłonkowe SV40, rotawirusy BHK chomik nerka fibroblastyczne polio 1-3, VSV, arbowirusy, herpes simplex Oprócz hodowli komórkowych do namnażania wirusów wykorzystywane są również hodowle tkankowe i narządowe. Hodowle te służą także do badania zmian patologicznych wywoływanych przez wirusy. Aby namnożyć wirusa w hodowli komórkowej lub tkankowej, należy uwzględnić: (i) wrażliwość danej komórki lub tkanki na infekcję namnażanego wirusa; (ii) dawkę zakaźną wirusa. Zakaźna dawka wirusa może być wyrażona w różnych jednostkach: - TCID 50 (ang. tissue culture infectious dose) 50% dawka zakaźna dla hodowli komórek, czyli takie rozcieńczenie wirusa, które wywołuje efekt cytopatyczny w 50% zakażonych hodowli komórkowych; - pfu (ang. plaque forming units) jednostka łysinkotwórcza, najwyższe rozcieńczenie wirusa wywołujące zniszczenie hodowli komórek w postaci łysinek dających się policzyć; - m.o.i. liczba cząstek wirusa przypadająca na jedną komórkę; Odpowiednia dawka zakaźna wirusa decyduje o końcowym mianie namnażanego wirusa i jego zdolnościach infekcyjnych. W zależności od rodzaju hodowli i wirusa, namnożonego wirusa 12
14 zbiera się po godzinnej inkubacji w temperaturze odpowiadającej danemu układowi (zwykle od 26 do 37 C). Efekt cytopatyczny to wywołany przez wirusa nieodwracalny proces zniszczenia komórki. Efekt cytopatyczny jest charakterystyczny dla określonej grupy wirusów i w zależności od rodzaju wirusa powoduje różne zmiany w hodowlach komórek in vitro: - Zmiana morfologii komórek; - Powiększenie i deformacja jądra komórkowego; - Powstanie wtrętów komórkowych; - Degranulacja mitochondriów; - Rozpad błony komórkowej; - Tworzenie wielojądrzastych komórek olbrzymich syncytiów; Miano to liczba cząstek wirusowych znajdujących się w jednostce objętości. Miano pfu ponieważ nie każda cząstka wirusowa znajdująca się w roztworze spowoduje powstanie widocznego efektu. Aby określić rzeczywistą liczbę infekcyjnych cząstek wirusowych, przeprowadza się proces miareczkowania wirusów. 13
15 Część praktyczna Materiały Szczepy bakteryjne: - E. coli Top ( - ) s r - m -, F - mcra (mrr-hsdrms-mcrbc) 80 LacZ M15 lacx74 deor reca1 arad139 (ara-leu) 7697 galu galk lambda^-rpsl enda1 nupg - szczepy bakteryjne uzyskane w czasie ćwiczenia 1 Bakteriofagi: - bakteriofag vir namnożony w czasie ćwiczenia 1 Podłoża - LB półpłynne - LB stałe - BHI Roztwory - bufor S - chloroform Wykonanie 1. Namnażanie bakteriofaga vir - każdą z szalek zalać 4 ml buforu SM i inkubować w temp. 4 C przez 2 h; - zlać zawiesinę razem z półpłynnym agarem z każdej szalki do 200 ml kolby (wszystkie pary do jednej kolby); - dodać 1 ml chloroformu i wytrząsać przez 10 min w temp. pokojowej; - zawiesinę odwirować 10 min rpm; - supernatant przenieść do czystej kolby, dodać 0,2 ml chloroformu i oznaczyć miano (pkt 2); 2. Oznaczanie miana faga vir Metoda płytkowa - przygotować 3 probówki wirówkowe typu eppendorf (eppendorfówki) zawierające po 1 ml buforu SM; - wykonać serię stukrotnych rozcieńczeń faga vir, pobierając do rozcieńczeń po 10 l zawiesiny (pamiętając o zmianie końcówek pipet); - z rozcieńczeń 10-4 i 10-6 pobrać po 0,1 ml faga vir, zmieszać z 0,2 ml nocnej hodowli szczepu E. coli Top10 (w probówce szklanej), inkubować 10 min w temperaturze pokojowej, dodać 3 ml LB półpłynnego i wysiać na szalki wg schematu przedstawionego na rys. 4. str. 11; - inkubować h w temp. 37 C; Test kroplowy (spot - test) - denko szalki z agarem podzielić markerem na cztery części i podpisać: 0, -2, -4, -6; - do probówki z półpłynnym LB schłodzonym do temp. 45 C dodać 0,2 ml nocnej hodowli szczepu E. coli Top10 i po wymieszaniu wylać na szalkę z LB stałym; - po zakrzepnięciu agaru półpłynnego na jego powierzchnię nanieść po 5 l odpowiedniego rozcieńczenia faga vir (tak, aby nie uszkodzić powierzchni agaru); - inkubować h w temp. 37 C; 14
16 3. Lizogenia Test 1. Izolacja lizogenów faga wt - pobrać ezą odrobinę materiału z łysinki na sektorze szalki podpisanej Top10/ wt (ćw.1; pkt 3; test 1); - wysiać na podłoże LB stałe posiewem redukcyjnym, w celu uzyskania pojedynczych, zlizogenizowanych kolonii; - inkubować h w temp. 37 C; Test 2. Izolacja lizogenów faga HP1 - pobrać ezą odrobinę materiału z łysinki z szalki podpisanej Haemophilus influenzae Rd30/HP1 (ćw.1; pkt 4) ; - wysiać na podłoże stałe BHI uzupełnione NAD i heminą, w celu uzyskania pojedynczych, zlizogenizowanych kolonii; - inkubować h w temp. 37 C; szczep fag wt vir CIts C 42 C 30 C 42 C 30 C 42 C Top10 ( - ) s ER1562( wt ) s ER3102( ci ) s 0 brak łysinek M łysinki mętne P łysinki przejrzyste LM liza murawki 15
17 ĆWICZENIA 3 i 4 Mechanizmy obronne bakterii przed infekcją bakteriofagami: restrykcja modyfikacja oraz abortywna infekcja Część teoretyczna Restrykcja modyfikacja DNA Systemy restrykcji-modyfikacji (RM) zostały przypadkowo odkryte i opisane na początku lat 50-tych XX wieku podczas prac nad bakteriofagami T-parzystymi E. coli. Ówczesnych badaczy zaintrygował fakt, że fag po udanej infekcji jednego ze szczepów nie jest w stanie się namnożyć lub słabo namnaża się na innym szczepie bakterii. Na tej podstawie stworzono teorię, według której bakteria musi posiadać mechanizm obrony przed fagiem, niezwiązany z brakiem receptorów dla faga. Takim właśnie mechanizmem są systemy restrykcji-modyfikacji. W roku 1978 Werner Arber, Daniel Nathans i Hamilton Smith otrzymali nagrodę Nobla w dziedzinie Medycyny za odkrycie enzymów restrykcyjnych. Systemy RM opierają się na dwóch aktywnościach: modyfikacji (metylacji) DNA w specyficznej sekwencji DNA i endonukleolitycznemu cięciu dwuniciowego niezmodyfikowanego DNA. Brak modyfikacji jest sygnałem do rozpoznania DNA jako obcego. Metylacja zasad w obrębie tylko jednej z nici DNA wystarcza, aby zablokować działanie endonukleazy restrykcyjnej. Systemy restrykcji-modyfikacji DNA występują powszechnie u prokariota, kodowane są zarówno przez chromosomy i plazmidy. Aktualnie systemy RM podzielono na 4 grupy (Tabela 3). Tabela 3. Właściwości poszczególnych typów systemów restrykcji-modyfikacji. Charakterystyka Typy systemów restrykcji-modyfikacji Typ I Typ II Typ III Typ IV Cechy strukturalne Wielofunkcyjny, wieloskładnikowy, 3 podjednostki, 3 geny Proste, oddzielne enzymy, 2 podjednostki, 2 geny Wielofunkcyjny, wieloskładnikowy, 2 podjednostki, 2 geny Szereg podjednostek i genów, brak enzymu modyfikacyjnego Aktywności enzymatyczne ATPaza Endonukleaza Metylotransferaza Endonukleaza Metylotransferaza Endonukleaza Metylotransferaza ATPaza Endonukleaza GTPaza Kofaktory niezbędne do aktywności metylotransferaz Kofaktory niezbędne do aktywności endonukleaz restrykcyjnych Rozpoznawana sekwencja Miejsce cięcia Ado-Met, Mg 2+ Ado-Met Ado-Met, Mg 2+ ATP, Ado-Met, Mg 2+ Mg 2+ ATP, Mg 2+, (Ado-Met) Dwuczęściowa, asymetryczna Cięcie przypadkowe, daleko od rozpoznawanej sekwencji Z reguły symetryczna Cięcie w stałej pozycji wewnątrz lub kilka nt od miejsca rozpoznawania Asymetryczna Cięcie w stałym miejscu, oddalonym od rozpoznawanej sekwencji nt Mg 2+, GTP Dwuczęściowa, zmetylowana Cięcie w zmetylowanej sekwencji Translokacja DNA Translokacja Brak Translokacja Translokacja Przedstawiciele EcoBI, EcoAI, EcoDXXI EcoRI, EcoRV, BamHI EcoP1, EcoP15, StyLTI McrA, McrB, DpnI 16
18 Endonukleazy należące do systemów RM typu II są powszechnie używane w biologii molekularnej, ze względu na przeprowadzanie reakcji w ściśle określonym miejscu. Metylotransferazy modyfikują DNA, wprowadzając grupę metylową, do reszt cytozyny bądź adeniny. Donorem grupy metylowej jest S-adenozylo-L-metionina. Produktami reakcji mogą być N6-metyloadenina, C5-metylocytozyna lub N4-metylocytozyna. Metylazy typu I i III metylują wyłącznie adeninę w pozycji N6. Enzymy typu IV rozpoznają i wprowadzają cięcie w DNA, jeżeli rozpoznana sekwencja jest zmetylowana (DpnI jest powszechnie używany w biologii molekularnej). Budowa, mechanizm rozpoznawania oraz cięcia DNA poprzez enzymy należące do systemów RM typu I i III są bardziej skomplikowane. Enzymy restrykcyjne typu I składają się z 3 podjednostek kodowanych przez 3 geny: hsds, hsdm i hsdr. Produkty wszystkich trzech genów są niezbędne dla uzyskania aktywności restrykcyjnej. Podjednostki HsdM i HsdS tworzą aktywną metylazę DNA (Rys. 5). Podjednostka HsdS odpowiedzialna jest za rozpoznawanie specyficznej sekwencji, ale nie posiada żadnej aktywności biochemicznej. Enzymy typu III kodowane są przez dwa geny mod i res. Metylaza Mod odpowiedzialna jest zarówno za rozpoznawanie specyficznej sekwencji DNA i jej metylację. W połączeniu z Res staje się endonukleazą. Rys. 5. Endonukleaza typu I (na biało: HsdM, na szaro HsdR i na czarno HsdS). Endonukleazy typu III (zbudowane z dwóch podjednostek mod i res) wprowadzają cięcie w stałym miejscu, oddalonym od rozpoznawanej sekwencji nt. W przypadku systemów RM typu I cięcie DNA jest powiązane z translokacją DNA (Rys. 6) i ma miejsce poza rozpoznawana sekwencją (nawet do 1500 pz). Rys. 6. Translokacja DNA przez enzym typu I EcoKI. Genom bakteriofaga T7 posiada na końcu, który jako pierwszy wprowadzany jest do komórki bakteryjnej, miejsce rozpoznawane przez enzym EcoKI. W naturze, genom tego faga penetruje do komórki bakteryjnej dzięki polimerazie RNA (translokacja powiązana jest z transkrypcją). Blokując transkrypcję poprzez dodanie ryfampicyny wykazano, że genom faga może zostać translokowany do cytoplazmy w komórkach kodujących enzym EcoKI. Translokowany DNA staje się wrażliwy na DpnI, który rozpoznaje i tnie DNA zmetylowany poprzez metylazę Dam. Translokacja DNA poprzez EcoKI odbywa się z szybkością 100 pz/sec. 17
19 Sekwencje rozpoznawane przez enzymy typu I są asymetryczne: składaja się z 3-4 nt rozdzielonych niespecyficznym fragmentem (6-8 nt) od dalszych specyficznych (3-4) nt. Związane jest to z budową podjednostki HsdS, którą podzielić można na 3 domeny konserwowane (CD conserved domain) i dwie domeny TRD (target recognition domain) odpowiedzialne za rozpoznawanie specyficznych sekwencj (Rys. 7). S.EcoR124 I CD CD CD N TRD TRD C GAA NNNNNNN RTCG Rys. 7. Struktura podjednostki HsdS. Enzymy typu I rozpoznają sekwencje : NgoAV: GCAN 8 TGC NgoAV T: GCAN 7 GTCA Enzymy typu III rozpoznają asymetryczną sekwencję i metylują tylko jedną nić DNA: EcoP1: AGACC EcoP15: CAGCAG StyLTI: CAGAG Rola systemów RM Systemy RM są powszechnie obecne u prokariota. Przyjmuje się, że obecność systemów RM zabezpiecza komórki bakterii przed inwazją przez obcy DNA, np. wirusowy lub plazmidowy (Rys. 8). Jednak hipoteza obrony komórkowej nie wyjaśnia różnorodności systemów RM, szczególnie takich co rozpoznają sekwencje 8-nukleotydowe, które niezwykle rzadko lub w ogóle nie występują w małych genomach plazmidów i wirusów. Ponadto niektóre komórki kodują jednocześnie kilkanaście systemów RM. 1. Ochronna funkcja systemów RM przed atakiem przez obcy DNA Ryc. 8. Ochronna funkcja systemów RM na przykładzie typu I. Zlokalizowany w komórce bakteryjnej system RM odpowiada za modyfikację komórkowego DNA. Wnikający do komórki fagowy DNA nie posiadający zmetylowanych odpowiednich sekwencji (może posiadać inny wzór metylacji) rozpoznawany jest jako obcy i podlega restrykcji. Ciemne i jasne kropki oznaczają metylację. Wg Tock i Dryden, 2005, rysunek zmodyfikowany. 2. Systemy RM zapewne odgrywają rolę w ewolucji bakterii (fitness) Możliwość pobierania obcego DNA ze środowiska MUSI być regulowana. Mycoplasma pulmonis posiada dwa geny hsds (RM typu I) o różnej specyficzności. Ich ekspresja jest zmienna dzięki inwersji DNA. Hodowle zawierają liczne subpopulacje bakterii, które nie posiadają wykrywalnej aktywności restrykcyjnej i są bardzo podatne na infekcje bakteriofagowe. 18
20 Podobnie geny mod i res systemu RM typu III z Pasteurella haemolytica zawierają powieloną pięcionukleotydową sekwencję: CACAG. Liczba powtórzeń tej sekwencji zmienia się w seryjnych hodowlach i skutkuje zmianą specyficzności lub brakiem aktywności tego systemu RM. Zmienność fazową w ekspresji genów należących do systemu RM typu III odkryto też u Helibacter pylori. Wykazano, że gen res poprzedzony jest homopolimeryczną sekwencją bogatą w cytozyny i że zmienność w długości tego odcinka reguluje ekspresję (lub jej brak) oraz siłę ekspresji genu res (odpowiedzialnego za restrykcję). 3. Aktualnie sądzi się, że systemy RM mogą brać udział w regulacji ekspresji genów oraz w zmienności fazowej. Zmienność fazowa jest procesem adaptacyjnym, polegającym na częstych, odwracalnych zmianach fenotypowych przez komórki bakteryjne. Zjawisku temu mogą podlegać pojedyncze geny bądź też całe grupy genów. Zmienność fazowa prowadzi do zwiększenia różnorodności w obrębie populacji bakterii. Tym, co odróżnia zjawisko zmienności fazowej od mutacji i klasycznej regulacji ekspresji genów jest istnienie epigenetycznego bądź też genetycznego mechanizmu, umożliwiającego dziedziczenie zmienności. Oznacza to, że komórka potomna odziedziczy fenotyp komórki rodzicielskiej, ale odziedziczona zostanie też zdolność do przełączania ekspresji genów. Częstość zmienności fazowej jest procesem losowym, chociaż proces ten może być modulowany przez czynniki zewnętrzne. Wykazano, że zmienność fazowa ekspresji metylaz DNA może mieć wpływ na ekspresję wielu genów. Np. system RM typu III z H. influenzae przez swoją zmienność fazową zmniejsza ekspresję grupy genów kodujących białka będące transporterami oraz wiążącymi niektóre składniki pokarmowe (hemoglobinę, cysteinę, hem), a podnosi poziom ekspresji molekularnych chaperonów i katalizatorów fałdowania białek. Wiele bakteriofagów (i plazmidów) wykształciło mechanizmy ochronne przed systemami RM. Strategie są różne, np. : 1. Brak miejsc rozpoznawanych przez dany enzym restrykcyjny (dotyczy raczej enzymów typu II), np. fagi T3 i T7 nie mają miejsc CCATGG rozpoznawanych przez EcoRII. 2. Ułożenie miejsc restrykcyjnych w orientacji uniemożliwiającej działanie enzymu restrykcyjnego (miejsce EcoP15 w DNA faga T7) 3. Modyfikowanie zasad : - Genomy bakteriofagów T2 i T4 zawierają hydroksymetylocytozynę zamiast cytozyny: takie sekwencje nie są rozpoznawane przez enzymy restrykcyjne; - Fag Mu koduje białko Mom, które tworzy N6-(1-acetamido)adeninę, dzięki czemu jego genom nie ulega restrykcji poprzez EcoKI i EcoBI; 4. Kodowanie specyficznych białek: - Fagi T2 i T4 kodują metylazę Dam (gen metylazy może także znajdować się na plazmidach oraz genomach innych fagów (fagi infekujące B. subtilis); - Stp (26 aa) białko kodowane przez faga T4 przeszkadza w formowaniu się kompleksu enzymatycznego (zmiana konformacji białek); - Produkt genu 0.3 faga T7, zwany także białkiem Ocr (overcoming restriction) wiąże kompleks enzymatyczny EcoKI; - Ard (alleviation of restriction of DNA): podobne do produktu genu 0,3 faga T7, białka kodowane przez plazmidy IncW, które mają wielu gospodarzy. Białka te mają strukturę podobną do podjednostki HsdS i prawdopodobnie uniemożliwiają poprawne tworzenie kompleksu endonukleazy; - Dar (defense against restriction), białko kodowane przez faga P1 prawdopodobnie przeszkadza w translokacji DNA; - Aktywacja metylazy gospodarza: Ral (restriction alleviation): białko kodowane przez faga lambda faworyzuje metylację cząsteczek DNA, które nie uległy restrykcji (Ral aktywuje wyrażanie genów hsds i hsdm); 19
21 - Hydroliza S-adenozylometioniny przez hydrolazę faga T3 (ochrona przed enzymami systemów RM typu IV); - System naprawy DNA: białka Red i Gam kodowane przez bakteriofaga lambda pozwalają na naprawę przeciętego DNA poprzez rekombinację z inną kopią fagowego DNA; 20
22 Bakteriofag T7: Większość bakteriofagów zdolna jest po zainfekowaniu komórki bakteryjnej do wywołania wyłącznioe cyklu litycznego tzn. kończącego się powstaniem potomnych cząstek fagowych. Fagi takie zwane są fagami wirulentymi. Należy do nich coliphage T7 (Rys. 9), wyizolowany w 1945 jako fag namnażający się w E. coli. Aktualnie wiadomo, że fag ten infekuje również bakterie z rodzajów Shigella, Yersinia, Salmonella i Klebsiella. Bakteriofag ten należy do rodziny Podoviridae: fagów posiadających ogonek oraz genom zbudowany z dwuniciowego DNA. Fag zbudowany jest z ikosahedralnej główki (ok. 60 nm średnicy), niekurczliwego ogonka (ok. 20 nm długości i 10 nm szerokości) oraz 6 cienkich włókienek. Receptorem faga jest lipopolisacharyd (LPS) obecny na powierzchni bakterii: bakteriofag nie infekuje większości szczepów E. coli ze środowiska. Fag ten koduje, jako białko wczesne własną polimerazę RNA, która specyficznie rozpoznaje promotory fagowe i przestawia maszynerię syntezy białek na produkcję białek fagowych Mapa genetyczna bakteriofaga przedstawiona jest na Rys. 10. Genomem bakteriofaga T7 jest dwuniciowy liniowy DNA o długości Ryc. 9. Bakteriofag T bp. Sekwencja kodująca zajmuje aż 92 % genomu, reszta to sekwencje sygnałowe. DNA zawiera podobny procent par GC do genomu E. coli (48 %). Genom zawiera tylko 4 sekwencje GATC, rozpoznawane przez metylazę Dam, regulującą wiele procesów w E. coli (statystycznie, w 40 kb powinno być ok. 156 sekwencji GATC). Ryc. 10. Mapa genetyczna bakteriofaga T7. W genomie faga wytypowano ok 56 potencjalnych ramek odczytu ale tylko ok. 20 genów, niezbędnych jest do namnażania faga na szczepach laboratoryjnych. Geny zorganizowane są w trzy bloki/klasy transkrypcyjne: - Klasa I to geny wczesne: ulegające wyrażeniu w pierwszych 8 minutach od wprowadzenia DNA do komórki. Produkty tych genów przygotowują komórkę do produkcji wyłącznie nowych cząstek fagowych. - Klasa II to geny kodujące enzymy biorące udział w metabolizmie DNA, geny te są eksprymowane w 6 do 15 minut od penetracji DNA do komórki. - Klasa III geny kodujące białka strukturalne, biorące udział w tworzeniu cząstek fagowych i lizie (8-25 minut) 21
23 Wejście faga T7 do komórki bakteryjnej przedstawiono na Rys. 11. Fag T7 adsorbuje się na powierzchni komórek rozpoznając LPS. Białka składnikowe rdzenia faga (gp14-16: core proteins) zostają wyrzucone z główki i tworzą kanał niezbędny do transportu DNA do komórki bakteryjnej. Rys. 11. Wejście faga T7 do komórki bakteryjnej. Cykl lityczny trwa około minut. Transfer fagowego DNA do komórki bakteryjnej jest powolny (cały DNA potrzebuje 10 min, aby wejść do komórki bakteryjnej) i zależny od transkrypcji, jedynie 850 pz (umowny lewy koniec genomu, w którym zlokalizowane są geny wczesne) może wejść do komórki bez udziału aktywnej transkrypcji. Ten fragment genomu zawiera 3 promotory rozpoznawane przez polimerazę RNA E.coli. Bakteryjna polimeraza odczytuje geny klasy I (ok 19% genomu). Funkcje 5 białek wczesnych są doskonale znane: - Białko Gp0.3 (zwane także ocr: overcomes classical restriction activity ) inaktywuje system restrykcji modyfikacji typu I (ale bakteriofag pozostaje wrażliwy na systemy typu II i III). Gp0.3 wchodzi w interakcję z EcoKI oraz hydrolizuje AdoMet (donor grup metylowych w reakcji metylacji DNA). Jest to możliwe dzięki udawaniu (mimikra) konformacji 24 nukleotydowej dwunicioweej struktury B DNA. - Białko Gp0.7 jest kinazą serynowo-treoninową odpowiedzialną za fosforylację wielu białek bakteryjnych (np. rybosomalnych). Białko to uniemozliwia także transkrypcję genów zależną od gospodarza poprzez inaktywację komórkowej polimerazy RNA. - Gen gp1 koduje polimerazę RNA T7 specyficznie rozpoznającą promotory fagowe (polimeraza ta ma zastosowanie w biologii molekularnej ponieważ ma bardzo dużą procesywność: pz/sec) - Gp1.3 jest ligazą DNA - GP1.2 jest inhibitorem GTPaz bakteryjnych. Mutacja w genie gp1.2 pozwala fagom na wzrost na komórkach E. coli zawierających plazmid F. Ogólnie produkty genów wczesnych mają za zadanie wyłączenie transkrypcji komórkowego DNA oraz zablokowanie aktywności białek bakteryjnych, które mogłyby zaburzyć proces infekcji fagowej. Ekspresja genów klasy II i III (81% genomu) ma miejsce dzięki polimerazie RNA faga T7. Wyrażane są 24 geny biorące udział w metabolizmie DNA np. gp3 i gp6, kodujące endonukleazę (Gp3) oraz egzonukleazę (Gp6), które degradują bakteryjny DNA (nie wiadomo, w jaki sposób aktywności tych białek jest kontrolowana in vivo tak, aby genom faga nie był degradowany) uwalniając w ten sposób nukleotydy, które są wykorzystywane podczas syntezy fagowego DNA (80% nukleotydów w potomstwie faga pochodzi z degradacji genomu gospodarza). Białko Gp2 jest inhibitorem bakteryjnej polimerazy RNA i jest niezbędne podczas pakowania cząstek fagowych. Wśród białek eksprymowanych w tym procesie znajduje się także polimeraza DNA faga T7. Klasa III genów, to geny zaangażowane w morfogenezę faga, pakowanie DNA oraz lizę komórki bakteryjnej. Geny te są transkrybowane z bardzo silnych promotorów rozpoznawanych 22
24 przez fagową polimerazę. Lizozym (Gp3.5) kodowany przez faga nie jest zaangażowany w lizę komórek ale w uwalnianie cząstek fagowych z resztek bakteryjnych. Fag nie koduje typowej kasety litycznej. Gen gp17.5 jest najprawdopodobniej holiną. Rozpoczęcie transkrypcji genomu fagowego przez polimerazę T7 pozwala na wprowadzenie całego fagowego DNA do komórki gospodarza. Podczas ekspresji genów klasy II ma także miejsce replikacja fagowego DNA. Replikacja DNA faga T7 jest procesem niezmiernie złożonym, fag koduje własne białka, biorące udział w tym procesie: polimerazę DNA, primazę oraz helikazę (Rys. 12). Rys. 12. Replikacja genomu faga T7- kompleks enzymatyczny a. Polimeraza DNA T7 jest kompleksem zbudowanym z dwóch podjednostek: produktu genu gp5 oraz czynnika Trx (tioredoksyna) kodowanego przez E. coli. Czynnik Trx ma wpływ na procesywność polimerazy. Polimeraza łączy się z kompleksem białkowym kodowanym przez gen gp4. b. Białko Gp4 ma dwie aktywności: helikazy i prymazy. Aktywność helikazy pozwala na rozwinięcie rodzicielskiego DNA, prymaza pozwala na syntezę nt startera RNA potrzebnego do rozpoczęcia replikacji nici opóźnionej. Białko to działa jako heksamer. Białko kodowane przez gen gp2.5 jest białkiem wiążącym jednoniciowy DNA obecny w widełkach replikacyjnych. W wyniku replikacji powstaje tzw. bubble (widoczny pod mikroskopem elektronowym, Rys. 13). W hodowli płynnej przejaśnienie widać już po 20 minutach hodowli (gwałtowny spadek OD 600 ), a miano faga (liczba wirionów) zwiększa już po 15 minutach (Rys. 14). Rys. 13. Replikacyjny bubble. Rys. 14. Łysinki bakteriofaga T7. A: 12 godzinach, B: 24 godzinach, C: 50 godzinach wielkie łysinki z przejrzystym środkiem. Fag szybko ewoluuje, mapy restrykcyjne fagów pobranych w różnych miejscach łysinki różnią się. Infekcja abortywna Istnieje wiele przykładów zahamowania infekcji fagowej, poprzez różne elementy obecne w komórce bakteryjnej. Dotyczy to np. zajęcia/zmiany w strukturze/ braku ekspresji receptorów zewnątrz- i wewnątrzkomórkowych (obecnosć profaga) specyficznych dla danego faga, ekspresji represorów (niemożności superinfekcji), cytoplazmatycznej degradacji genomowego DNA 23
25 bakteriofaga (restrykcja DNA). Bakteriofag w tym przypadku nie zaczyna cyklu litycznego. Podczas infekcji abortywnej przerwany zostaje rozpoczęty cykl replikacyjny bakteriofaga. Blokowanie replikacji fagowej przez elementy pozachromosomalne jest rozpowszechniona. Np. fag T4 nie namnaża się na komórkach E. coli zlizogenowanych fagiem lambda: pierwsze 10 minut infekcji przebiega klasycznie, a następnie gwałtownie zatrzymuje się synteza DNA oraz białek. Wykazano, że w tym przypadku zlizogenizowane bakterie nie są w stanie utrzymać odpowiedniego stężenia Mg 2+ wewnątrz komórki. Prawdopodobnie, w podobny sposób dochodzi do infekcji abortywnej wywołanej poprzez system rex, kodowany przez faga lambda (białka o nieznanej funkcji, zlokalizowane w błonie komórkowej). Wiele wirusów nie namnaża się w komórkach E. coli posiadających plazmid F lub F`. Są to np. fagi: tau, T7, phii, phiii, W13 (a np. fag T3 jest niewrażliwy na obecnosć F`). Plazmid F (fertility factor): to element (ok. 100 kpz) pozwalający bakterii na syntezę pilusów niezbędnych do koniugacji (F` to plazmid F, który zawiera jakąś część chromosomu E. coli). Infekcja abortywna na przykładzie faga T7: Najintensywniej badana była infekcja abortywna faga T7, ale fenomen nie został wyjaśniony do końca i jest mnóstwo kontrowersji dotyczących mechanizmu tego zjawiska. Na szalkach można zaobserwować mniejszą liczbę łysinek (ok. 100 do 100 milionów razy), łysinki mogą mieć (w zależności od użytego szczepu laboratoryjnego) mniejszą średnicę. Cykl lityczny zaczyna się klasyczną absorpcją, DNA faga nie jest degradowany jak w przypadku ataku poprzez systemy RM. Białka klasy I są prawidłowo syntetyzowane (powstaje np. T7 polimeraza RNA), lecz nagle następuje represja replikacji DNA. Jakość syntetyzowanego mrna jest poprawna, jednakże mrna klasy III mają często nieprawidłową długość. Długo sądzono, że ma to związek z niekompletnym wejściem genomowego DNA faga do komórek bakteryjnych, jednakże infekcja abortywna ma miejsce nawet po wprowadzeniou DNA do komórek bakteryjnych metoda transfekcji. Nieprawidłowa jest natomiast translacja mrna białek klasy II i III, syntetyzowane są tylko w 10 %. Nie wyjaśniono mechanizmu, który zatrzymuje translację. Badania wykazały, że zarówno przy zastosowaniu polimerazy RNA faga jak i gospodarza, synteza białek zatrzymuje się w 9-12 minut po infekcji. Być może infekcja abortywna ma związek z kontrolą transkrypcji lub replikacją. Wykazano, że w komórkach E. coli zawierających plazmid F, synteza DNA gospodarza zatrzymuje się całkowicie podczas infekcji, a w komórkach pozbawionych plazmidu F spada tylko o 50%. Plazmidy F zmutowane w genach pifa i pifb (phage inhibition factor) pozwalają na namnażanie faga T7. PifA wykazuje podobieństwo sekwencji do transporterów typu ABC, lecz jego rola w komórce bakteryjnej jest nieznana. Nie przypisano także temu białku żadnej funkcji biochemicznej. Inna hipoteza dotyczy zaburzonej przepuszczalności błony: wykazano, że ONPG (chemiczny chromogenny substrat -galaktozydazy) dyfunduje wyłącznie z komórek z plazmidem F. Z komórek wypływa ATP oraz inne fosforylowane cząsteczki. Takie komórki nie są w stanie akumulować glutaminy i proliny oraz mają problem z utrzymaniem odpowiedniego stężenia potasu. Wyizolowano mutanty bakteriofaga T7 zdolne do przeżycia w komórkach E. coli zawierających plazmid F. Mutacje dotyczą genów gp1.2 i gp10 (białko kapsydu). Zmutowane bakteriofagi T7 nie są wrażliwe na uwalnianie nukleotydów z komórki. Kiedy dochodzi do infekcji abortywnej, komórka umiera, ale nie lizuje: praktycznie nie pojawiają się nowe cząstki fagowe. Nowo powstałe fagi nie nabywają żadnych cech, które pozwoliłyby im na namnażanie się na komórkach E. coli F`. W następnym cyklu infekcyjnym nadal będą ulegać infekcji abortywnej w obecności plazmidu F`. 24
26 Wybrana literatura: 1. Beck PJ, Molineux IJ. Defective transcription of the right end of bacteriophage T7 DNA during an abortive infection of F plasmid-containing Escherichia coli. J Bacteriol. 1991;173(3): Bickle TA, Krüger DH.Biology of DNA restriction. Microbiol Rev. 1993; 57(2): Dryden DT, Davies GD, Martin I, Powell LM, Murray NE, Ellis DJ, Berge T, Edwardson JM, Henderson RM.The assembly of the EcoKI type I DNA restriction/modification enzyme and its interaction with DNA. Biochem Soc Trans. 1999; 27(4): Duckworth DH, Glenn J, McCorquodale DJ. Inhibition of bacteriophage replication by extrachromosomal genetic elements. Microbiol Rev. 1981; 45(1): García LR, Molineux IJ. Incomplete entry of bacteriophage T7 DNA into F plasmid-containing Escherichia coli. J Bacteriol. 1995l; 177(14): Krüger DH, Bickle TA. Bacteriophage survival: multiple mechanisms for avoiding the deoxyribonucleic acid restriction systems of their hosts. Microbiol Rev Sep;47(3): Loenen WA.Tracking EcoKI and DNA fifty years on: a golden story full of surprises. Nucleic Acids Res. 2003; 31(24): Murray NE.Type I restriction systems: sophisticated molecular machines (a legacy of Bertani and Weigle). Microbiol Mol Biol Rev. 2000; 64(2): Studier FW. Bacteriophage T7. Science. 1972; 176(33): Tock MR, Dryden DT. The biology of restriction and anti-restriction. Curr Opin Microbiol. 2005; 8(4):
27 Część praktyczna ĆWICZENIE 3 Materiały Szczepy bakteryjne - E. coli Top10( - ) s, r - m -, F- mcra (mrr-hsdrms-mcrbc) 80 LacZ M15 lacx74 deor reca1 arad139 (ara-leu) 7697 galu galk lambda^-rpsl enda1 nupg - E. coli TopF (F proab laciqz)m15 Tn10 (Tet r ) - E. coli K12 ( - ) s koduje system restrykcji-modyfikacji typu I EcoKI, który rozpoznaje sekwencję AAC(N 6 )GTGC lub GCAC(N 6 )GTT - E. coli Top10 (pno12): koduje sklonowany na plazmidzie puc19 system restrykcji-modyfikacji typu I NgoAV (z Neisseria gonorrhoeae), który rozpoznaje sekwencję GCAN 6 TGC - Haemophilus influenzae Rd30 Bakteriofagi (miana fagów zostaną podane podczas ćwiczeń) - zawiesiny faga lambda: vir - zawiesina faga T7 Podłoża - LB płynne - LB półpłynne - LB stałe - BHI stałe i półpłynne Roztwory - bufor SM - chloroform Wykonanie 1. Liza i lizogenia - pobrać materiał z 5 kolonii z posiewu redukcyjnego ćw.2, pkt 4, test 1; - wsiać do 0,25 ml płynnego LB (w eppendorfie) i inkubować 3 h w temp. 37 o C; - nakroplić 5 l hodowli na murawkę szczepu E. coli Top10 wysianego wcześniej w agarze półpłynnym na podłoże stałe; - inkubować h w temp. 37 o C; 2. Izolacja lizogenów faga HP1 - materiał pobrać z 5 kolonii z posiewu redukcyjnego ćw. 2. pkt 4, test 2; - hodowlę lizogenów prowadzić w 0,25 ml płynnego BHI z NAD i heminą (w eppendorfie) i inkubować 3 h w temp. 37 o C ; - nakroplić 5 l hodowli na murawkę szczepu H. influenzae Rd30, wysianego wcześniej na podłoże stałe BHI uzupełnione NAD i heminą; - inkubować h w temp. 37 o C; 3. Oznaczanie miana bakteriofaga vir - obliczyć miano zawiesiny fagowej tj. liczba jednostek tworzących łysinki w 1 ml (pfu: plaque forming units) według podanego niżej wzoru: pfu (na ml) = N R 10 N = liczba łysinek R = odwrotność rozcieńczenia 10 = przeliczenie na 1 ml - w teście kroplowym (w przypadku uzyskania policzalnej liczby łysinek) liczbę faga w 1 ml obliczyć ze wzoru: pfu = N R 200 (200 to przeliczenie na 1 ml) 26
28 4. Abortywna (poronna) infekcja faga T7 - wykonać rozcieńczenia faga T7 (10-1, 10-2, 10-3, 10-4, 10-5, 10-6 ) w buforze SM; - metodą płytek dwuwarstwowych wysiać po 0,1 ml rozcieńczeń 10-5 i 10-6 z hodowlą nocną szczepu E. coli Top10 (0,2 ml); - metodą płytek dwuwarstwowych wysiać po 0,1 ml rozcieńczeń 10-2 i 10-3 z hodowlą nocną szczepu E. coli Top10 F (0,2 ml;) - inkubować h w temp. 37 o C; - UWAGA: rozcieńcznia zostaną podane podczas ćwiczeń (zależą od miana faga w zawiesinie początkowej). 5. Zjawisko restrykcji-modyfikacji DNA - wykonać rozcieńczenia faga vir (10-1, 10-2, 10-3, 10-4, 10-5, 10-6 ) w buforze SM; - 0,1 ml z rozcieńczeń 10-5 i 10-6 wysiać metodą płytek dwuwarstwowych na szczep E. coli Top10; - 0,1 ml z rozcieńczeń 10 0 wysiać metodą płytek dwuwarstwowych na szczep E. coli K12; - 0,1 ml z rozcieńczeń 10 2 i 10 3 wysiać metodą płytek dwuwarstwowych na szczep E. coli Top10 (pno12); - inkubować h w temp. 37 o C; - UWAGA: rozcieńcznia zostaną podane podczas ćwiczeń (zależą od miana faga w zawiesinie początkowej). 27
29 ĆWICZENIE 4 Część praktyczna Materiały Szczepy bakteryjne: - E. coli Top10( - ) s, r - m -, F- mcra (mrr-hsdrms-mcrbc) 80 LacZ M15 lacx74 deor reca1 arad139 (ara-leu) 7697 galu galk lambda^-rpsl enda1 nupg - E. coli TopF (F proab laciqz)m15 Tn10 (Tet r ) - E. coli K12 ( - ) s koduje system restrykcji-modyfikacji typu I EcoKI, który rozpoznaje sekwencję AAC(N 6 )GTGC lub GCAC(N 6 )GTT - E. coli Top10 (pno12): koduje sklonowany na plazmidzie puc19 system restrykcji-modyfikacji typu I NgoAV (z Neisseria gonorrhoeae), który rozpoznaje sekwencję GCAN 6 TGC - E. coli Top10 (psmrx): koduje sklonowany na plazmidzie pacyc184 system NgoAV z punktową delecja w genie hsds: NgoAV T (z Neisseria gonorrhoeae), który rozpoznaje sekwencję GCAN 7 GTCA Bakteriofagi: - zawiesiny fagów lambda: Top10, No12 oraz SMRX Podłoża - LB płynne - LB półpłynne - LB stałe Roztwory - bufor SM - chloroform - roztwór białka docelowego (stężenie 100 g/ml) do Phage Display w 0,1 M NaHCO 3 (ph 8,6) Wykonanie 1. Liza i lizogenia: odczytać wyniki testu 1 i 2 z ćw Abortywna (poronna) infekcja faga T7 - obliczyć miana fagów T7 uzyskane na obu szczepach; - pobrać ezą 2-3 łysinki ze szczepu E. coli Top10F i zawiesić w 0,5 ml buforu SM; - dodać kroplę chloroformu (40 l) i energicznie wytrząsać przez kilka minut (ewentualnie osadzić chloroform poprzez wirowanie); - następnie wykonać rozcieńczenia 10-1, 10-2, 10-3 w buforze SM; - 0,1 ml odpowiedniego rozcieńczenia faga T7 (10-2 i 10-3 ) zmieszać z 0,2 ml nocnej hodowli szczepu E. coli Top10 i wysiać metodą płytek dwuwarstwowych, natomiast rozcieńczenia 10-1, 10-2 wysiać na nocną hodowlę szczepu E. coli Top10 F metodą płytek dwuwarstwowych; - inkubować h w temp. 37 C; 3. Zjawisko restrykcji-modyfikacji DNA - obliczyć miana fagów vir namnożonych na obu szczepach: E. coli Top10 i E. coli K12 oraz E. coli (pno12); - pobrać ezą kilka łysinek faga Top10 i zawiesić w 0,5 ml buforu SM; 28
30 - dodać 1 kroplę chloroformu i energicznie wytrząsać przez kilka minut; - analogicznie postąpić z fagiem K12 i No12 ; - zawiesiny fagowe rozcieńczyć 10-1,10-2, 10-3 ; - 0,1 ml z rozcieńczenia 10-2, 10-3 wysiać metodą płytek dwuwarstwowych na szczepy E. coli Top10, E. coli No12 i E. coli K12; - inkubować h w temp. 37 o C; 4. Test krzyżowy (ang. cross - streaking) - na szalce ze stałym LB nanieśc pipetą linię (200 l) z każdego faga : Top10, No12 oraz SMRX (nie dotykając szalki); - odczekać aż materiał wsiąknie w podłoże; - nanieść ezą szczepy E. coli: Top10 i E. coli: Top10 (pno12), E. coli: Top10 (psmrx) oraz E. coli: K12 przecinając powierzchnię z fagiem i pamiętając o każdorazowym opalaniu ezy przed naniesieniem na następną rysę z materiałem fagowym; - inkubować h w temp. 37 o C; 5. Phage Display - rozprowadzić 1,5 ml roztworu białka docelowego na polipropylenowej szalce tak, aby pokryć dokładnie całą jej powierzchnię; - inkubować szalkę przez noc w temperaturze 4 o C (w wilgotnym środowisku); 29
31 Schemat prezentujący kolejne etapy procedury, otrzymania biblioteki fragmentów restrykcyjnych faga HP1 w wektorze plazmidowym pbluescript KS II (+), realizowanej podczas kolejnych ćwiczeń od 5 do
32 ĆWICZENIE 5 Metody indukcji profagów Część teoretyczna Bakteriofag lambda Bakteriofag jest prototypem fagów łagodnych. Odkryty został w 1951 w szczepie E. coli K12. Genom tego faga upakowany jest w główce w formie dwuniciowego liniowego DNA o długości pz. Potencjalnie fag koduje aż 71 białek i funkcja większości z nich jest dobrze znana. Ekspresja genów jest regulowana poprzez proteolizę czynników transkrypcyjnych. Geny kodujące białka potrzebne na określonym etapie rozwoju zgrupowane są w bloki transkrypcyjne: blok kodujący elementy regulatorowe, zwany rejonem Rys. 15. Bloki transkrypcyjne w genomie faga. odpornościowym (antyrepresor Cro, represor CI, białko N, białka CII i CIII), blok z genami biorącymi udział w replikacji DNA (geny o i p), blok genów biorących udział w lizie (s, r, r z ), rekombinacji (int i xis), oraz blok z genami strukturalnymi (Rys. 15 i 16). Podczas infekcji fag wstrzykuje (posiada kurczliwy ogonek) do cytoplazmy liniową cząsteczkę DNA. Lepkie końce fagowego DNA składają się z komplementarnych 12 nukleotydowych sekwencji cos, dzięki którym dwa enzymy bakteryjne, ligaza oraz gyraza, przekształcają liniowy DNA w kolistą superhelikalną cząsteczkę DNA. Rys. 16 Mapa genetyczna bakteriofaga. 31
33 Bakteriofag nie koduje własnej polimerazy, lecz wykorzystuje bakteryjną polimerazę RNA. Fag ma 7 promotorów odczytywanych na różnych etapach cyklu: Dwa silne promotory odpowiedzialne są za wyrażanie genów wczesnych: - P R kieruje ekspresją genów niezbędnych do replikacji DNA, antyrepresora Cro, aktywatora transkrypcji CII i antyterminatora Q - P L kieruje ekspresją genów niezbędnych do rekombinacji DNA, antyterminatora N i białka stabilizującego CIII Dwa promotory odpowiedzialne są za kontrolę ekspresji represora CI: - P RE pozwala na ekspresję represora CI, aby zainicjować cykl lizogeniczny - P RM pozwala na ekspresję represora CI, aby utrzymać lizogenię. Trzy promotory regulują ekspresję genów potrzebnych w cyklu litycznym lub lizogennym: - P R' kieruje ekspresją genów niezbędnych do lizy komórki oraz genów strukturalnych (cykl lityczny) - P I pozwala na ekspresję genu int (integraza) - P AQ pozwala na syntezę antysensownego DNA, który blokuje transkrypcję mrna genu q (antyterminator). Rys. 17. Sekwencje operatorowe (17 nt) rozpoznawane przez czynniki transkrypcyjne nakładają się na promotory fagowe P R oraz P L Promotor prawy w genomowym DNA bakteriofaga, odpowiedzialny za produkcję białka Cro. We wczesnym etapie infekcji, transkrypcja zapoczątkowana jest z promotora P L, który umożliwia wyłącznie powstanie mrna obejmującego gen kodujący białko N oraz z promotora P R, który umożliwia powstanie mrna obejmujący gen cro (Rys 17). Spowodowane jest to obecnością terminatorów transkrypcji. Białko N jest antyterminatorem transkrypcji: rozpoznaje sekwencje nutl lub nutr (Nutilisation) na powstającym mrna, ponieważ dzięki tym sekwencjom mrna przybiera rozpoznawaną przez N strukturę II rzędową. Dzięki kompleksowi pomiędzy białkiem N, bakteryjną polimerazą RNA, sekwencjami nut, RNA oraz bakteryjnym białkom Nus, polimeraza RNA nie zatrzymuje się na sekwencjach terminatorowych i w ten sposób mrna powstały z promotorów P L i P R jest wydłużony. Następuje transkrypcja genów kodujących białka nazwane opóźnionymi wczesnymi : CIII, CII, Q, genów związanych z rekombinacją (gam, red) oraz genów związanych z replikacja DNA (o i p). CII jest czynnikiem transkrypcyjnym, który aktywuje dwa promotory: P I (transkrypcja int) i P RE (promotor białka CI) bezpośrednio łącząc się z operatorami nakładającymi się na te promotory. CII jest białkiem niestabilnym, podatnym na działanie proteaz bakteryjnych typu Hfl (Hfl- high frequency of lysogeny). Białko CII jest stabilizowane poprzez białko CIII. Bez białka CIII czas półtrwania białka CII wynosi ok 1 minuty, w obecności CIII 5 minut. Nadekspresja białka CII jest toksyczna dla komórki gospodarza. 32
34 Białko CII aktywuje także trzeci promotor: P AQ odpowiedzialny za syntezę antysensownego RNA do genu Q. Białko Q jest antyterminatorem pozwalający na transkrypcję białek litycznych (s i r) i strukturalnych. Białko CII faworyzuje więc lizogenię, uniemożliwiając ekspresje białek potrzebnych w cyklu litycznym. Mutanty bakteriofaga w genach C (clear) tworzą przejrzyste łysinki na murawce E. coli. Lizogenia Aktywność represora CI oraz integrazy Int są wystarczające, aby bakteriofag wszedł w stan lizogenii. Białko CI jest represorem bakteriofaga. Jest to niewielkie białko o wielkości 26 kda, które można podzielić na dwie domeny: część aminowa łączy się z operatorem (5 -helis) oraz część karboksylowa odpowiedzialna za łączenie się białek w aktywne dimery a także oddziaływanie dimerów pomiędzy sobą. Białko CI działa jako dimer, ale łączy się także w tetramery i oktomery. CI przyłącza się do operatorów OR1 i OR2 (tetramer) blokując ekspresję z P R i aktywując Rys. 18. Oddziaływanie represorów CI z operatorami. własną ekspresję z promotora P RM. Podobnie CI blokuje transkrypcję z promotora P L przyłączając się do OL1 i OL2. CI przyłączone do operatorów OR i OL oddziałują między sobą (oktomer) wyginając DNA. Powstanie pętli DNA faworyzuje przyłączenie się CI także do OR3 i OL3 (Rys. 18). Następuje wtedy represja promotora P RM (ok. 50 % miejsc OR3 jest zajętych przez represor CI w stanie lizogenii). W cyklu lizogennym, genom bakteriofaga integruje się z chromosomem bakteryjnym poprzez rekombinację specyficznych sekwencji: attp (fag) i attb (bakteria). W rekombinacji genomu faga lambda do chromosomu bakteryjnego biorą udział białka fagowe: produkty genów int i xis oraz białko bakteryjne IHF (integration host factor). Tylko gen ci może być transkrybowany w stanie lizogenii, dzięki promotorowi P RM (P RE wymaga aktywacji przez CII, a CII nie jest transkrybowany, ponieważ zablokowany jest P R ). O tym że zjawisko lizogenii jest powszechne może świadczyć fakt, że w roku 2003 zostało zsekwencjonowanych około 83 genomów bakteryjnych i w około 50 znaleziono profagi (lub jakieś resztki 230 sekwencji fagowych). W genomie E. coli K-12 odkryto aż 4 niekompletne sekwencje profagów lambdoidalnych. W genomie Neisseria gonorrhoeae znaleziono 7 fagów (dsdna i ssdna) lub niekompletnych sekwencji fagowych. Cykl lityczny: Białko Cro jest anty-represorem (represor represora). Białko to zbudowane jest z 66 aa i rozpoznaje sekwencje operatorowe obecne w P R i P L. Dzięki temu białku następuje blokada ekspresji genu ci (poprzez blokadę promotora P RE ) ale nie blokuje ekspresji genu antyterminatora Q (blokując promotor P AQ ). Akumulacja białka Cro redukuje progresywnie wczesną transkrypcję. Białko Q jest antyterminatorem transkrypcji pozwalającym na wydłużenie mrna i transkrypcję genów kodujących białka lityczne: holiny (s) i endolizyny (r), a także genów kodujących białka strukturalne. Białko Q łączy się z DNA powyżej promotora pr i oddziałuje z polimerazą RNA. Cykl lityczny trwa około 35 minut w 37º C i z jednej zainfekowanej komórki powstaje ok. 100 potomnych cząstek fagowych. 33
35 Liza czy lizogenia? Białka Cro i CI mają bardzo podobną budowę w domenie rozpoznającej DNA, co nie jest dziwne, ponieważ rozpoznają te same sekwencje DNA. Białka te współzawodniczą w przyłączaniu się do tych samych sekwencji operatorowych, ale z innym powinowactwem (Rys. 19): Cro łączy się z preferencyjnie z OR3 > OR2/OR1 oraz OL3 > OL2/OL1 Represor także łączy się z każdą sekwencją z innym powinowactwem: OR1 > OR2/OR3 oraz OL1 > OL2/OL3 W zależnościod tego, które białko szybciej rozpozna swój operator (co zależy od stężenia danego białka) następuje synteza Cro, O P i innych i fag wchodzi w cykl lityczny lub wygrywa CI i fag wchodzi w stan lizogenii. Co faworyzuje lizę? - Fakt, że Cro jest białkiem wczesnym a CI białkiem opóźnionym ; - Niski poziom camp (dobre warunki pokarmowe dla bakterii): kompleksy proteaz bakteryjnych Hfl szybciej degradują CII. Lizogenia jest korzystna dla faga, który może przeżyć w niekorzystnych warunkach (potrzebuje maszynerii gospodarza do namnażania); - Mutacje w genach kodujących IHF (integration host factor); Rys. 19. Liza czy lizogenia? Co faworyzuje lizogenię? - Wysokie stężenie CII reguluje wejście w cykl lizogeniczny; - Infekcja wygłodzonych bakterii faworyzuje lizogenię. Wysoki poziom camp (złe warunki pokarmowe dla bakterii): Kompleks CAP/CAP site/polimeraza faworyzuje ekspresję CI; - Bakteria atakowana przez kilka fagów; - Mutacje w genach kodujących proteazy Hfl (odpowiedzialne za degradację CII); - Lizogenia zachodzi w 1/1000-1/100 przypadków; W przypadku bakteriofaga superinfekcja jest niemożliwa: represor CI blokuje ekspresję wszystkich genów posiadajacych sekwencje OR lub OL (działa in trans). Miejsce attb, niezbędne do rekombinacji genomowego DNA faga, nie istnieje. Rys. 20. Indukcja cyklu litycznego. 34
36 Indukcja/cykl lityczny Spontaniczna indukcja profaga jest zjawiskiem bardzo rzadkim: <1/ profagów przechodzi w cykl lityczny. Indukcja ma miejsce dzięki inaktywacji białka represorowego CI, które ulega proteolizie poprzez bakteryjne proteazy. Kluczowym białkiem w tym mechanizmie jest białko RecA, które rozpoznając jednoniciowy DNA aktywuje wiele proteaz bakteryjnych. Celem białka RecA jest inaktywacja poprzez proteolizę represora systemu naprawczego SOS, białka LexA (Rys. 20). Spadek odpowiedniego stężenia represora CI w zlizogenizowanej komórce skutkuje natychmiastową transkrypcją białek fagowych biorących udział w cyklu litycznym. Promotory PL i PR nie są już reprymowane, rusza ekspresja antyrepresora Cro. Indukcja faga jest nieodwracalna. W praktyce zaindukować cykl lityczny mogą: światło UV, czynniki mutagenne, wysoka temperatura. Bakteriofaga CIts857Sam7 zaindukujemy temperaturą: mutacja CIts857 powoduje inaktywację represora w 42 C (zmiana konformacji). Bakteriofaga HP1 zaindukujemy używając mitomycyny C (Rys. Rys. 21. Mitomycyna C. 21). Mitomycyny są produktami naturalnymi, izolowanymi z promieniowców Streptomyces lavendulae i S. caespitosa. Mitomycyna C używana jest w chemioterapii wielolekowej raka: żołądka, piersi, płuca, pęcherza moczowego, szyjki macicy, okolic głowy i szyi oraz w innych lokalizacjach. Mitomycyna C wiąże się kowalencyjnie z DNA. Jest to związek wysoce toksyczny, mutagenny (alkilujący), który generuje powstawanie wolnych rodników. Bakteriofag HP1/S2 Jest to bakteriofag łagodny, infekujący Haemophilus influenzae. Genom tego bakteriofaga zbudowany jest z dwuniciowego liniowego DNA z lepkimi końcami. Genom składa się z pz i zawiera, podobnie jak genom gospodarza, 39% par GC. Bakteriofag koduje potencjalnie 41 białek. Nie wszystkie funkcje potencjalnych ORF są znane (Rys. 22). Rys. 22. Mapa genetyczna genomu bakteriofaga HP1. Wejście w cykl lityczny lub w stan lizogenii regulowane jest przez dwa białka: Cox (cro-like) i represor typu CI. Białka te są pod kontrolą dwóch głównych promotorów bakteriofaga, od których zaczyna sie proces transkrypcji w dwóch przeciwstawnych kierunkach: promotora lewego (CI) i prawego (cox). 35
37 Promotory te są podobnie rozpoznawane przez bakteryjną polimerazę RNA (mają podobną siłę). Białko Cox blokuje P L, łącząc się z sekwencjami operatorowymi usytuowanymi tuż za promotorem P L, blokuje ekspresję genu ci. Podobnie białko CI, łącząc się z sekwencjami operatorowymi, blokuje ekspresję genu cox (Rys. 23). W przypadku bakteriofaga HP1 liza zachodzi z tą sama częstością jak lizogenia. Wydaje się, że gen ci jest częścią Rys. 23. Organizacja promotorów wczesnych bakteriofaga HP1. operonu, w którego skład wchodzi także gen int: ekspresja int ma miejsce także w zlizogenizowanych komórkach. ORF5 jest prawdopodobnie białkiem pełniącym funkcje lambdowego białka CII. W genomie faga znajdują się 2 inne promotory rozpoznawane przez bakteryjną polimerazę RNA, np. promotor orf14. Białko Cox bierze także udział w wycinaniu genomu faga podczas indukcji. Białko to ma aktywność wycinazy. Proteaza RecA nie aktywuje proteolizy represora faga HP1: mechanizm indukcji faga HP1 poprzez mitomocynę C w H. influenzae jest nieznany. Wybrana literatura: 1. Campbell A. The future of bacteriophage biology. Nature Reviews Genetics 4, ; 2. Dodd IB, Shearwin KE, Egan JB. Revisited gene regulation in bacteriophage lambda. Curr Opin Genet Dev. 2005; 15(2): Gottesman ME, Weisberg RA. Little lambda, who made thee? Microbiol Mol Biol Rev. 2004; 68(4): Esposito D, Fitzmaurice WP, Benjamin RC, Goodman SD, Waldman AS, Scocca JJ.The complete nucleotide sequence of bacteriophage HP1 DNA. Nucleic Acids Res. 1996; 24(12): Esposito D, Scocca JJ. Purification and characterization of HP1 Cox and definition of its role in controlling the direction of site-specific recombination. J Biol Chem. 1997; 272(13):
38 Część praktyczna Materiały Szczepy bakteryjne - E. coli NM 258 ( ci857 S7 ) - Haemophilus influenzae Rd30 (HP1) - Haemophilus influenzae Rd30 (HP1 - ) Odczynniki A. Indukcja lizogenów HP1 i ci857 S7 - bufor SM: 50 mm TrisCl (ph 7,5), 100 mm NaCl, 8 mm MgSO 4, 2% żelatyna (w/v) - RNaza: 10 mg/ml - DNaza: 1 mg/ml - chloroform - mitomycyna C: 1mg/ml - bufor do wytrącania: 2,5 M NaCl, 20% PEG 8000 B. Phage Display - TBST: 50 mm Tris-HCl, ph 7,5, 150 mm NaCl, 0,1% Tween-20 - bufor blokujący: 0,1 M NaHCO 3, 0,02% NaN 3, 3% mleko odtłuszczone, ph 8,6 - bufor do wymywania: 0,2 M glicyna, 0,5% mleko odtłuszczone, ph 2,2-1M TrisHCl ph 9,1 Wykonanie 1. Abortywna (poronna) infekcja faga T7 c.d. - porównać miana fagów i wygląd łysinek wyrosłych na obu szczepach po h inkubacji w temp. 37 o C; 2. Zjawisko restrykcji - modyfikacji DNA c.d. - obliczyć miana fagów i poziom restrykcji; - zaobserwować liczbę i miejsce pojawienia się łysinek; 3. Indukcja faga ci857 S7 - przenieść trzygodzinną hodowlę E. coli NM 258 do wytrząsanej łaźni o temp. 45 C - inkubować 20 min; - przenieść hodowlę do wytrząsarki (temp. 37 C) i inkubować 2 h; - zwirować hodowlę bakteryjną w temp. 4 C, przy rpm, 10 min; - zlać supernatant, usunąć pipetą resztki podłoża i zawiesić osad w 1 ml buforu SM - przenieść zawieszony osad po 500 l do dwóch eppendorfówek; - do każdej eppendorfówki dodać kroplę chloroformu (POD WYCIĄGIEM) - wytrząsać zawartość 10 min; - odwirować resztki bakterii w temp. 4 C, rpm, 10 min, przenieść supernatant do 2 eppendorfówek; - dodać RNazę do końcowego stężenia 10 g/ml i DNAzę do stężenia 1 g/ml; - inkubować 10 min w temp. 37 C; - przechowywać w temp. 4 C; 37
39 4. Indukcja lizogenów faga HP1 - do 20 ml 2-godzinnej hodowli Haemophilus influenzae Rd30 (HP1) dodać mitomycynę C do końcowego stężenia 35 ng/ml; - kontynuować hodowlę przy intensywnym wytrząsaniu i w ciemności 2 h (do wystąpienia lizy); - dodać do kolbki 0,4 ml chloroformu (POD WYCIĄGIEM); - wytrząsać 10 min w temp. 37 C; - odwirować resztki bakterii w temp. 4 C, rpm, 10 min; - podzielić supernatant na 2 części i każdą przelać do 2 probówek wirowniczych; - dodać RNazę do stężenia 10 g/ml i DNazę do stężenia 1 g/ml; - inkubować w temp. 37 C, 15 min; - dodać równą objętość (10 ml) roztworu do wytrącania i inkubować h, w temp. 4 C; 5. Phage Display (część teoretyczna na stronie 74) - wylać roztwór białka z inkubowanej szalki; - napełnić szlakę buforem blokującym i inkubować przez 1 h w temp. 4 C z delikatnym mieszaniem; - zlać bufor blokujący i przemyć szalkę 6 razy buforem TBST; - rozcieńczyć 10 l faga z biblioteki fagowej M13 w buforze TBST do miana , po czym wylać go na szalkę i inkubować przez 1 h w temp. pokojowej; - przemyć szalkę 10 razy buforem TBST; - wylać na szalkę 1 ml buforu do wymywania fagów i inkubować 10 min w temp. pokojowej z delikatnym mieszaniem; - wymyte fagi zebrać do probówki eppendorfa i dodać 150 l 1 M TrisHCl ph 9,1; - zebrane bakteriofagi przechowywac w temp. 4 C; 38
40 ĆWICZENIE 6 Metody izolacji DNA z bakteriofagów i ich gospodarzy Część teoretyczna Ogólne zasady izolacji DNA. Czysty DNA to taki, który jest chemicznie stabilny, biologicznie aktywny oraz wolny od RNA i białek. Ze względu na wielkość genomowego DNA (chromosom bakteryjny, genom faga) i wrażliwość praktycznie niemożliwa jest jego izolacja w formie niezmienionej. Część DNA ulega mechanicznemu uszkodzeniom, co nie powoduje zniszczenia drugorzędowej struktury DNA, ale zmniejsza długość cząsteczki (fragmentacja). Główne etapy izolacji DNA z komórek bakteryjnych: 1. Rozbicie ściany i błony komórkowej - użycie lizozymu. Lizozym, muramidaza (ang. lysozyme) to białko kationowe o ciężarze 14,4 kda, które ma właściwości enzymu hydrolitycznego rozkładającego peptydoglikan ściany komórkowej bakterii. Działanie lizozymu polega na rozrywaniu wiązań β-1,4-glikozydowych pomiędzy cząsteczkami kwasu N-acetylomuraminowego (NAM) i N-acetyloglukozaminą (NAG). Efektem jest zniszczenie błony zewnętrznej i uwolnienie zawartości komórki, w tym DNA. Komórki pozostają zawieszone w roztworze I zawierającym Tris (zapewnia właściwe ph), glukozę (warunkuje właściwe warunki osmotyczne), EDTA (chelatuje jony dwuwartościowe Mg 2+, Ca 2+, dzięki czemu destabilizowane są osłony komórkowe). 2. Usunięcie białek - zastosowanie detergentu SDS (Laurylosiarczan sodu; dodecylosiarczan(vi) sodu). Sól sodowa siarczanu dodecylu wiąże się z białkami i nadaje im silnie anionowy charakter. Zdenaturowane białka oddysocjowują od DNA. 3. Oddzielenie DNA od innych rozpuszczalnych komponentów komórkowych - odbiałczenie DNA z użyciem mieszanin fenol : chloroform : alkohol izoamylowy oraz chloroform : alkohol izoamylowy. W rezultacie powstają 3 fazy: górna wodna, dolna organiczna i trzecia pomiędzy nimi gdzie gromadzi się zdenaturowane białko. Fenol i chloroform powodują denaturację białek a alkohol izoamylowy redukuje pienienie i stabilizuje granicę fazową (gdzie koncentruje się białko). Do pobranej górnej fazy wodnej dodawany jest etanol, który zmniejsza polarność roztworu. To z kolei zmniejsza rozpuszczalność DNA. Wytrącony DNA należy odwirować. Izolacja bakteriofagowego DNA Pierwszym etapem izolacji DNA bakteriofagów jest oddzielenie cząstek fagowych od składników komórkowych i pożywki. Dzięki temu, że wiriony są uwalniane z komórek bakteryjnych do podłoża, cząstki fagowe można oddzielić od komórek bakteryjnych poprzez wirowanie. Wiriony pozostaną w supernatancie, a komórki i komórkowe debris zostaną zwirowane w odsadzie. Cząstki fagowe wytrąca się z supernatantu z użyciem PEG (glikol polietylenowy) i NaCl. PEG absorbuje wodę, co sprawia, że cząstki fagowe agregują i wytrącają się. Po zwirowaniu tworzą osad (pelet). Aby uwolnić DNA z kapsydu, należy zastosować detergent (SDS) lub proteinazę K. Całkowite odbiałczenie następuje po ekstrakcji mieszaninami: fenol:chloroform alkohol izoamylowy oraz chloroform: alkohol izoamylowy. 39
41 Część praktyczna Materiały Szczepy bakteryjne - Nocne hodowle szczepów Haemophilus influenzae Rd30 oznaczone jako A i B. Jeden z nich jest zlizogenizowany fagiem HP1. Podczas zajęć izolowany jest chromosomalny DNA z obu szczepów. Na jednym z kolejnych ćwiczeń (9) przeprowadzony będzie test z użyciem techniki PCR, aby zidentyfikować szczep H. influenzae Rd30 zlizogenizowany HP1. Do testu indukcji faga ci857 S7 : - E. coli LE392 ( - ) s F -, e14 - (McrA - ), hsdr514 (r k - m k + ), supf58, lacy1 or (lacizy)6, galk2, galt22, metb1, trpr55 Phage Display do namnożenia fagów: - E. coli ER2738 F proa + B + laci q Δ(lacZ)M15 zzf::tn10(tet R )/ fhua2 glnv Δ(lac-proAB) thi-1 Δ(hsdS-mcrB)5 Odczynniki Izolacja DNA faga HP1-5 M NaCl - 10 % SDS - izopropanol - mieszanina fenol:chloroform:alkohol izoamylowy (25:24:1), wysycona buforem TE - bufor TE: 10 mm TrisCl (ph 8,0), 1 mm EDTA (ph 8,0) - etanol 96 % i 70 % Izolacja chromosomalnego DNA (H. influenzae Rd30; szczepy A i B): - roztwór I: 50 mm glukoza, 25 mm TrisCl (ph 8,0), 10 mm EDTA (ph 8,0) - lizozym - stężenie początkowe 10 mg/ml - SDS 10 % - mieszanina fenol : chloroform : alkohol izoamylowy (25:24:1) - mieszanina chloroform : alkohol izoamylowy (24:1) - etanol 96 %, 70 % Test indukcji faga ci857 S7 - podłoże pólpłynne LB - podłoże LB stałe (2 szalki na parę) - bufor SM: 10 mm TrisCl (ph 7,4), 5 mm MgCl 2, 50 mm NaCl Phage Display - podłoże płynne LB - roztwór do wytrącania fagów: 20% PEG 8000, 2,5 M NaCl Wykonanie 1. Izolacja DNA faga HP1 - odwirować wytrącone cząstki fagowe w temp. 4 C, rpm, 10 min - zlać supernatant i ponownie wirować osad w temp. 4 C, rpm, 2 min - usunąć resztki płynu, otrzymany osad zawiesić w 750 l buforu TE i całość przenieść do eppendorfówki - po dokładnym zawieszeniu osadu (vortex), odwirować kłaczki heminy przez 2 min rpm - supernatant przenieść do nowej eppendorfówki - dodać 7,5 l SDS 10% - inkubować 3 min w temp. 68 C - dodać 15 l NaCl - schłodzić do temp. pokojowej 40
42 - POD WYCIĄGIEM: Ekstrahować kolejno: 2 mieszaniną fenol : chloroform : alkohol izoamylowy (równa objętość) 1 mieszaniną chloroform : alkohol izoamylowy (równa objętość) Fazy rozdzielać za każdym razem wirując 1 min rpm i zbierając fazę wodną, tj. górną, do nowej eppendorfówki - dodać równą objętość izopropanolu; - wytrącać DNA przez minimum 15 min w temp. -20 C; - odwirować w temp. 4 C, rpm, 15 min - osad przepłukać 1 ml 70 % etanolu, wysuszyć w temp. 42 C przez 10 min i zawiesić w 65 l buforu TE. 2. Izolacja chromosomalnego DNA (wg Sambrook i Russel, ed. 3) - odwirować (w temp. 4 C, rpm, 1 min) 1,5 ml hodowli nocnej H. influenzae; - osad zawiesić w 0,5 ml roztworu I; - dodać lizozymu do stężenia końcowego 1 mg/ml (50 l); - inkubować min w temp. 37 C; - dodać SDS do końcowego stężenia 0,5% (27,5 l); - inkubować 10 min w temp. 37 C; - POD WYCIĄGIEM: Ekstrahować kolejno: 2 mieszaniną fenol : chloroform : alkohol izoamylowy (równa objętość) 1 mieszaniną chloroform : alkohol izoamylowy (równa objętość) Po dodaniu mieszaniny fenol:chloroform:alkohol izoamylowy wytrząsać 5 min Fazy rozdzielać za każdym razem wirując 5 min rpm i zbierając fazę wodną, tj. górną, do nowej eppendorfówki, czynność powtórzyć również w przypadku chloroformu - dodać delikatnie 2 objętości 96% etanolu; - wirować 10 min rpm; - delikatnie zlać etanol; - ponownie wirować przez 3 min rpm; - wybrać resztki etanolu pipetą; - suszyć w temp. 42 C, 15 min w bloku grzejnym; - rozpuścić w 100 l H 2 O. 3. Indukcja faga ci857 S7 cd. - na 2 przygotowane szalki ze stałym podłożem LB wysiać powierzchniowo szczep E. coli LE392 (3ml półpłynnego LB o temp. 45 C + 0,2 ml nocnej hodowli bakteryjnej); - na powierzchnię zestalonego półpłynnego agaru nakroplić 5 l uzyskanego faga ci857 S7 ; - inkubować h w temp. 30 o C (jedna szalka) i 42 o C (druga szalka). 4. Phage Display - etap: Namnożenie specyficznych fagów w komórkach bakteryjnych E. coli - do dużych kolb (300 ml) dodać 20 ml podłoża LB, a następnie 0,2 ml nocnej hodowli E. coli ER2738 oraz 20 l wyizolowanych fagów; - kolby wytrząsać przez 2,5-3 h w 37 C; - hodowlę zwirować 10 min rpm; przenieść supernatant do nowej probówki i zwirować ponownie; - zebrać górne 80% supernatantu (16 ml), przenieść go do nowej probówki, dodać 1/6 objętości buforu do wytrącania PEG/NaCl (2,7 ml) i wytrącać przez noc w temp. 4 C. 41
43 ĆWICZENIE 7 Otrzymanie biblioteki fragmentów restrykcyjnych faga HP1 w wektorze plazmidowym pbluescript KS II (+) (etap: Uzyskanie zrekombinowanego plazmidu zawierającego fragmenty restrykcyjne faga HP1) Część teoretyczna Zjawisko restrykcji modyfikacji (patrz też ćwiczenie 3) W systemie restrykcji-modyfikacji (RM) wyróżnia się dwie aktywności enzymatyczne: endonukleolityczną (endonukleaza restrykcyjna, ang. restriction endonuclease, REaza) oraz modyfikującą (metylotransferaza DNA, metylaza DNA, ang. methyltransferase, MTaza), które rozpoznają w DNA tę samą, specyficzną sekwencję. MTaza modyfikuje określoną zasadę w sekwencji rozpoznawanej. Jeśli sekwencja docelowa nie jest zmetylowana, REaza przeprowadza cięcie endonukleolityczne DNA (Rys. 24). Zmodyfikowany genomowy DNA, w komórce posiadającej system RM, nie jest substratem dla REazy z tego systemu. Natomiast obcy DNA (np. infekującego faga) zostaje rozpoznany przez REazę i strawiony. Mechanizm obronny komórek bakteryjnych przed obcym DNA np. bakteriofagowym jest główną postulowaną funkcją systemów RM (Arber i Dussoix, 1962). Według innej hipotezy są one samolubnymi elementami genetycznymi, których utrzymanie w genomie jest warunkiem przetrwania - rodzaj systemu trucizna-odtrutka (Kobayashi, 2001). Sugeruje się też ich udział w utrzymywaniu tożsamości gatunkowej bakterii (Jeltsch, 2003) oraz, to, że przez indukowanie rearanżacji genomowych przyczyniają się do zróżnicowania genetycznego (Arber, 2000). 5 G AATTC 3 3 CTTAA G 5 REaza MTaza CH 3 CH 3 Rys. 24 Ta sama substratowa sekwencja w DNA dla endonukleazy restrykcyjnej i metylotransferazy DNA różne produkty reakcji przeprowadzanych przez te enzymy. Ze względu na różnorodność i mnogość przedstawicieli REaz oraz towarzyszących im MTaz, systemy RM zostały podzielone na cztery typy (Loenen i wsp., 2014 Roberts i wsp., 2003; patrz też ćwiczenie 3 Tabela 3). Do typu I zaliczono kompleksy białkowe złożone z trzech rodzajów podjednostek: rozpoznających specyficzną sekwencję (S), endonukleolitycznych (R) i modyfikacyjnych (M). Enzym do cięcia wymaga obecności ATP. Sekwencja rozpoznawana jest asymetryczna i składa się z dwóch krótkich odcinków zasad o określonej specyficzności, przedzielonych kilkoma niespecyficznymi parami nukleotydów (np. EcoKI AACN 6 GTGC). Systemy typu II stanowi najliczniejszą grupę, składającą się najczęściej z dwóch oddzielnych białek: monomerycznej MTazy i dimerycznej ENazy. Sekwencja docelowa jest krótka (4 do 8 par zasad) i często palindromiczna. Systemy typu III RM składając się z dwóch rodzajów podjednostek: 42
44 modyfikacyjnej (Mod) i endonukleolitycznej (Res). Miejsce rozpoznawane przez te enzymy, o długości 5-6 par zasad, jest niepalindromiczne (do cięcia wymagane są jego dwie kopie), a także ATP. Natomiast do typu IV zaliczono REazy, które trawią tylko zmetylowany DNA. Endonukleazy typu I i III charakteryzuje brak ścisłej kontroli nad położeniem miejsca cięcia względem rozpoznawanej sekwencji. Natomiast enzymy retrykcyjne typu II przecinają DNA w sekwencji rozpoznawanej albo blisko niej. Wynikiem trawienia określonej sekwencji DNA jest, w tym przypadku, powtarzalny zestaw fragmentów o przewidywanej długości. Właściwość ta spowodowała szerokie wykorzystanie enzymów restrykcyjnych typu II jako narzędzi w manipulacjach DNA. Endonukleazy restrykcyjne typu II Obecnie w bazie danych REBASE ( katalogującej enzymy restrykcyjne, metylotransferazy DNA i pokrewne białka, znajduje się ponad 3949 ENaz typu II rozpoznających 318 specyficznych sekwencji (dane z ). Komercyjnie dostępnych jest 236 różnych specyficzności REaz. Enzymy należące do tej grupy są najczęściej homodimerami lub tetramerami, wymagającymi tylko jonów Mg 2+ jako kofaktora. Nić DNA jest cięta wewnątrz krótkiej sekwencji rozpoznawanej lub w pewnej stałej odległości od niej. Mimo wspólnych cech, przedstawiciele tego typu są dość różnorodną grupą endonukleaz i dlatego wprowadzono podział na podtypy, uwzględniając przy tym budowę białek, rodzaj sekwencji docelowej, sposób cięcia DNA i inne cechy (Tabela 4). To samo białko może należeć do różnych podtypów, gdyż nie wykluczają się one wzajemnie (Roberts i wsp., 2003). Tabela 4. Podział enzymów resthrykcyjnych typu II na podtypy. Podtyp A Opis niepalindromiczna sekwencja rozpoznawana; często jeden gen koduje REazę a dwa geny kodują MTazy Przykładowe enzymy FokI, HphI B dwuniciowe cięcia po obu stronach sekwencji docelowej AloI, HaeIV C E obie domeny, endonukleolityczna i metylująca, w jednym polipeptydzie do cięcia potrzebne dwie kopie sekwencji docelowej, jedna cięta, druga działa jako efektor allosteryczny GsuI, HaeIV FokI, Sau3AI F interakcja z dwiema kopiami sekwencji rozpoznawanej i obie cięte BspMI, Cfr10I G obie domeny, endonukleolityczna i metylująca, w jednym polipeptydzie; AdoMet działa stymulująco na aktywność AloI, Eco57I H podobna struktura genów do REaz typu I AhdI, BcgI M sekwencja rozpoznawana cięta gdy zmetylowana DpnI P sekwencja rozpoznawana palindromiczna EcoRI, HaeIII S cięcie poza niepalindromiczną sekwencją rozpoznawaną FokI, Eco57MI T heterodimery Bpu10I, MlyI Nazewnictwo enzymów restrykcyjnych: stosowane są literowe skróty (np. HindIII, EcoRI). Pierwsza litera oznacza rodzaj bakterii, z której wyizolowano enzym, druga i trzecia - gatunek, a kolejna z nich pochodzi od szczepu lub typu bakterii. Następujące po niej cyfry rzymskie odpowiadają kolejnym enzymom wyizolowanym z określonego szczepu. HindIII - Haemophilus influenzae Rd; Mph1103I - Moraxella phenylpyruvica RFL
45 Izoschizomery enzymy restrykcyjne izolowane z różnych bakterii rozpoznające tę samą sekwencję w DNA i wprowadzające identyczny typ cięcia. Przykład: HaeIII, BsuRI, BshFI, PhoI, BsnI - GG CC CC GG Neoschizomery - restrykcyjne izolowane z różnych bakterii rozpoznające identyczną sekwencję w DNA, ale wprowadzające odmienny typ cięcia. Przykład: SmaI Cfr9I Acc65I KpnI CCC GGG C CCGGG G GTACC GGTAC C GGG CCC GGGCC C CCATG G C CATGG Jedna jednostka enzymu restrykcyjnego oznacza taką jego ilość, która katalizuje kompletną hydrolizę wiązań w markerowego DNA o masie 1 g w czasie 1 godziny i w temperaturze i warunkach właściwych dla danego enzymu. REazy pozostawiają w rozciętej cząsteczce DNA dwa rodzaje końców (Rys. 25): tępe końce - obie nici rozcięte są naprzeciwko siebie - nukleotydy znajdujące się na końcach są sparowane z komplementarnymi nukleotydami na przeciwnej nici; 5 -GG CC-3 5 -GG 3 5 -CC-3 3 -CC GG-5 3 -CC 5 3 -GG-5 lepkie końce - 3` lub 5` - jednoniciowe odcinki występujące na obu końcach przeciętej niesymetrycznie cząsteczki (Rys. 25). HindIII A AGCTT TTCGA A Mph1103I ATGCA T T ACGTA 5 -A 3 5 -AGCTT-3 5 -ATGCA T-3 3 -TTCGA-5 3 -A-5 3 -T ACGTA 5 Rys. 25 Enzym restrykcyjny HindIII generuje jednoniciowe wystające (lepkie) końce 5 a enzym Mph1103I jednoniciowe wystające (lepkie) końce 3. Rodzaj pozostawianych końców ma znaczenie przy manipulacjach przeprowadzanych przy klonowaniu: warunkuje połączenie końców zgodnych (kompatybilnych) wymusza konieczność przekształcenia lepkich końców w tępe, np. przez zastosowaniu polimerazy DNA Sekwencja palindromiczna sekwencja czytana w kierunku od 5 do 3 końca jest taka sama jak na nici komplementarnej. 5 - AAGCTT TTCGAA -5 44
46 Czynniki wpływające na aktywność enzymu restrykcyjnego 1. Bakterie, z których izolowane są enzymy restrykcyjne bytują w różnych, często ekstremalnych, środowiskach. Stąd wynikają różne preferencje warunków reakcji związane np. z temperaturą inkubacji. Inne elementy, które różnią bufory wykorzystywane do przeprowadzenia reakcji z danym enzymem to: siła jonowa (stężenie soli), główny kation (sód lub potas) i ph. Każdy enzym restrykcyjny wymaga obecności kofaktora Mg 2+! 2. Czasami w buforach do reakcji dostarczane są dodatkowe związki chemiczne, które wspomagają działanie niektórych enzymów restrykcyjnych - np. detergent (Triton-X-100 do redukcji napięcia powierzchniowego), BSA (ang. bovine serum albumin, albumina z grasicy cielęcej) zwiększa ogólne stężenie białka. 3. Obecność zanieczyszczeń pozostałych po preparatyce izolacji DNA (EDTA, fenol, etanol, proteinazy itd.) może inaktywować enzym restrykcyjny. 4. W wyniku zastosowania nieodpowiednich warunków reakcji może pojawić się niespecyficzna aktywność enzymu - tzw. star activity. Czynniki sprzyjające: zbyt długi czas reakcji (zmiana stężenia buforu w wyniku odparowywania wody z mieszaniny reakcyjnej) nieoptymalna temperatura reakcji, zbyt wysokie stężenie enzymu, nieoptymalne stężenie soli, nieoptymalne ph, obecność w mieszaninie reakcyjnej jonów Mn 2+, zamiast Mg 2+ (jony Mg 2+ są niezbędne dla aktywności endonukleaz); obecność rozpuszczalników organicznych, zbyt wysokie stężenie glicerolu (enzymy przechowuje się w temp -20 C w 50% glicerolu jednak, aby enzym działał prawidłowo końcowe stężenie glicerolu nie powinno przekraczać 5%). Endonukleazy restrykcyjne są wrażliwe na obecność zmetylowanej zasady w obrębie sekwencji DNA przez nie rozpoznawanej, co wyraża się nie rozpoznaniem jako substratu sekwencji docelowej. Zmetylowane zasady występujące w DNA to: N6-metyloadenina, N6mA, m 6 A), N4-metylocytozyna (N4mC, m 4 C) i 5-metylocytozyna (5mC, m 5 C) (Rys. 26) 45
47 AdoMet adenina cytozyna N6-metyloadenina N4-metylocytozyna 5mC -metylocytozyna Rys. 26. Zmetylowane zasady w DNA oraz kofaktor grup metylowych S-adenozylometionina (AdoMet). Niemal wszystkie szczepy Escherichia coli używane w laboratoriach zawierają, co najmniej dwie metylotransferazy DNA: - metylazę Dam (M.Dam), która dodaje grupę metylową do adeniny w sekwencji GATC (produkt Gm 6 ATC) - metylazę Dcm (M.Dcm), która modyfikuje wewnętrzną cytozynę w sekwencji CCWGG (W=A lub T) CC(A/T)GG do Cm 5 C(A/T)GG Praktyczną konsekwencją tego zjawiska jest fakt, że wiele endonukleaz nie będzie trawić DNA zmetylowanego przez w/w metylazy DNA. Poniżej kilka przykładów. Metylaza Dam E. coli, modyfikując w DNA adeninę w sekwencjach GATC, czyni je niewrażliwymi na trawienie enzymem MboI, natomiast tak zmetylowany DNA pozostanie wciąż substratem dla enzymu Sau3AI. 46
48 Metylaza Dcm E. coli, modyfikując w DNA drugą cytozynę w sekwencjach CCWGG, czyni je nie wrażliwymi na trawienie enzymem EcoRII, natomiast tak zmetylowany DNA pozostanie wciąż substratem dla enzymu MvaI. Miejsca rozpoznawane przez niektóre enzymy mogą zawierać (np. BclI TGATCA) lub pokrywać się częściowo z sekwencją GATC np TCGA (TaqI), ATCGA (ClaI). Gdy sekwencje te zostaną zmetylowane przez M.Dam, staną się niewrażliwe na trawienie w/w enzymami. Podobna sytuacja ma miejsce w przypadku nakładania się sekwencji docelowej danego enzymu z sekwencją CCWGG modyfikowaną przez M.Dcm np AGGCCT (StuI); TGGCCA (MscI). Gdy sekwencje te zostaną zmetylowane przez M.Dcm, staną się niewrażliwe na trawienie w/w enzymami. nntgatcann nnactagtnn nntcgatcnn nnagctagnn nnatcgatcnn nntagctagnn nnaggcctggnn nntccggaccnn nntggccaggnn nnaccggtccnn Jeśli (nieoczekiwanie) DNA nie został pocięty, przez użyty przez Ciebie enzym restrykcyjny, lub trawienie jest tylko częściowe, sprawdź, czy ten enzym niejest wrażliwy na metylację! Inaktywacja enzymów restrykcyjnych po reakcji enzymatycznej Nieodwracalna: inaktywacja termiczna (zwykle w temperaturze 65 C lub 80 C); denaturacja białek poprzez dodanie mieszaniny fenol:chloroform lub każdego z nich oddzielnie (po odbiałczeniu preparatu należy poddać go precypitacji); denaturacja białek poprzez dodanie HCl Odwracalna: dodanie EDTA do stężenia końcowego 12,5 mm (następuje wymiareczkowanie jonów Mg 2+ ) Połączenie fragmentów DNA w reakcja ligacji Ligacja jest przeprowadzana przez enzymy ligazy, które łączą dwa fragmenty DNA poprzez wytworzenie wiązania kowalencyjnego - fosfodiestrowego między końcem hydroksylowym 3' jednego nukleotydu a końcem 5' z grupą fosforanową drugiego nukleotydu przy wykorzystaniu energii z hydrolizy ATP (adenozynotrifosforanu). np ligaza faga T4 lub NAD + np ligaza E. coli. Dezaktywację ligazy przeprowadza się w temperaturze 65 o C przez 20 minut. 47
49 Analiza zrekombinowaneo DNA - elektroforeza Elektroforeza jest techniką, w której wymusza się ruch cząsteczek za pomocą pola elektrycznego. W przypadku DNA służy do identyfikacji, rozdziału i izolacji. Cząsteczki kwasów nukleinowych posiadają ładunek negatywny wynikający z ich szkieletu fosforanowego i migrują w kierunku anody (+). Agaroza - polisacharyd będący polimerem pochodnych galaktozy otrzymywany z glonów. Agaroza zawieszona w wodzie w temperaturze pokojowej tworzy koloid - żel. Służy do rozdziału nawet bardzo dużych cząsteczek, ale posiada relatywnie małą rozdzielczość. Typowe stężenia do rozdziału DNA to 0,5-2%. W standardowych warunkach poprzez użycie różnych stężeń agarozy mogą być rozdzielone fragmenty DNA między 50 a pz (Tabela 5). Tabela 5. Zależność pomiędzy stężeniem żelu agarozowego a zakresem długości rozdzielanego liniowego DNA. Stężenie agarozy (%) Zakres długości rozdzielanego liniowego DNA (kpz) 0, , ,7 0,8-10 0,9 0,5-7 1,2 0,4-6 1,5 0,2-3 2,0 0,1-2 Elektroforeza w żelach agarozowych prowadzona jest w aparatach ustawionych poziomo, a rozdział elektroforetyczny prowadzony jest w buforze: TBE 1 (90 mm Tris-base, 90 mm kwas borowy, 2 mm EDTA, ph 8) lub TAE 1 (40 mm Tris-base, 40 mm lodowy kwas octowy, 1 mm EDTA, ph 8. Akryloamid - polimer z grupy poliakrylanów otrzymywany przez polimeryzację akryloamidu. Ma niską zdolność rozdziału (fragmenty DNA do ok. 500 pz), ale bardzo wysoką rozdzielczość. Do uwidaczniania DNA po lub w trakcie elektroforezy stosowany jest rutynowo bromek etydyny (EtBr), który interkaluje pomiędzy sąsiednie pary dwuniciowego DNA (powinowactwo EtBr do jednoniciowego DNA jest znacznie słabsze). Obecność związku interkalującego w żelu agarozowym zmniejsza ruchliwość elektroforetyczną DNA o około 15%. EtBr to bardzo silny mutagen prawdopodobnie także karcynogen i teratogen! Dzięki właściwościom fluorescencyjnym EtBr możliwa jest wizualizacja DNA w świetle UV (300 nm). Innym odczynnikiem stosowanym do wizualizacji DNA jest np. SYBR Green, którego czułość jest 25 większa niż EtBr. Z reguły nakładając próbkę DNA na żel agarozowy mieszamy ją uprzednio z tzw. barwnikiem do nakładania na żel, lub krótko barwnikiem (ang. loading dye). Przyczyn jego użycia jest kilka: próbka DNA, która ma kolor jest wygodna w nakładaniu (łatwość wizualizacji), glicerol (lub sacharoza, ficoll 400) będący składnikiem tego barwnika zwiększa gęstość próbki i zapewnia, że znajdzie się ona na dnie dołka, negatywnie naładowany barwnik migruje przez żel w tym samym kierunku co DNA i stąd ułatwia śledzenie postępu rozdziału elektroforetycznego. 48
50 Najpopularniejsze barwniki: błękit bromofenolowy (ang. bromophenol blue), cyjanian ksylenu (ang. xylene cyanol), orange G. Różnią się kolorem, tempem migracji w danym żelu (stężenie agarozy, rodzaj buforu). Enzymy służące do modyfikacji DNA Obecnie stosuje się wiele różnorodnych, komercyjnie dostępnych enzymów służących do modyfikacji i rekombinacji DNA. Są to m.in.: enzymy restrykcyjne, alkaliczna fosfataza (AP, ang. alkaline phosphatase), kinaza polinukleotydowa, ligaza DNA, polimeraza DNA I, fragment Klenowa, nukleaza S1, odwrotna transkryptaza, terminalna transferaza, RNaza, DNaza, egzonukleazy (I, III), endonukleazy (IV, V), polimeraza DNA (Taq, Pfu), topoizomeraza. Alkaliczna fosfataza (EC ) jest hydrolitycznym enzymem odpowiedzialnym za usuwanie reszt 5 fosforanowych z DNA, RNA oraz nukleotydów. Wszystkie alkaliczne fosfatazy są metaloenzymami (Zn(II)). Proces usuwania grup fosforanowych nazywany jest defosforylacją i przebiega z wytworzeniem produktu pośredniego zawierającego ufosforylowaną serynę. Enzym ten wykazuje najwyższą aktywność w środowisku alkalicznym stąd jego nazwa. Możemy wyróżnić kilka rodzajów alkalicznej fosfatazy, pochodzących z różnych źródeł, które odróżnia między innymi możliwość inaktywacji: bakteryjna alkaliczna fosfataza (BAP bacterial alkaline phosphatase) wykazuje najwyższą aktywność i jest najbardziej trwała (najtrudniej ją inaktywować po zakończeniu reakcji defosforylacji). BAP jest wydzielana w postaci monomeru (M r = 47 kda) do przestrzeni peryplazmatycznej Escherichia coli, gdzie dimeryzuje i staje się katalitycznie aktywna. W obojętnym lub alkalicznym ph, dimer BAP zawiera do 6 jonów Zn 2+, z których dwa są niezbędne dla aktywności enzymu. Tylko jedno z dwóch miejsc katalitycznych dimeru jest aktywne przy niskim stężeniu sztucznego substratu, podczas gdy oba stają się aktywne przy wysokim stężeniu. alkaliczna fosfataza z jelita cielęcego (CIAP - calf intestinal alkaline phosphatase) najczęściej używana w laboratoriach biologii molekularnej. Wykazuje niewiele niższą aktywność w stosunku do BAP, a można ją efektywnie inaktywować termicznie lub poprzez trawienie proteolityczne. CIAP jest glikoproteiną zbudowaną z dwóch 514- aminokwasowych monomerów. Aktywność enzymu zależy od stężenia jonów Mg 2+ i Zn 2+. Zn 2+ wiąże się w centrum katalitycznym i jest wymagany do aktywności katalitycznej. Mg 2+ wiąże się do różnych miejsc enzymu i jest allosterycznym aktywatorem. alkaliczna fosfataza z krewetek (SAP - shrimp alkaline phosphatase) izolowana z krewetek Pandalus borealis, jest stosunkowo łatwa do inaktywacji termicznej. Przy manipulacjach DNA alkaliczna fosfataza używana jest głównie do: usuwania grupy 5 fosforanowej z plazmidów i wektorów bakteriofagowych, które uprzednio zostały strawione enzymami restrykcyjnymi (Rys. 28). Zapobiega to ponownej cyrkularyzacji wektora, a tym samym wydajność klonowania jest większa (Rys. 28). usuwania grupy 5 fosforanowej z fragmentów DNA poprzedzającego radioaktywne znakowanie 32 P (przygotowanie DNA do radioaktywnego znakowania w reakcji z wykorzystaniem polinukleotydowej kinazy faga T4); usuwania grupy 5 fosforanowej z RNA, rntps i dntps (Rys. 27); jako enzym reporterowy w nieradioaktywnych systemach do wykrywania i lokalizacji kwasów nukleinowych i białek. AP jest skoniugowana z ligandem, który specyficznie oddziałuje z cząsteczką docelową. 49
51 (A) (B) (C) alkaliczna fosfataza 5 p DNA lub 5 p RNA 5 OH DNA lub 5 OH RNA Rys. 27 Usuwanie grupy 5 fosforanowej DNA/RNA. (A) struktura nukleotydu, zaznaczono usuwaną grupę fosforanową; (B) i (C) reakcja defosforylacji. wektor nie poddany działaniu alkalicznej fosfatazy może ulegać ponownej cyrkularyzacji, co prowadzi do otrzymania dużej ilości pustych wektorów defosforylowany wektor nie może ulec cyrkularyzacji, bez insertu 3 OH 5 P 3 OH 3 OH 5 P 3 OH Rys. 28 Schemat klonowania insertu (powstałego po trawieniu enzymem restrykcyjnym) do defosforylowanego wektora. 50
52 Kinaza polinukleotydowa bakteriofaga T4 (T4 PNK) katalizuje przeniesienie -fosforanu z ATP (Rys. 29) na (patrz struktura nukleotydu po defosforylacji) koniec 5 OH jedno- lub dwu-niciowego DNA, RNA lub oligonukleotydów. Rys. 29 Struktura ATP (ang. adenosine triphosphate) i ADP (ang. adenosine diphosphate). Aktywność T4 PNK wykorzystywana jest głównie w dwóch typach reakcji: reakcji podstawienia (ang. forward reaction) i reakcji wymiany (ang. exchange reaction) (Rys. 30). W reakcji podstawienia -fosforan przenoszony jest z ATP na DNA/RNA. Docelowy nukleotyd nie posiada reszty fosforanowej na końcu 5 (została ona usunięta w reakcji defosforylacji lub w takiej postaci została zsyntetyzowana chemicznie). Reakcja ta jest odwracalna. W reakcji wymiany, nadmiar ADP powoduje, że T4 PNK najpierw przenosi 5 -fosforan z ufosforylowanego DNA, a następnie DNA jest ponownie fosforylowany przez przeniesienie -fosforanu z ATP (np. [ - 32 P]ATP). W reakcji katalizowanej przez T4 PNK stężenie ATP powinno wynosić 1 M (reakcja podstawienia) lub 2 M (reakcja wymiany). Ponadto enzym posiada aktywność 3 fosfatazy. Rys. 30 Aktywność enzymatyczna T4 PNK. Oczyszczenie kinazy polinukleotydowej bakteriofaga T4 z zainfekowanych komórek gospodarza jest niewydajne i trudne, dlatego też źródłem T4 PNK są komórki Escherichia coli z wklonowanym genem pset bakteriofaga T4. T4 PNK jest homotetramerem zbudowanym z podjednostek o masie 28,9 kda każda. T4 PNK jest głównie wykorzystywana do: radioaktywnego znakowania na końcu 5 kwasów nukleinowych (reakcja podstawienia lub wymiany), które następnie wykorzystywane są: (i) jako sonda hybrydyzacji; (ii) do mapowania transkryptu; (iii) jako markery do elektroforezy żelowej; (iv) jako startery w reakcji sekwencjonowania DNA; (v) jako startery do reakcji PCR lub w innych metodach wymagających terminalnie wyznakowanego DNA. 5'-fosforylacji oligonukleotydowych łączników i DNA/RNA przed ligacją. 51
53 Polimeraza DNA I (holoenzym) (PolI, polimeraza Kornberga) syntetyzowana przez E. coli zbudowana jest z pojedynczego łańcucha polipeptydowego o masie cząsteczkowej 109 kda, kodowanego przez gen pola. Enzym ten posiada następujące aktywności: (i) polimerazy DNA 5 3 ; (ii) egzonukleazy 3 5 ; (iii) egzonukleazy 5 3 oraz (iv) RNazy H. Aktywność RNazy H nie jest powszechnie wykorzystywana w biologii molekularnej. Poszczególne aktywności katalizowane są przez odrębne domeny enzymu (Tabela 6). Tabela 6. Domeny polimerazy DNA I kodowanej przez Escherichia coli. Domena Aktywność Karboksylowa, aminokwasy , ~46 kda Centralna, aminokwasy , ~22 kda* Aminowa, aminokwasy 1-325* *karboksylowa i centralna domena stanowią fragment Klenowa polimeryzacyjna 5 3 egzonukleolityczna 3 5 egzonukleolityczna 5 3 Fragment Klenowa jest dużym fragmentem polimerazy DNA I kodowanej przez E. coli. Posiada aktywność polimeryzacyjną 5 3 oraz aktywność egzonukleolityczną 3 5 (aktywność naprawcza). W porównaniu z PolI nie posiada aktywności egzonukleolitycznej 5 3. Do przeprowadzenia syntezy DNA w kierunku 5 3 wymagana jest obecność jednoniciowej matrycy oraz startera. Niezbędnym kofaktorem reakcji katalizowanych przez enzym są jony Mg 2+. Fragment Klenowa jest monomerem o wielkości 76 kda. Enzym ten można otrzymać poprzez proteolizę polimerazy Kornberga subtylizyną lub też poprzez sklonowanie odpowiedniego fragmentu genu pola w komórkach E. coli. Fragment Klenowa jest wykorzystywany w biologii molekularnej m.in. do: - wypełniania lepkich końców 5 (Rys. 31), powstałych po trawieniu enzymami restrykcyjnymi pozostawiającymi tego typu końce. Enzym ten katalizuje w sposób zależny od matrycy dodawanie trifosforanów nukleotydów do cofniętej reszty hydroksylowej 3. Synteza zachodzi w kierunku 5 3 do momentu, aż wystający lepki koniec zostanie całkowicie wypełniony. 5...GTCCA 3 OH fragment Klenowa 5...GTCCAAGCT 3 OH 3...CAGGTTCGA p CAGGTTCGA p 5 datp, dttp, dgtp, dctp Mg 2+ Rys. 31 Wypełnianie wystających lepkich końców 5 przez fragment Klenowa. - wytępiania lepkich końców 3, które powstały po trawieniu enzymami restrykcyjnymi zostawiającymi wystające końce 3. Jednak wytępianie lepkich końców 3 jest wydajniej katalizowane przez polimerazę DNA faga T4, która ma większą aktywność egzonukleolityczną. W niektórych przypadkach aktywność egzonukleolityczna 3 5 fragmentu Klenowa jest niepotrzebna lub szkodliwa. Należy wtedy zastosować fragment Klenowa exo -. Enzym ten zachowuje aktywność polimeryzacyjną, nie posiada natomiast 52
54 aktywności egzonukleolitycznej 3 5. Fragment Klenowa exo - uzyskuje się poprzez wprowadzenie odpowiedniej mutacji w genie kodującym fragment Klenowa. Polimeraza DNA bakteriofaga T4 jest białkiem o wielkości 114 kda kodowanym przez gen 43 colifaga T4. Otrzymywany jest z komórek E. coli niosących plazmid zawierających gen 43. Enzym ten posiada aktywności zbliżone do aktywności fragmentu Klenowa, tj. aktywność polimeryzacyjną 5 3 oraz aktywność egzonukleolityczną 3 5. Nie posiada aktywności egzonukleolitycznej 5 3. Polimeraza DNA bakteriofaga T4 jest wykorzystywana w biologii molekularnej m.in do: - wytępiania lepkich końców 3, które powstały po trawieniu enzymami restrykcyjnymi zostawiającymi wystające końce 3 (Rys. 32). Aktywność egzonukleolityczna w stosunku do dwuniciowego DNA blokowana jest przez aktywność polimeryzacyjną 5 3. polimeraza DNA 5... p C p G p C p A p T p C p T 3 bakteriofaga T4... p C p G p C OH G p C p G p 5... G p C p G p 5 Mg 2+ + pa + 5 pc + 5 pt Rys. 32 Wytępianie lepkich końców 3 przez polimerazę DNA bakteriofaga T4. - wypełniania lepkich końców 5. Aktywność polimeryzacyjna 5 3 wymaga obecności matrycy, startera, jonów Mg 2+ oraz dntp. Aktywność egzonukleolityczną 3 5 polimerazy DNA bakteriofaga T4 jest ok. 200 razy silniejsza niż fragmentu Klenowa. Powoduje to, że enzym ten jest wydajniejszy w wytępianiu lepkich końców 3. 53
55 Część praktyczna Materiały Rekombinacja DNA Podstawowymi materiałami są wyizolowane przez studentów DNA faga HP1 i plazmidu pbluescript KSII(+) oraz - enzymy restrykcyjne (endonukleazy restrykcyjne) BamHI i BglII - bufor do trawienia enzymami restrykcyjnymi (10 stężony): 100 mm TrisCl (ph 8,5), 100 mm MgCl 2, 1000 mm KCl, 0.1mg/ml BSA - bufor TBE (10 stężony): 1M Tris, 0,9 M kwas borowy, 0,01 M EDTA - barwnik (obciążnik) do nakładania na żel (6 stężony): 15 % glicerol, 0,25 % błękit bromofenolowy, 0,25% xylen cyanol FF - bromek etydyny 10 mg/ml - ATP 10 mm. Phage Display - PEG/NaCl: 20% PEG-8000, 2,5 M NaCl - TBS: 50 mm Tris-HCl ph 7,5, 150 mm NaCl - 3 szalki LB z IPTG, X-gal i tetracykliną - nocna hodowla E. coli ER2738 Wykonanie 1. Nastawienie trawień DNA wektora pbluescript KSII(+) enzymem restrykcyjnymi BamHI: G GATCC CCTAG G - 3 l DNA (ok. 0,5 g) - 2 l buforu do trawienia - 14, 7 l H 2 O - 0, 3 l enzymu restrykcyjnego BamHI - DNA faga HP1 enzymem restrykcyjnym: BglII: A GATCT TCTAG A - 6 l DNA (ok. 1,5 g) - 2 l buforu do trawienia - 11, 7 l H 2 O - 0, 3 l enzymu restrykcyjnego BglII inkubacja w temp. 37 o C, 1,5 h Uwaga! Dokładne ilości składników mieszaniny (trawienie 1 i 2) podane zostaną przez osobę prowadzącą ćwiczenia. 2. Elektroforeza DNA w żelu agarozowym - przygotowanie żelu agarozowego (0,7 % w buforze 1 TBE) - przygotowanie próbek trawionego DNA do naniesienia na żel (pobrać 10 l mieszaniny reakcyjnej i po zmieszaniu z 2 l barwnika, nanieść na żel) - rozwijać elektroforezę przez 1 h, a następnie barwić żel w bromku etydyny - wizualizacja żelu w świetle UV (dł. fali 300 nm) 54
56 3. Przygotowanie reakcji ligacji - pobrać 6 l pociętego DNA plazmidu - 6 l pociętego DNA faga HP1 - dodać 8 l mieszaniny ligacyjnej: bufor do trawienia 0,8 l, 10 mm ATP 2 l, H 2 O 4,2 l ligaza - 1 l, Reakcję inkubować w temp. pokojowej h Fot. Elektroforeza produktów trawienia DNA faga HP1 enzymem BglII. NC BglII M nie cięty DNA 10504bp 9591bp 6941bp 5208bp 1000bp 8000bp 6000bp 5000bp 4000bp 3500bp 3000bp 2500bp 2000bp Tor NC: nie cięty DNA faga HP1 Tor BglII: DNA faga HP1 strawiony enzymem BglII M: marker wielkości 1500bp 1200bp 1000bp 900bp 800bp 700bp 600bp 500bp 55
57 5. Odczytanie wyników (oglądanie szalek) z ćwiczenia 6 pkt 4. (Indukcja faga ci857 S7 ) 6. Phage Display - na początku ćwiczeń odświeżyć nocną hodowlę szczepu E. coli ER wytrącone cząstki fagowe zwirować 15 minut, rpm w 4 C, zlać supernatant - ponownie zwirować 5 minut, rpm w 4 C i dokładnie usunąć supernatant - osad faga zawiesić w 1 ml TBS, przenieść do probówki eppendorfa i wirować 1 min 5000 rpm - przenieść supernatant do nowej probówki eppendorfa, dodać 1/6 objetości PEG/NaCl i wytrącać fagi w lodzie przez 30 minut - wirować 10 minut rpm w temp 4 C i usunąć supernatant - osad faga zawiesić w 200 l TBS - wirować 1 min 5000 rpm i przenieść supernatant do nowej probówki - wykonać szalki dwuwarstwowe z hodowlą E. coli ER2738, na podłożu LB z IPTG, X-gal i tetracykliną z użyciem rozcieńceń faga 10-4, 10-6 i 10-8 Wybrana literatura: 1. Arber W, Dussoix D. Host specificity of DNA produced by Escherichia coli. I. Host controlled modification of bacteriophage lambda. J Mol Biol : Arber W. Genetic variation: molecular mechanisms and impact on microbial evolution. FEMS Microbiol Rev : Bertani G, Weigle JJ. Host controlled variation in bacterial viruses. J Bacteriol : Ishikawa, K., Fukuda, E., Kobayashi, I. Conflicts targeting epigenetic systems and their resolution by cell death: novel concepts for methyl-specific and other restriction systems DNA Res. 17: Jeltsch A. Maintenance of species identity and controlling speciation of bacteria: a new function for restriction/modification systems? Gene : Kobayashi I.Behavior of restriction-modification systems as selfish mobile elements and their impact on genome evolution. Nucleic Acids Res : Loenen WA, Dryden DT, Raleigh EA, Wilson GG, Murray NE. Highlights of the DNA cutters: a short history of the restriction enzymes. Nucleic Acids Res : Luria SE, Human ML. A nonhereditary, host-induced variation of bacterial viruses. J Bacteriol : Orlowski, J., Bujnicki, J.M. Structural and evolutionary classification of Type II restriction enzymes based on theoretical and experimental analyses Nucleic Acids Res. 36: Roberts RJ et. al. A nomenclature for restriction enzymes, DNA methyltransferases, homing endonucleases and their genes. Nucleic Acids Res : Sambrook,J. and Russell,D. (2001) Molecular Cloning: A Laboratory Manual, 3rd ed Cold Spring Harbor Laboratory, Cold Spring Harbor, New York 56
58 ĆWICZENIE 8 Otrzymanie biblioteki fragmentów restrykcyjnych faga HP1 w wektorze plazmidowym pbluescript KS II (+) (etap: Wprowadzenie zrekombinowanego DNA do komórek Escherichia coli, transformacja). Transfekcja Haemophilus influenzae DNA faga HP1 Część teoretyczna Transformacja: rys historyczny W 1928 r. F. Griffith jako pierwszy zbadał i opisał zjawisko transformacji komórek bakteryjnych. Opracowując szczepionkę przeciw zapaleniu płuc, Griffith badał dwa szczepy Streptococcus pneumoniae: zjadliwy szczep S, które wytwarzał polisacharydową otoczkę oraz niewirulentny bezotoczkowy szczep R. Wirulentne bakterie szczepu S, po zabiciu przez podgrzanie i wstrzyknięciu myszom, nie powodowały objawów chorobowych. Jednak jeśli martwe bakterie szczepu S wstrzyknięto razem z żywymi niezjadliwymi bakteriami szczepu R, myszy umierały. Istniał więc czynnik transformujący szczep niepatogenny w patogenny. Dalsze badania wykazały, że czynnikiem transformującym jest DNA (Avery, MacLeod, McCarthy, 1944). Transformacją nazywamy proces pobierania obcego, zewnątrzkomórkowego, wolnego DNA przez komórkę biorcy. Jedynie komórki kompetentne są w stanie pobrać DNA ze środowiska. Wiele bakterii jest naturalnie kompetentnych. Większość osiąga ten stan spontanicznie w określonych warunkach (np. Bacillus, Streptococcus, Azotobacter czy Haemophilus), inne są konstytutywnie kompetentne (np. Neisseria gonorrhoeae). Zazwyczaj pobierany jest każdy DNA, jednak niektóre bakterie, jak np. N. gonorrhoeae lub H. influenzae, pobierają tylko DNA homologiczny, tzn. taki, który posiada specyficzne sekwencje (tzw. uptake sequence). Istnieją także komórki, które w warunkach naturalnych nigdy nie osiągają stanu kompetencji, np. komórki E. coli. Transformacja chemiczna Cząsteczki DNA są zbyt duże, aby przenikać przez błony komórkowe. W warunkach laboratoryjnych, transformację E. coli przeprowadza się metodą chemiczną. Mieszanina DNA dodawana jest do zawiesiny komórek uprzednio traktowanych dwuwartościowymi kationami: Ca 2+, Mg 2+, Mn 2+ lub Ba 2+ w niskiej temperaturze. Całość poddaje się szokowi termicznemu. Mechanizm transformacji chemicznej jest nieznany. Podejrzewa się, że w komórkach będących w logarytmicznej fazie wzrostu, błony komórkowe (zewnętrzna i wewnętrzna) tworzą tzw. obszary adhezji (ang. adhesion zone), dzięki czemu powstają kanały. W niskiej temperaturze, cząsteczki LPS, obecne na powierzchni błony zewnętrznej, dezorganizują się, odsłaniając te kanały. Dwuwartościowe jony neutralizują ujemny ładunek DNA, który może się przykleić do kanałów. Krótki szok cieplny pozwala na wejście DNA do cytoplazmy komórki (Rys. 33). Rys 33. Przypuszcalny mechanizm transformacji E.coli metodą chemiczną. W przypadku E. coli wydajność transformacji zależy od formy DNA: najwyższa jest dla formy CCC (covalently closed circular), niższa dla OC (open circular) a najniższa dla formy liniowej. 57
59 Transformując komórki mieszaniną DNA w warunkach saturacyjnych (nadmiarze DNA) do komórek mogą wniknąć dwa plazmidy, jednakże jeżeli należą one do tej samej grupy niezgodności to nie mogą razem koegzystować w jednej komórce, juz przy pierwszym podziale jeden z plazmidów będzie usunięty. Naturalną transformację można podzielić na 4 etapy: - przyłączanie DNA do komórki bakteryjnej - fragmentacja DNA - degradacja i transport DNA - rekombinacja z chromosomem gospodarza U bakterii gram dodatnich, dwuniciowy DNA łączy się z powierzchnią komórki w specyficznych miejscach. W kompetentnej komórce B. subtilis jest około 50 takich miejsc (tzw. DNA binding site), do których przyłącza się DNA, u S. pneumoniae-ok U B. subtilis i S. pneumoniae receptorem DNA jest białko ComEA (Rys. 34). Wydaje się, że jest ono także zaangażowane w transport DNA do komórki. Istnieje wiele innych receptorów wiążących DNA u B. subtilis: np. białka kodowane przez operony comg i comc. Nie wydaje się, aby sekwencja pobieranego DNA miała tu wpływ na adsorpcję na powierzchni komórkowej. Rys. 34. Transformacja bakterii gramdodatnich. Dwuniciowy DNA łączy się z receptorem ComEA. Następuje cięcie DNA i degradacja poprzez nukleazę N jednej nici (EndA u S. pneumoniae). Białka ComG tworzą transporter umożliwiający przejście DNA przez ścianę komórkową. Białka ComEC tworzą pory w błonie komórkowej. Białko ComFA translokuje DNA do cytoplazmy. Natychmiast po połączeniu się z receptorem zaczyna się fragmentacja DNA (wykazano u B. subtilis i S. pneumoniae). U tej ostatniej DNA jest najpierw nikowany a później następuje cięcie obu nici. Przez błonę cytoplazmatyczną przechodzi tylko jednoniciowy DNA (druga nić jest degradowana). U S. pneumoniae za nikowanie, cięcie i degradację DNA odpowiedzialna jest błonowa endonukleaza EndA (obecna także w E.coli). U Bacillus subtilis transport DNA do wewnątrz komórki trwa ok 1,5 min w 37 C. Prędkość transferu to ok. 180 nukleotydów/sek. (S. pneumonie ok nt/sek) W tym samym czasie w podłożu pojawiają się nukleotydy oraz nukleozydy, potwierdzając fakt, że degradacja DNA ma miejsce na zewnątrz komórki. Za transport jednoniciowego DNA do komórek, odpowiedzialne są białka ComEA i ComEC, kodowane przez operon come. U B. subtilis w transporcie uczestniczy także błonowe białko ComFA, które translokuje DNA. 58
60 Ryc. 35. Transformacja komórek Gram ujemnych U bakterii gramujemnych DNA musi przejść przez błonę zewnętrzną, ścianę komórkową oraz błonę cytoplazmatyczną (Rys. 35). Transformacja bakterii H. influenzae oraz N. gonorrhoeae wymaga obecności w pobieranym DNA specyficznych sekwencji, tzw. uptake sequence. Te dwie bakterie pobierają więc tylko homologiczny DNA. Natomiast Acinetobacter calcoaceticus zdolny jest do pobrania każdego DNA. U H. influenzae wykazano obecność tylko 4-8 miejsc wiązania zewnątrzkomórkowego DNA/komórkę kompetentną. Receptorami DNA są białka zaangażowane w wykształcanie pili (Pil Q), u N. gonorrhoeae: dodatkowo potrzebny jest produkt genu pilc. Zarówno u H. influenzae jak i u N. gonorrhoeae DNA jest transportowany z szybkością nt/sek do wnętrza komórki. U bakterii gramujemnych także ma miejsce degradacja DNA, jednakże tylko kolistych lub bardzo dużych cząsteczek liniowych (Rys. 36). Podejrzewa się, że dwuniciowy DNA jest transportowany do peryplazmy i wtedy dopiero ma miejsce degradacja jednej nici DNA. W translokacji DNA do cytoplazmy, podobnie jak u B. subtilis, uczestniczy białko ComFA (H. influenzae i prawdopodobnie Neisseriaceae). Ryc. 36. Transformacja bakterii gramujemnych. Dwuniciowy DNA wchodzi do peryplazmy przez błonę zewnętrzną (OM) dzięki porom zbudowanym z białek PilQ. U N. gonorrhoeae w tym etapie uczestniczy także białko PilC. Prawdopodobnie białka PilE tworzą strukturę umożliwiającą przejście przez ścianę komórkową (z pomocą białek ComL i Tpc). Białka receptorowe oraz nukleaza (N) degradująca jedną nić DNA nie są znane. Kanał pozwalający na przejście jednoniciowego DNA przez błonę wewnętrzną (IM) zbudowany jest z białek ComA (homologów ComEC). Za translokację odpowiedzialne jest prawdopodobnie białko ComFA (T). Transformasomy H. influenzae H. influenzae wchodzi w stan kompetencji fakultatywnie, np. w niekorzystnych warunkach wzrostu. Stan kompetencji można u H. influenzae wywołać, np. zwiększając stężenie camp w podłożu (obniżone stężenie węglowodanów), obniżając stężenie puryn w komórce (hodowla na podłożu minimalnym MIV) lub poprzez metodę tlenowo-beztlenową. Podwyższone stężenie camp powoduje aktywację białka CRP (camp responsive protein), a obniżone stężenie puryn aktywuje białko TfoX (homolog białka Sxy u E. coli). Oba te białka 59
61 rozpoznając elementy CRE (camp responsive element) kontrolują ekspresję genów kompetencji. Znanych jest wiele białek kodowanych przez geny kompetencji, są to: receptory wiążące DNA na powierzchni komórki egzonukleazy, odpowiedzialne za usuwanie jednej z nici DNA białka transportujące DNA do wnętrza komórki (np. permeaza oligonukleotydowa) białka stabilizujące jednoniciowy DNA składniki transformasomu Transformasomy są to wyspecjalizowane wypustki/uwypuklenia błony, licznie obecne na kompetentnych komórkach H. influenzae (Rys. 37). Transformasomy zbudowane są z dwóch warstw lipidowych, w które wbudowane są białka błony zewnętrznej oraz LPS. W miejscu, gdzie transformasomy stykają się z błoną zewnętrzną, odkryto obecność małych porów/kanałów. DNA wchodzi do transformasomu i tam jest chroniony (np. przed enzymami restrykcyjnymi). Rys. 37. Transformasomy u Haemophilus influenzae. genomowym DNA z bakteriofaga HP1 (8 miejsc AAGTGCGGTCA). DNA z transformasomu (II) jednym końcem wnika do cytoplazmy (III). Nić o polarności 5-3 jest degradowana i ostatecznie do cytoplazmy dociera jedynie nić 3-5 Pobrany DNA nie utrzymuje się w komórce bakteryjnej, lecz musi zostać zrekombinowany z chromosomem gospodarza (IV). Tylko dwuniciowy liniowy DNA może opuścić transformasom: kolisty DNA zostaje uwięziony w transformasomie, ponieważ nie ma wolnych końców (IIA). H. influenzae pobiera wyłącznie homologiczny DNA (sekwencja uptake sequence 5 - AAGTGCGGTCA-3, ok. 600 kopii w chromosomie H. influenzae). Na ćwiczeniach przeprowadzimy transfekcję wolnym Czemu służy zdolność do pobierania DNA ze środowiska: - Możliwość naprawy uszkodzeń we własnym DNA: u B. subtilis system naprawczy DNA jest częścią regulonu obejmującego kompetencję. Czynniki uszkadzające DNA nie zawsze indukują kompetencję, jednakże wywołanie kompetencji u B. subtilis potraktowanego UV zwiększa liczbę bakterii, które przeżyją naświetlanie. - Nabywanie nowych cech fenotypowych: aby pobrany DNA doprowadził do zmian w genomie biorcy, DNA musi zostać ustabilizowany (rekombinacja z homologiczną sekwencją w genomie biorcy/ ustabilizowanie plazmidu). W ten sposób pobranie obcego DNA może zwiększyć przewagę selekcyjną transformanta. - Niektórzy badacze sugerują, ze pobieranie wolnego DNA służy jako swoiste sondowanie terenu w poszukiwaniu nowych cech. Np. u N. meningitidis znaleziono gen socd, który posiada dwie uptake sequences z H. influenzae i prawdopodobnie został nabyty w drodze transformacji. - Pozakomórkowy DNA może służyć jako składnik pokarmowy; 60
62 Wybrana literatura: 1. Chen I, Christie PJ, Dubnau D.The ins and outs of DNA transfer in bacteria. Science. 2005; 2;310(5753): Claverys JP, Martin B, Polard P. The genetic transformation machinery: composition, localization, and mechanism. FEMS Microbiol Rev. 2009; 33(3): Goodgal SH. DNA uptake in Haemophilus transformation. Annu Rev Genet. 1982;16: Goodgal SH, Mitchell M. Uptake of heterologous DNA by Haemophilus influenzae. J Bacteriol. 1984;157(3):
63 Cześć praktyczna Materiały Szczepy bakteryjne E. coli TopF ( - ) s, r - m -, mcra (mrr-hsdrms-mcrbc) 80 LacZ M15 lacx74 deor reca1 arad139 (ara-leu) 7697 galu galk lambda^-rpsl enda1 nupg (F proab laciqz)m15 Tn10 (Tet r ) E. coli ER2738 F proa+b+ laciq Δ(lacZ)M15 zzf::tn10(tetr)/ fhua2 glnv Δ(lac-proAB) thi-1 Δ(hsdS-mcrB)5 H. influenzae Rd30 (HP1 - ) DNA DNA bakteriofaga HP1 (ćw. 6) DNA fagmidu pbluescript (ćw. 6) DNA fagmidu pbluescript - uliniowione enzymami restrykcyjnymi (ćw. 7) mieszanina ligacyjna (ćw. 7) Odczynniki Podłoża LB stałe z dodatkiem: tetracykliny (10 g/ml) ampicyliny (100 g/ml) IPTG (izopropylo- -D-tiogalaktopiranozyd; 0,5 mm) X-gal (5-bromo-4-chloro-3-indolyl- -D-galaktozyd; 40 g/ml) LB płynne BHI stałe (ang. Brain Heart Infusion wyciąg mózgowo-sercowy) uzupełnione heminą (stężenie końcowe w pożywce 10 g/ml) i NAD (10 g/ml) BHI płynne bez uzupełnień BHI półpłynne uzupełnione heminą i NAD (j.w.) Wykonanie 1. Transformacja E. coli - podzielić komórki kompetentne na 2 porcje po 100 l - dodać do 100 l komórek kompetentnych: a) 5 l mieszaniny ligacyjnej lub b) 0,5 l DNA plazmidowego lub c) 0,5 l uliniowionego DNA plazmidowego - inkubować min w łaźni lodowej - przenieść na 1,5 min do temp. 42 C - umieścić na 3 minuty w łaźni lodowej - dodać 0,9 ml LB płynnego (nie uzupełnionego antybiotykami!!!) - inkubować transformowane komórki przez 60 min w temp. 37 C - nanieść 100 l na szalkę z LB + tetracyklina + ampicylina + X-gal + IPTG i rozprowadzić głaszczką równomiernie po całej powierzchni. - odwirować resztę bakterii (przez 2 min, 6000 rpm), zawiesić w 100 l LB płynnego i wysiać na szalkę (j.w.) - inkubować h w temp. 37 C 62
64 2. Przygotowanie bakterii kompetentnych Haemophilus influenzae - zaszczepić 20 ml podłoża BHI uzupełnionego NAD i heminą całonocną hodowlą szczepu H. influenzae Rd30 - inkubować z wytrząsaniem, aż hodowla osiągnie OD 600 = 0,5 (2 h) - przenieść całość hodowli do szklanej probówki, tak aby wypełnić jak największą objętość - inkubować w temp. 37 C przez 1 h (bez wytrząsania: warunki beztlenowe) - przelać hodowlę do kolbki i inkubować z wytrząsaniem 1 h w temp. 37 (warunki tlenowe) - po tych dwóch inkubacjach komórki H. influenzae nabywają stan kompetencji 3. Transfekcja komórek H. influenzae Rd30 DNA faga HP1 - zrobić mieszaninę transfekcyjną: do 1,3 ml podłoża BHI bez heminy i NAD dodać: 0,1 ml kompetentnych bakterii H. influenzae 10 l DNA faga HP1 - inkubować w temp. 37 C przez 30 min - do 3 ml półpłynnego BHI uzupełnionego NAD i heminą dodać 0,2 ml nocnej hodowli szczepu wskaźnikowego H. influenzae Rd30 i 0,3 ml mieszaniny transfekcyjnej - wymieszać i wylać na płytkę ze stałym podłożem BHI uzupełnionym NAD i heminą - inkubować h w temp. 37 C 4. Phage Display - nocną hodowlę E.coli ER2738 rozcieńczyć podłożem LB w stosunku 1:100, odświeżyć i rozdzielić do 5 probówek Eppendorfa po 1 ml - do każdej z probówek przenieść jałowo materiał z pojedynczej łysinki i hodować 2,5-3 h w temp 37 C z wytrząsaniem - hodowle zwirować 1 minutę przy 5000 rpm - supernatanty przenieść do nowych probówek, ponownie zwirować - przenieść górne 80% supernatantów do nowych probówek i przechowywać w 4 C 63
65 ĆWICZENIE 9 Otrzymanie biblioteki fragmentów restrykcyjnych faga HP1 w wektorze plazmidowym pbluescript KS II (+) (izolacja i analiza restrykcyjna DNA rekombinantów) Część teoretyczna Izolacja plamidowego DNA metodą lizy alkalicznej (Brinboim i Doly, 1979) jest najbardziej popularną metodą stosowaną do komórek Escherichia coli. Ekspozycja zawiesiny bakterii na silny jonowy detergent w wysokim ph otwiera ścianę komórkową, denaturuje chromosomalny DNA i białka oraz uwalnia plazmidowy DNA do supernatantu. Chociaż alkaliczne środowisko kompletnie rozrywa wiązania wodorowe, nici koliście zamkniętego plazmidowego DNA nie są zdolne do rozdzielenia się ze względu na topologiczne super zwinięcie (forma CCC ang. covalently closed circular). Gdy ph zostaje zneutralizowane (dodanie octanu potasu) następuje renaturacja plazmidowego DNA. Natomiast chromosomalny DNA i białka tworzą nierozpuszczalny kompleks wraz z dodecylosiarczanem potasu KDS. Dodany octanu potasu wypiera jony sodu z SDS, zastępując je jonami potasu. Wykorzystane jest tu zjawisko niskiej rozpuszczalności KDS oraz wysokiego powinowactwa SDS do białek. Kluczowym etapem w izolacji plazmidowego DNA jest efektywna liza komórek bakteryjnych (ilość DNA i jego jakość zależy od jakości lizatu komórkowego). Główne etapy lizy alkalicznej: 1. Zawieszenie komórek bakteryjnych. Ważne jest aby zawiesina komórek była homogenna, bowiem wówczas odczynniki mogą dotrzeć do wszystkich komórek. Roztwór I: glukoza (zapewnia odpowiednie warunki osmotyczne, zapobiega zbyt wczesnemu pęknięciu komórek), EDTA (chelatuje jony dwuwartościowe Mg 2+, Ca 2+, dzięki czemu destabilizowane są osłony komórkowe), Tris-Cl (zapewnia właściwe ph) 2. Liza. SDS rozpuszcza fosfolipidowe i białkowe komponenty błony komórkowej, prowadząc do lizy i uwolnienia zawartości komórek. NaOH denturuje DNA i białka. Zabronione jest gwałtowne wstrząsanie lizatu, gdyż może to doprowadzić do fragmentacji chromosomu. Pozostawione w supernatancie wolne fragmenty chromosomu będą oczyszczać się wspólnie z plazmidowym DNA. 3. Neutralizacja. ph lizatu zostaje zobojętnione poprzez dodanie kwaśnego octanu sodu. Wysokie stężenie soli powoduje wytrącenie dodecylosiarczanu potasu (KDS) wraz ze zdenaturowanymi białkami, chromosomalnym DNA i resztami osłon komórkowych w postaci nierozpuszczalnego kompleksu. Wytrącaniu sprzyja użycie zimnego buforu neutralizującego i inkubacja w lodzie. 4. Oczyszczenie lizatu wirowanie lub filtracja. 64
66 Część praktyczna Materiały: Nocne hodowle zaszczepione z białych kolonii uzyskanych w ćwiczeniu 8, tj. szczep Escherichia coli XL-1 Blue (niosący zrekombinowany plazmid pbluescript KS) Odczynniki do izolacja plazmidowego DNA (metodą lizy alkalicznej): - roztwór I: 50 mm glukoza, 25 mm TrisCl (ph 8,0), 10 mm EDTA (ph 8,0) - roztwór II: 0,2 M NaOH, 1 % SDS - roztwór III: 3 M octan sodowy, 2 M kwas octowy - mieszanina fenol:chloroform:alkohol izoamylowy (25:24:1) - etanol 96 %, 70 % - bufor TE: 10 mm TrisCl (ph 8,0), 1 mm EDTA (ph 8,0) Wykonanie: 1. Izolacja izolacja plazmidowego DNA - przenieść 1,5 ml hodowli do eppendorfówki i odwirować (7 000 rpm), w temp. 4 C, 1,5 min - zlać supernatant, powtórzyć czynność z pkt. 1 - zawiesić bakterie w 100 l roztworu I - dodać 200 l roztworu II, wymieszać zawartość przez kilkakrotne DELIKATNE odwracanie eppendorfówki - dodać 150 l roztworu III, wymieszać zawartość przez kilkakrotne odwracanie eppendorfówki - inkubować w lodzie przez 5 min - odwirować w temp. 4 C, maksymalne obroty ( rpm), 8 min - przenieść supernatant do nowej eppendorfówki - ekstrahować równą objętością mieszaniny fenol:chloroform:alkohol izoamylowy - odwirować 2 min rpm - przenieść fazę wodną (górną) do nowej eppendorfówki - dodać 1 ml (2 objętości) etanolu (96 %) i wytrącać DNA przez 5 min w temp. pokojowej - odwirować w temp. 4 C, rpm, 5 min - zlać ostrożnie supernatant - przepłukać osad 0,5 ml etanolu (70%) - odwirować przez 1 min - zlać supernatant; usunąć dokładnie resztki etanolu przy użyciu pipety automatycznej - suszyć 15 min w temp. 42 C - osad zawiesić w 50 l wody lub TE z RNazą 20 g/ml 2. Nastawienie trawień enzymami restrykcyjnymi Trawienie 1 enzymem BsuRI - zmieszać: 10 l wyizolowanego DNA plazmidowego 2 l buforu R + 7, 7 l H 2 O 0,3 l enzymu BsuRI: GG CC CC GG 65
67 Trawienie 2 enzymem BglI - zmieszać: 10 l wyizolowanego DNA plazmidowego 2 l buforu O + 7, 7 l H 2 O 0,3 l enzymu BglI: GCCNNNN NGGC CGGN NNNNCCG Uwaga! Dokładne ilości składników mieszaniny (trawienie 1 i 2) podane zostaną przez osobę prowadzącą ćwiczenia. 66
68 Diagnostyka wirusologiczna: wykrywanie materiału genetycznego wirusów z zastosowaniem łańcuchowej reakcji polimerazy - PCR Co to jest PCR? PCR (ang. Polymerase Chain Reaction), czyli łańcuchowa reakcja polimerazy jest jedną z podstawowych technik biologii molekularnej. Wykorzystywana jest do powielania konkretnych fragmentów DNA. Jej podstawowym atutem jest szybkość oraz prostota. Pomysłodawca PCR Kary Mullis (Nagroda Nobla w dziedzinie Chemii w 1993) wykorzystał podstawowe zasady replikacji DNA przeprowadzanej przez enzym - polimerazę DNA. Enzym ten jest w stanie dołączyć nukleotyd tylko do wolnej grupy -OH na końcu 3 nici, dlatego kierunek polimeryzacji jest określony: 5 -> 3. Ponadto, polimeraza do rozpoczęcia polimeryzacji potrzebuje tzw. startera, nie jest bowiem w stanie przyłączyć się do pojedynczej nici DNA. Startery wyznaczają fragment DNA, który chcemy powielić. Do przeprowadzenia reakcji PRC używa się aktualnie termostabilnych polimeraz DNA, a reakcję PCR przeprowadza się w specjalnych aparatach, zwanych termocyklerami, które umożliwiają szybkie i precyzyjne zmiany temperatury. Reakcja PCR rozpoczyna się od denaturacji DNA. W wysokiej temperaturze (ok. 95 C) dochodzi do zerwania wiązań wodorowych łączących obie nici. Następnie temperatura zostaje obniżona do około C w tej temperaturze startery odnajdują swoje sekwencje komplementarne na niciach matrycowych i łączą się z nimi. Po około sekundach temperatura zostaje podwyższona do 72 C - temperatury optymalnej dla polimerazy DNA. Polimeraza odnajduje hydroksylowe końce 3 starterów i na podstawie nici matrycowej syntetyzuje nową nić DNA. Czas polimeryzacji (zwany elongacją) jest skorelowany z długością amplifikowanego fragmentu. Tak kończy się pierwszy cykl reakcji PCR. Aby namnożyć wybrany fragment w ilości nadającej się do dalszych analiz zwykle powtarza się taki cykl około razy. Pozwala to uzyskać ogromną liczbę kopii danego fragmentu DNA. Z każdym kolejnym cyklem liczba cząsteczek DNA ulega podwojeniu. Po 30 cyklach liczba cząsteczek dwuniciowego DNA wyniesie (ok ). Reakcję łańcuchowej polimeryzacji można więc postrzegać jako molekularną kopiarkę. Dzięki reakcji PCR następuje selektywna amplifikacja (powielenie) określonego odcinka DNA. Ten odcinek może reprezentować niewielką część dużej i złożonej mieszaniny różnych DNA np. DNA chromosomalny, DNA totalny (chromosom plus plamidy). Dzięki PCR można w ok. 2 h zamplifikować śladowe ilości DNA (np. pojedynczą kopię genu) do ilości użytecznych (tj. widocznych w żelu), Matryca do PCR nie musi być oczyszczona wystarczy zagotowana kolonia bakteryjna. Produkt PCR może zostać następnie strawiony enzymami restrykcyjnymi, zsekwencjonowany lub sklonowany. Klasyczny cykl syntezy DNA w reakcji PCR (3 etapy, Rys. 38): - denaturacja (94-95 C) sek - przyłączenie (hybrydyzacja) starterów (55-65 C) sek - wydłużanie (72 C), czas zależny od wielkości produktu Cykl ten jest powtarzany razy. Zwiększenie liczby cykli >35 ma niewielki efekt pozytywny, gdyż plateau jest osiągane gdy: wyczerpują się odczynniki, produkty łączą się same ze sobą oraz polimeraza ulega inaktywacji. Efektem może być: akumulacja niechcianych produktów (niespecyficznych)! 67
69 Rys. 38. Profil termiczny reakcji PCR. Co znajduje się w reakcji PCR? Matryca DNA lub RNA, startery (ang. primers), bufor, substraty dntp, jony Mg 2+, termostabilna polimeraza DNA Optymalny rezultat reakcji PCR to obecność tylko właściwego produktu, przy braku produktów niespecyficznych! Jak duży może być produkt PCR? Typowy produkt ma długość pz Większe produkty (nawet > 25 kb), są możliwe do uzyskania. Reakcja wymaga jednak zmodyfikowanego buforu, koktailu polimeraz i długiego czasu syntezy. Ograniczeniem jest integralność matrycy. Duże DNA jest mechanicznie fragmentowane do odcinków < 50kb. Projektowanie starterów - przypuszczalnie najbardziej krytyczny parametr efektywnej reakcji PCR Krytyczne zmienne w wyborze startera: - długość - temperatura topnienia (Tm) - specyficzność - sekwencja komplementarna - zawartość par GC - sekwencja na końcu 3' Długość startera - specyficzność i temperatura hybrydyzacji startera w dużej mierze zależą od jego długości. Przyjmuje się, że startery o długości pz są wysoce specyficzne. Długość startera jest związana z wydajnością hybrydyzacji: ogólnie - im dłuższy jest starter tym hybrydyzacja jest mniej wydajna. Ale starter nie powinien być zbyt krótki, gdyż wówczas spada specyficzność! Specyficzność (unikalność) startera Startery muszą być unikalne! Specyficzność startera zależy częściowo od jego długości. Sekwencja 24 nukleotydowa jest bardziej unikalna niż ta o długości 15 pz Naturalnie startery muszą flankować odcinek, który ma być powielony! Temperatura topnienia startera zależy od jego długości i sekwencji. Im więcej par GC tym wyższa wartość Tm. Zbyt niska temperatura topnienia faworyzuje niewłaściwe łączenie nukleotydów w pary. 68
70 Zasady projektowania starterów: - Długość przynajmniej 15 pz - Zawartość par GC ok. 50%; równomierna dystrybucja wszystkich 4 nukleotydów, brak odcinków wielokrotnych powtórzeń pojedynczego nukleotydu, szczególnie poli-g i poli-c. 5 GGTTGACTTGGGGGGGGATG 3 - Temperatura hybrydyzacji z matrycą C. Z reguły im wyższa temperatura hybrydyzacji tym lepiej (wzrasta specyficzność startera). Starter kierunkowy i odwrotny powinny mieć mniej więcej tę samą temperaturę hybrydyzacji. - Brak odcinków wewnętrznej homologii powyżej 3 par zasad (odcinków typu szpilka do włosów lub pętla łodyga ). - Brak wzajemnej homologii międzycząsteczkowej - pomiędzy starterami tworzącymi parę. Gdy homologia dotyczy odcinka środkowego utrudnia to hybrydyzację startera. Homologia końców 3' skutkuje wytworzeniem produktu primer-dimer. - Sekwencja na końcu 3 kluczowa w zabezpieczaniu przed błędnym parowaniem startera. Najlepiej aby ostatnim nukleotydem była G lub C: 5 GGTTGACTTGGCGAATG 3 - Temperatura topnienia (Tm). Temperatura hybrydyzacji przynajmniej 50 C Zasada ustawienia temperatury etapu hybrydyzacji startera: 5 niżej niż temperatura topnienia Wyliczenie Tm zależy od użytego algorytmu. Prosta formuła kalkulacji (formuła Wallacea) może być zastosowana tylko do krótkich starterów o długości do 25 nukleotydów: Tm = 4x (#C+#G) + 2x (#A+#T) C Gdy starter jest dłuższy (>25 nukleotydy) Tm oblicza się używając specjalistycznego programu komputerowego. 69
71 Część praktyczna Materiały: - PCR bufor (10 ): 100 mm Tris (ph 8,8), 15 mm MgCl 2, 500 mm KCl - startery oligonukleotydowe: (10 M) Nazwa Sekwencja 5 3 % GC długość Tm LgtF_F GGCGGATCCGTGGGCGTAAAAAGTCGTAAAAGTA 40%, 50% 25bp, 34bp 54,66 C LgtF_R CGGGCGAATTCCCGAGCCAAGTTAGAAGCCATTGTTA 46%, 51% 26bp, 37bp 58,68 C ORF14ATG GCCGAATTCATGTTATACCCAATTTGTATCG 32%, 39% 22bp, 31bp 49,59 C ORF14TAA GGCAAGCTTTTATGCAGAAAGTAATCTATTTGTTG 27%, 34% 26bp, 35bp 50,60 C - polimeraza DNA Taq (5 U/ l) - matrycowy DNA chromosomalny DNA Haemophilus influenzae (z lub bez profaga) 100 ng 1 g - nukleotydy - mix (datp, dttp, dctp, dgtp) (10 mm) Wykonanie: 1. Przygotowanie mieszaniny reakcyjnej - zmieszać: 5 l buforu do reakcji PCR (końcowo 1 ) 5 MgCl 2 (końcowo 1,5 mm) 5 l dntps (końcowo 200 nm) 5 l 4 starterów (po 100 pmoli każdego) 5 l matrycowego DNA 0,5 l polimerazy DNA Taq (5U/ l) 2. Warunki reakcji PCR prowadzonej w termocyklerze a - wstępna denaturacja 94 C przez 2 min b - denaturacja 94 C przez 30 sek. c - przyłączanie starterów (annealing) 45 C przez 30 sek d - elongacja 72 C przez 1,5 min - etapy b, c, d powtórzyć w 2 cyklach e - denaturacja 94 C przez 30 sek. f - przyłączanie starterów (annealing) 55 C przez 30 sek g - elongacja 72 C przez 1,5 min - etapy e, f, g powtórzyć w 28 cyklach - w cyklu 30 elongację wydłużyć do 7 min w temp. 72 C - próbki schłodzić do temp. 4 C - Fot. Elektroforeza produktów rekacji PCR. M-marker wielkości Tor 1 produkt PCR 1400 bp z matrycy DNA chromosomalne H.influenzae Tor 2 - produkty PCR 1400 bp i 450bp z matrycy DNA chromosomalnego H. influenzae zlizogenizowanego fagiem HP1 Tor 3 produkt PCR 450 bp z DNA faga HP1 M
72 ĆWICZENIE 10 Wykorzystanie elementów bakteriofagowych w biologii molekularnej. Phage Display Część teoretyczna W biologii molekularnej i biotechnologii znalazły zastosowanie całe wirusy lub poszczególne elementy lub enzymy wirusowe. Wykorzystywane enzymy bakteriofagowe to m.in: Ligaza DNA faga T4 - enzym ten katalizuje tworzenie wiązań fosfodiestrowych pomiędzy grupą fosforanową 5 a grupą hydroksylową 3 w DNA lub RNA; łączy lepkie lub tępe końce; naprawia jednoniciowe pęknięcia w dwuniciowym DNA; wymaga ATP jako kofaktora reakcji; jest kodowany przez gen 30 bakteriofaga T4 E. coli; jej aktywność jest hamowana przez wysokie stężenie NaCl lub KCl (> 200 mm); Ligaza RNA faga T4 - kodowana jest przez gen 63 bakteriofaga T4 E. coli, katalizuje ATPzależne tworzenie wiązań fosfodiestrowych pomiędzy grupą fosforanową 5 a grupą hydroksylową 3 jednoniciowego RNA i DNA; Kinaza polinukleotydowa faga T4 (patrz ćwiczenie 7); Polimeraza DNA faga phi29 - białko to jest kodowane przez gen 2 bakteriofaga phi29 Bacillus subtilis; enzym ten jest wysoceprocesywną polimerazą DNA, pozwalającą na amplifikację DNA w stałej temperaturze; Polimeraza DNA faga T4 (patrz ćwiczenie 7); Egzonukleaza faga - enzym ten kodowany jest przez gen exo bakteriofaga λ E. coli; posiada aktywność egzonukleolityczną w kierunku 5 3 ; Ponadto zastosowanie znalazły promotory i represory bakteriofagowe. Promotory bakteriofaga T5 i T7 wykorzystuje się m.in. w wektorach służących do uzyskiwania nadprodukcji białek, których geny sklonowane są na tych wektorach. Promotor faga T5 znajduje się m.in. w wektorze pqe-30, pqe-40, promotor faga T7 znalazł zastosowanie w wektorach z serii pet, np. pet28a, pet30a. Promotor bakteriofaga T5 czytany jest zarówno przez polimerazę RNA faga T5 jak i polimerazę RNA komórek gospodarza. Promotor bakteriofaga T7 czytany jest jedynie przez polimerazę RNA faga T7. Gen tej polimerazy znajduje się pomiędzy nt genomu faga i koduje 883 aminokwasowe białko. Polimeraza RNA faga T7 jest, wysoce specyficznym zależnym od promotora T7, enzymem katalizującym syntezę RNA w kierunku 5' 3. Białko to wymaga DNA jako matrycy i jonów Mg 2+ jako kofaktora reakcji, a jego aktywność stymulowana jest przez BSA (ang. bovine serum albumine) i spermidynę. Dodatkowa charakterystyka polimerazy RNA faga T7 zawarta jest na Rys. 39. Rys. 39. Charakterystyka polimerazy RNA bakteriofaga T7. 71
CZĘŚĆ IV METODY BIOTECHNOLOGICZNE W OCHRONIE ŚRODOWISKA ZASTOSOWANIE WIRUSÓW BAKTERYJNYCH DO OCENY JAKOŚCI I CZYSTOŚCI WODY
 CZĘŚĆ IV METODY BIOTECHNOLOGICZNE W OCHRONIE ŚRODOWISKA ĆWICZENIE: ZASTOSOWANIE WIRUSÓW BAKTERYJNYCH DO OCENY JAKOŚCI I CZYSTOŚCI WODY PROWADZĄCY: dr Agnieszka Kwiatek CEL Celem ćwiczeń jest zapoznanie
CZĘŚĆ IV METODY BIOTECHNOLOGICZNE W OCHRONIE ŚRODOWISKA ĆWICZENIE: ZASTOSOWANIE WIRUSÓW BAKTERYJNYCH DO OCENY JAKOŚCI I CZYSTOŚCI WODY PROWADZĄCY: dr Agnieszka Kwiatek CEL Celem ćwiczeń jest zapoznanie
Zawartość. Wstęp 1. Historia wirusologii. 2. Klasyfikacja wirusów
 Zawartość 139585 Wstęp 1. Historia wirusologii 2. Klasyfikacja wirusów 3. Struktura cząstek wirusowych 3.1. Metody określania struktury cząstek wirusowych 3.2. Budowa cząstek wirusowych o strukturze helikalnej
Zawartość 139585 Wstęp 1. Historia wirusologii 2. Klasyfikacja wirusów 3. Struktura cząstek wirusowych 3.1. Metody określania struktury cząstek wirusowych 3.2. Budowa cząstek wirusowych o strukturze helikalnej
KLONOWANIE DNA REKOMBINACJA DNA WEKTORY
 KLONOWANIE DNA Klonowanie DNA jest techniką powielania fragmentów DNA DNA można powielać w komórkach (replikacja in vivo) W probówce (PCR) Do przeniesienia fragmentu DNA do komórek gospodarza potrzebny
KLONOWANIE DNA Klonowanie DNA jest techniką powielania fragmentów DNA DNA można powielać w komórkach (replikacja in vivo) W probówce (PCR) Do przeniesienia fragmentu DNA do komórek gospodarza potrzebny
Podstawy mikrobiologii. Wirusy bezkomórkowe formy materii oŝywionej
 Podstawy mikrobiologii Wykład 3 Wirusy bezkomórkowe formy materii oŝywionej Budowa wirusów Wirusy nie mają budowy komórkowej, zatem pod względem biologicznym nie są organizmami Ŝywymi! Są to twory nukleinowo
Podstawy mikrobiologii Wykład 3 Wirusy bezkomórkowe formy materii oŝywionej Budowa wirusów Wirusy nie mają budowy komórkowej, zatem pod względem biologicznym nie są organizmami Ŝywymi! Są to twory nukleinowo
Inżynieria Genetyczna ćw. 3
 Materiały do ćwiczeń z przedmiotu Genetyka z inżynierią genetyczną D - blok Inżynieria Genetyczna ćw. 3 Instytut Genetyki i Biotechnologii, Wydział Biologii, Uniwersytet Warszawski, rok akad. 2018/2019
Materiały do ćwiczeń z przedmiotu Genetyka z inżynierią genetyczną D - blok Inżynieria Genetyczna ćw. 3 Instytut Genetyki i Biotechnologii, Wydział Biologii, Uniwersytet Warszawski, rok akad. 2018/2019
Dr. habil. Anna Salek International Bio-Consulting 1 Germany
 1 2 3 Drożdże są najprostszymi Eukariontami 4 Eucaryota Procaryota 5 6 Informacja genetyczna dla każdej komórki drożdży jest identyczna A zatem każda komórka koduje w DNA wszystkie swoje substancje 7 Przy
1 2 3 Drożdże są najprostszymi Eukariontami 4 Eucaryota Procaryota 5 6 Informacja genetyczna dla każdej komórki drożdży jest identyczna A zatem każda komórka koduje w DNA wszystkie swoje substancje 7 Przy
Diagnostyka wirusologiczna w praktyce klinicznej
 Diagnostyka wirusologiczna w praktyce klinicznej Ponad 60% zakażeń w praktyce klinicznej jest wywołana przez wirusy. Rodzaj i jakość materiału diagnostycznego (transport!) oraz interpretacja wyników badań
Diagnostyka wirusologiczna w praktyce klinicznej Ponad 60% zakażeń w praktyce klinicznej jest wywołana przez wirusy. Rodzaj i jakość materiału diagnostycznego (transport!) oraz interpretacja wyników badań
Klonowanie molekularne Kurs doskonalący. Zakład Geriatrii i Gerontologii CMKP
 Klonowanie molekularne Kurs doskonalący Zakład Geriatrii i Gerontologii CMKP Etapy klonowania molekularnego 1. Wybór wektora i organizmu gospodarza Po co klonuję (do namnożenia DNA [czy ma być metylowane
Klonowanie molekularne Kurs doskonalący Zakład Geriatrii i Gerontologii CMKP Etapy klonowania molekularnego 1. Wybór wektora i organizmu gospodarza Po co klonuję (do namnożenia DNA [czy ma być metylowane
DNA musi współdziałać z białkami!
 DNA musi współdziałać z białkami! Specyficzność oddziaływań między DNA a białkami wiążącymi DNA zależy od: zmian konformacyjnych wzdłuż cząsteczki DNA zróżnicowania struktury DNA wynikającego z sekwencji
DNA musi współdziałać z białkami! Specyficzność oddziaływań między DNA a białkami wiążącymi DNA zależy od: zmian konformacyjnych wzdłuż cząsteczki DNA zróżnicowania struktury DNA wynikającego z sekwencji
Metody odczytu kolejności nukleotydów - sekwencjonowania DNA
 Metody odczytu kolejności nukleotydów - sekwencjonowania DNA 1. Metoda chemicznej degradacji DNA (metoda Maxama i Gilberta 1977) 2. Metoda terminacji syntezy łańcucha DNA - klasyczna metoda Sangera (Sanger
Metody odczytu kolejności nukleotydów - sekwencjonowania DNA 1. Metoda chemicznej degradacji DNA (metoda Maxama i Gilberta 1977) 2. Metoda terminacji syntezy łańcucha DNA - klasyczna metoda Sangera (Sanger
ĆWICZENIE 5 MECHANIZMY PROMUJĄCE WZROST ROŚLIN
 ĆWICZENIE 5 MECHANIZMY PROMUJĄCE WZROST ROŚLIN CZĘŚĆ TEORETYCZNA Mechanizmy promujące wzrost rośli (PGP) Metody badań PGP CZĘŚĆ PRAKTYCZNA 1. Mechanizmy promujące wzrost roślin. Odczyt. a) Wytwarzanie
ĆWICZENIE 5 MECHANIZMY PROMUJĄCE WZROST ROŚLIN CZĘŚĆ TEORETYCZNA Mechanizmy promujące wzrost rośli (PGP) Metody badań PGP CZĘŚĆ PRAKTYCZNA 1. Mechanizmy promujące wzrost roślin. Odczyt. a) Wytwarzanie
E.coli Transformer Kit
 E.coli Transformer Kit zestaw do przygotowywania i transformacji komórek kompetentnych Escherichia coli. Metoda chemiczna. wersja 1117 6 x 40 transformacji Nr kat. 4020-240 Zestaw zawiera komplet odczynników
E.coli Transformer Kit zestaw do przygotowywania i transformacji komórek kompetentnych Escherichia coli. Metoda chemiczna. wersja 1117 6 x 40 transformacji Nr kat. 4020-240 Zestaw zawiera komplet odczynników
Prokariota i Eukariota
 Prokariota i Eukariota W komórkach organizmów żywych ilość DNA jest zazwyczaj stała i charakterystyczna dla danego gatunku. ILOŚĆ DNA PRZYPADAJĄCA NA APARAT GENETYCZNY WZRASTA WRAZ Z BARDZIEJ FILOGENETYCZNIE
Prokariota i Eukariota W komórkach organizmów żywych ilość DNA jest zazwyczaj stała i charakterystyczna dla danego gatunku. ILOŚĆ DNA PRZYPADAJĄCA NA APARAT GENETYCZNY WZRASTA WRAZ Z BARDZIEJ FILOGENETYCZNIE
Ćwiczenie 5 Klonowanie DNA w wektorach plazmidowych
 Ćwiczenie 5 Klonowanie DNA w wektorach plazmidowych Celem ćwiczenia jest sklonowanie fragmentu DNA (powielonego techniką PCR i wyizolowanego z żelu agarozowego na poprzednich zajęciach) do wektora plazmidowego
Ćwiczenie 5 Klonowanie DNA w wektorach plazmidowych Celem ćwiczenia jest sklonowanie fragmentu DNA (powielonego techniką PCR i wyizolowanego z żelu agarozowego na poprzednich zajęciach) do wektora plazmidowego
ĆWICZENIA Z MECHANIZMÓW DZIAŁANIA WYBRANYCH GRUP LEKÓW
 UNIWERSYTET MARII CURIE-SKŁODOWSKIEJ WYDZIAŁ BIOLOGII I BIOTECHNOLOGII ZAKŁAD BIOLOGII MOLEKULARNEJ ĆWICZENIA Z MECHANIZMÓW DZIAŁANIA WYBRANYCH GRUP LEKÓW dla studentów I roku II 0 biotechnologii medycznej
UNIWERSYTET MARII CURIE-SKŁODOWSKIEJ WYDZIAŁ BIOLOGII I BIOTECHNOLOGII ZAKŁAD BIOLOGII MOLEKULARNEJ ĆWICZENIA Z MECHANIZMÓW DZIAŁANIA WYBRANYCH GRUP LEKÓW dla studentów I roku II 0 biotechnologii medycznej
POLIMERAZY DNA- PROCARYOTA
 Enzymy DNA-zależne, katalizujące syntezę DNA wykazują aktywność polimerazy zawsze w kierunku 5 3 wykazują aktywność polimerazy zawsze wobec jednoniciowej cząsteczki DNA do utworzenia kompleksu z ssdna
Enzymy DNA-zależne, katalizujące syntezę DNA wykazują aktywność polimerazy zawsze w kierunku 5 3 wykazują aktywność polimerazy zawsze wobec jednoniciowej cząsteczki DNA do utworzenia kompleksu z ssdna
Nowoczesne systemy ekspresji genów
 Nowoczesne systemy ekspresji genów Ekspresja genów w organizmach żywych GEN - pojęcia podstawowe promotor sekwencja kodująca RNA terminator gen Gen - odcinek DNA zawierający zakodowaną informację wystarczającą
Nowoczesne systemy ekspresji genów Ekspresja genów w organizmach żywych GEN - pojęcia podstawowe promotor sekwencja kodująca RNA terminator gen Gen - odcinek DNA zawierający zakodowaną informację wystarczającą
Inżynieria genetyczna- 6 ECTS. Inżynieria genetyczna. Podstawowe pojęcia Część II Klonowanie ekspresyjne Od genu do białka
 Inżynieria genetyczna- 6 ECTS Część I Badanie ekspresji genów Podstawy klonowania i różnicowania transformantów Kolokwium (14pkt) Część II Klonowanie ekspresyjne Od genu do białka Kolokwium (26pkt) EGZAMIN
Inżynieria genetyczna- 6 ECTS Część I Badanie ekspresji genów Podstawy klonowania i różnicowania transformantów Kolokwium (14pkt) Część II Klonowanie ekspresyjne Od genu do białka Kolokwium (26pkt) EGZAMIN
POLIMERAZY DNA- PROCARYOTA
 Enzymy DNA-zależne, katalizujące syntezę DNA wykazują aktywność polimerazy zawsze w kierunku 5 3 wykazują aktywność polimerazy zawsze wobec jednoniciowej cząsteczki DNA do utworzenia kompleksu z ssdna
Enzymy DNA-zależne, katalizujące syntezę DNA wykazują aktywność polimerazy zawsze w kierunku 5 3 wykazują aktywność polimerazy zawsze wobec jednoniciowej cząsteczki DNA do utworzenia kompleksu z ssdna
E.coli Transformer Zestaw do przygotowywania i transformacji komórek kompetentnych Escherichia coli
 E.coli Transformer Zestaw do przygotowywania i transformacji komórek kompetentnych Escherichia coli Wersja 0211 6x40 transformacji Nr kat. 4020-240 Zestaw zawiera komplet odczynników do przygotowania sześciu
E.coli Transformer Zestaw do przygotowywania i transformacji komórek kompetentnych Escherichia coli Wersja 0211 6x40 transformacji Nr kat. 4020-240 Zestaw zawiera komplet odczynników do przygotowania sześciu
2015-11-18. DNA i RNA ENZYMY MODYFIKUJĄCE KOŃCE CZĄSTECZEK. DNA i RNA. DNA i RNA
 Fosfataza alkaliczna CIP Calf Intestine Phosphatase- pochodzenie: jelito cielęce BAP Bacterial Alcaline Phosphatase- pochodzenie: E. coli SAP Shrimp Alcaline Phosphatase- pochodzenie: krewetki Pandalus
Fosfataza alkaliczna CIP Calf Intestine Phosphatase- pochodzenie: jelito cielęce BAP Bacterial Alcaline Phosphatase- pochodzenie: E. coli SAP Shrimp Alcaline Phosphatase- pochodzenie: krewetki Pandalus
Polimeraza Taq (1U/ l) 1-2 U 1 polimeraza Taq jako ostatni składniki mieszaniny końcowa objętość
 Ćwiczenie 6 Technika PCR Celem ćwiczenia jest zastosowanie techniki PCR do amplifikacji fragmentu DNA z bakterii R. leguminosarum bv. trifolii TA1 (RtTA1). Studenci przygotowują reakcję PCR wykorzystując
Ćwiczenie 6 Technika PCR Celem ćwiczenia jest zastosowanie techniki PCR do amplifikacji fragmentu DNA z bakterii R. leguminosarum bv. trifolii TA1 (RtTA1). Studenci przygotowują reakcję PCR wykorzystując
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA
 Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa IZOLACJA DNA Z HODOWLI KOMÓRKOWEJ.
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa IZOLACJA DNA Z HODOWLI KOMÓRKOWEJ.
Saccharomyces Transformer Kit zestaw do przygotowywania i transformacji komórek kompetentnych Saccharomyces cerevisiae. Metoda chemiczna.
 Saccharomyces Transformer Kit zestaw do przygotowywania i transformacji komórek kompetentnych Saccharomyces cerevisiae. Metoda chemiczna. wersja 0916 6 x 20 transformacji Nr kat. 4010-120 Zestaw zawiera
Saccharomyces Transformer Kit zestaw do przygotowywania i transformacji komórek kompetentnych Saccharomyces cerevisiae. Metoda chemiczna. wersja 0916 6 x 20 transformacji Nr kat. 4010-120 Zestaw zawiera
ENZYMY RESTRYKCYJNE ENZYMY RESTRYKCYJNE CZYM RÓŻNIĄ SIĘ POSZCZEGÓLNE ENZYMY? nazewnictwo: EcoRV
 ENZYMY RESTRYKCYJNE Enzymy z klasy endonukleaz (hydrolaz), wykazujące powinowactwo do specyficznych fragmentów dwuniciowych cząsteczek DNA i hydrolizujące wiązania fosfodiestrowe w obu niciach Naturalnie
ENZYMY RESTRYKCYJNE Enzymy z klasy endonukleaz (hydrolaz), wykazujące powinowactwo do specyficznych fragmentów dwuniciowych cząsteczek DNA i hydrolizujące wiązania fosfodiestrowe w obu niciach Naturalnie
Biologia molekularna wirusów. Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego.
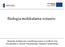 Biologia molekularna wirusów Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Co to jest wirus? Cząsteczka złożona z kwasu nukleinowego (DNA
Biologia molekularna wirusów Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Co to jest wirus? Cząsteczka złożona z kwasu nukleinowego (DNA
wykład dla studentów II roku biotechnologii Andrzej Wierzbicki
 Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ 1. Gen to odcinek DNA odpowiedzialny
Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ 1. Gen to odcinek DNA odpowiedzialny
października 2013: Elementarz biologii molekularnej. Wykład nr 2 BIOINFORMATYKA rok II
 10 października 2013: Elementarz biologii molekularnej www.bioalgorithms.info Wykład nr 2 BIOINFORMATYKA rok II Komórka: strukturalna i funkcjonalne jednostka organizmu żywego Jądro komórkowe: chroniona
10 października 2013: Elementarz biologii molekularnej www.bioalgorithms.info Wykład nr 2 BIOINFORMATYKA rok II Komórka: strukturalna i funkcjonalne jednostka organizmu żywego Jądro komórkowe: chroniona
KLONOWANIE DNA REKOMBINACJA DNA WEKTORY
 KLONOWANIE DNA Klonowanie DNA jest techniką powielania fragmentów DNA DNA można powielać w komórkach (replikacja in vivo) W probówce (PCR) Do przeniesienia fragmentu DNA do komórek gospodarza potrzebny
KLONOWANIE DNA Klonowanie DNA jest techniką powielania fragmentów DNA DNA można powielać w komórkach (replikacja in vivo) W probówce (PCR) Do przeniesienia fragmentu DNA do komórek gospodarza potrzebny
Definicje. Białka rekombinowane (ang. recombinant proteins, r-proteins) Ukierunkowana mutageneza (ang. site-directed/site-specific mutagenesis)
 Definicje Białka rekombinowane (ang. recombinant proteins, r-proteins) Białka, które powstały w żywych organizmach (lub liniach komórkowych) w wyniku ekspresji rekombinowanego DNA. Rekombinowany DNA jest
Definicje Białka rekombinowane (ang. recombinant proteins, r-proteins) Białka, które powstały w żywych organizmach (lub liniach komórkowych) w wyniku ekspresji rekombinowanego DNA. Rekombinowany DNA jest
2. Enzymy pozwalające na manipulację DNA a. Polimerazy DNA b. Nukleazy c. Ligazy
 Metody analizy DNA 1. Budowa DNA. 2. Enzymy pozwalające na manipulację DNA a. Polimerazy DNA b. Nukleazy c. Ligazy 3. Klonowanie in vivo a. w bakteriach, wektory plazmidowe b. w fagach, kosmidy c. w drożdżach,
Metody analizy DNA 1. Budowa DNA. 2. Enzymy pozwalające na manipulację DNA a. Polimerazy DNA b. Nukleazy c. Ligazy 3. Klonowanie in vivo a. w bakteriach, wektory plazmidowe b. w fagach, kosmidy c. w drożdżach,
POLITECHNIKA ŁÓDZKA INSTRUKCJA Z LABORATORIUM W ZAKŁADZIE BIOFIZYKI. Ćwiczenie 3 ANALIZA TRANSPORTU SUBSTANCJI NISKOCZĄSTECZKOWYCH PRZEZ
 POLITECHNIKA ŁÓDZKA INSTRUKCJA Z LABORATORIUM W ZAKŁADZIE BIOFIZYKI Ćwiczenie 3 ANALIZA TRANSPORTU SUBSTANCJI NISKOCZĄSTECZKOWYCH PRZEZ BŁONĘ KOMÓRKOWĄ I. WSTĘP TEORETYCZNY Każda komórka, zarówno roślinna,
POLITECHNIKA ŁÓDZKA INSTRUKCJA Z LABORATORIUM W ZAKŁADZIE BIOFIZYKI Ćwiczenie 3 ANALIZA TRANSPORTU SUBSTANCJI NISKOCZĄSTECZKOWYCH PRZEZ BŁONĘ KOMÓRKOWĄ I. WSTĘP TEORETYCZNY Każda komórka, zarówno roślinna,
Ćwiczenia 1 Wirtualne Klonowanie Prowadzący: mgr inż. Joanna Tymeck-Mulik i mgr Lidia Gaffke. Część teoretyczna:
 Uniwersytet Gdański, Wydział Biologii Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią Biologia II rok ===============================================================================================
Uniwersytet Gdański, Wydział Biologii Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią Biologia II rok ===============================================================================================
Zasady oceniania rozwiązań zadań 48 Olimpiada Biologiczna Etap centralny
 Zasady oceniania rozwiązań zadań 48 Olimpiada Biologiczna Etap centralny Zadanie 1 1 pkt. za prawidłowe podanie typów dla obydwu zwierząt oznaczonych literami A oraz B. A. ramienionogi, B. mięczaki A.
Zasady oceniania rozwiązań zadań 48 Olimpiada Biologiczna Etap centralny Zadanie 1 1 pkt. za prawidłowe podanie typów dla obydwu zwierząt oznaczonych literami A oraz B. A. ramienionogi, B. mięczaki A.
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii
 Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Prowadzący: dr Lidia Boss znacznik fluorescencyjny (np. SYBR Green II)
 Uniwersytet Gdański, Wydział Biologii Biologia i Biologia Medyczna II rok Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią ============================================================================
Uniwersytet Gdański, Wydział Biologii Biologia i Biologia Medyczna II rok Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią ============================================================================
Biologia Molekularna z Biotechnologią ===============================================================================================
 Uniwersytet Gdański, Wydział Biologii Biologia i Biologia Medyczna II rok Katedra Genetyki Molekularnej Bakterii Katedra Biologii i Genetyki Medycznej Przedmiot: Katedra Biologii Molekularnej Biologia
Uniwersytet Gdański, Wydział Biologii Biologia i Biologia Medyczna II rok Katedra Genetyki Molekularnej Bakterii Katedra Biologii i Genetyki Medycznej Przedmiot: Katedra Biologii Molekularnej Biologia
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA
 Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa tel. 22 572 0735, 606448502
Zakład Biologii Molekularnej Materiały do ćwiczeń z przedmiotu: BIOLOGIA MOLEKULARNA Zakład Biologii Molekularnej Wydział Farmaceutyczny, WUM ul. Banacha 1, 02-097 Warszawa tel. 22 572 0735, 606448502
PODSTAWY BIOTECHNOLOGII Piotr Solarczyk
 PODSTAWY BIOTECHNOLOGII Piotr Solarczyk EFEKT KOŃCOWY Po zakończeniu seminarium powinieneś umieć: wyjaśnić pojęcia plazmid, fag, wektor, wektor ekspresyjny i czółenkowy, klonowanie, transfekcja, transformacja,
PODSTAWY BIOTECHNOLOGII Piotr Solarczyk EFEKT KOŃCOWY Po zakończeniu seminarium powinieneś umieć: wyjaśnić pojęcia plazmid, fag, wektor, wektor ekspresyjny i czółenkowy, klonowanie, transfekcja, transformacja,
Biologia medyczna, materiały dla studentów
 Zasada reakcji PCR Reakcja PCR (replikacja in vitro) obejmuje denaturację DNA, przyłączanie starterów (annealing) i syntezę nowych nici DNA (elongacja). 1. Denaturacja: rozplecenie nici DNA, temp. 94 o
Zasada reakcji PCR Reakcja PCR (replikacja in vitro) obejmuje denaturację DNA, przyłączanie starterów (annealing) i syntezę nowych nici DNA (elongacja). 1. Denaturacja: rozplecenie nici DNA, temp. 94 o
wykład dla studentów II roku biotechnologii Andrzej Wierzbicki
 Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Wykład 5 Droga od genu do
Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Wykład 5 Droga od genu do
TATA box. Enhancery. CGCG ekson intron ekson intron ekson CZĘŚĆ KODUJĄCA GENU TERMINATOR. Elementy regulatorowe
 Promotory genu Promotor bliski leży w odległości do 40 pz od miejsca startu transkrypcji, zawiera kasetę TATA. Kaseta TATA to silnie konserwowana sekwencja TATAAAA, występująca w większości promotorów
Promotory genu Promotor bliski leży w odległości do 40 pz od miejsca startu transkrypcji, zawiera kasetę TATA. Kaseta TATA to silnie konserwowana sekwencja TATAAAA, występująca w większości promotorów
Hybrydyzacja kwasów nukleinowych
 Hybrydyzacja kwasów nukleinowych Jaka jest lokalizacja genu na chromosomie? Jakie jest jego sąsiedztwo? Hybrydyzacja - powstawanie stabilnych struktur dwuniciowych z cząsteczek jednoniciowych o komplementarnych
Hybrydyzacja kwasów nukleinowych Jaka jest lokalizacja genu na chromosomie? Jakie jest jego sąsiedztwo? Hybrydyzacja - powstawanie stabilnych struktur dwuniciowych z cząsteczek jednoniciowych o komplementarnych
Najważniejsze z nich to: enzymy restrykcyjne wektory DNA inne enzymy np. ligazy, fosfatazy, polimerazy, nukleazy
 Aby manipulować genami niezbędne są odpowiednie narzędzia molekularne, które pozwalają uzyskać tzw. zrekombinowane DNA (umożliwiają rekombinację materiału genetycznego in vitro czyli w próbówce) Najważniejsze
Aby manipulować genami niezbędne są odpowiednie narzędzia molekularne, które pozwalają uzyskać tzw. zrekombinowane DNA (umożliwiają rekombinację materiału genetycznego in vitro czyli w próbówce) Najważniejsze
Wykład 14 Biosynteza białek
 BIOCHEMIA Kierunek: Technologia Żywności i Żywienie Człowieka semestr III Wykład 14 Biosynteza białek WYDZIAŁ NAUK O ŻYWNOŚCI I RYBACTWA CENTRUM BIOIMMOBILIZACJI I INNOWACYJNYCH MATERIAŁÓW OPAKOWANIOWYCH
BIOCHEMIA Kierunek: Technologia Żywności i Żywienie Człowieka semestr III Wykład 14 Biosynteza białek WYDZIAŁ NAUK O ŻYWNOŚCI I RYBACTWA CENTRUM BIOIMMOBILIZACJI I INNOWACYJNYCH MATERIAŁÓW OPAKOWANIOWYCH
WPROWADZENIE DO GENETYKI MOLEKULARNEJ
 WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
Przemiana materii i energii - Biologia.net.pl
 Ogół przemian biochemicznych, które zachodzą w komórce składają się na jej metabolizm. Wyróżnia się dwa antagonistyczne procesy metabolizmu: anabolizm i katabolizm. Szlak metaboliczny w komórce, to szereg
Ogół przemian biochemicznych, które zachodzą w komórce składają się na jej metabolizm. Wyróżnia się dwa antagonistyczne procesy metabolizmu: anabolizm i katabolizm. Szlak metaboliczny w komórce, to szereg
Bożena Nejman-Faleńczyk
 Kontrola replikacji fagów lambdoidalnych niosących geny toksyn Shiga w świetle potencjalnych nowych metod ich wykrywania i terapii zakażeń enterokrwotocznymi szczepami Escherichia coli Bożena Nejman-Faleńczyk
Kontrola replikacji fagów lambdoidalnych niosących geny toksyn Shiga w świetle potencjalnych nowych metod ich wykrywania i terapii zakażeń enterokrwotocznymi szczepami Escherichia coli Bożena Nejman-Faleńczyk
WPROWADZENIE DO GENETYKI MOLEKULARNEJ
 WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
Mechanizmy kontroli ekspresji genów w regulacji rozwoju bakteriofagów lambdoidalnych
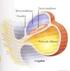 Mechanizmy kontroli ekspresji genów w regulacji rozwoju bakteriofagów lambdoidalnych Sylwia Bloch Bakteriofagi lambdoidalne to grupa wirusów bakteryjnych, zaliczanych do rodziny Siphoviridae, których najlepiej
Mechanizmy kontroli ekspresji genów w regulacji rozwoju bakteriofagów lambdoidalnych Sylwia Bloch Bakteriofagi lambdoidalne to grupa wirusów bakteryjnych, zaliczanych do rodziny Siphoviridae, których najlepiej
woda do 1000 ml ph=6,9-7,1. Po sterylizacji dodać nystatynę (końcowe stężenie ok. 50 μg/ml). Agar z wyciągiem glebowym i ekstraktem drożdżowym (YS)
 Ćwiczenie 1, 2, 3, 4 Skrining ze środowiska naturalnego: selekcja promieniowców zdolnych do produkcji antybiotyków. Testowanie zdolności do syntezy antybiotyków przez wyselekcjonowane szczepy promieniowców
Ćwiczenie 1, 2, 3, 4 Skrining ze środowiska naturalnego: selekcja promieniowców zdolnych do produkcji antybiotyków. Testowanie zdolności do syntezy antybiotyków przez wyselekcjonowane szczepy promieniowców
Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV
 Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV Cel ćwiczenia Określenie podatności na zakażenie wirusem HIV poprzez detekcję homo lub heterozygotyczności
Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV Cel ćwiczenia Określenie podatności na zakażenie wirusem HIV poprzez detekcję homo lub heterozygotyczności
TRANSKRYPCJA - I etap ekspresji genów
 Eksparesja genów TRANSKRYPCJA - I etap ekspresji genów Przepisywanie informacji genetycznej z makrocząsteczki DNA na mniejsze i bardziej funkcjonalne cząsteczki pre-mrna Polimeraza RNA ETAP I Inicjacja
Eksparesja genów TRANSKRYPCJA - I etap ekspresji genów Przepisywanie informacji genetycznej z makrocząsteczki DNA na mniejsze i bardziej funkcjonalne cząsteczki pre-mrna Polimeraza RNA ETAP I Inicjacja
Transformacja pośrednia składa się z trzech etapów:
 Transformacja pośrednia składa się z trzech etapów: 1. Otrzymanie pożądanego odcinka DNA z materiału genetycznego dawcy 2. Wprowadzenie obcego DNA do wektora 3. Wprowadzenie wektora, niosącego w sobie
Transformacja pośrednia składa się z trzech etapów: 1. Otrzymanie pożądanego odcinka DNA z materiału genetycznego dawcy 2. Wprowadzenie obcego DNA do wektora 3. Wprowadzenie wektora, niosącego w sobie
TRANSLACJA II etap ekspresji genów
 TRANSLACJA II etap ekspresji genów Tłumaczenie informacji genetycznej zawartej w mrna (po transkrypcji z DNA) na aminokwasy budujące konkretne białko. trna Operon (wg. Jacob i Monod) Zgrupowane w jednym
TRANSLACJA II etap ekspresji genów Tłumaczenie informacji genetycznej zawartej w mrna (po transkrypcji z DNA) na aminokwasy budujące konkretne białko. trna Operon (wg. Jacob i Monod) Zgrupowane w jednym
TESTY Z BIOLOGII (fragment)
 TESTY Z BIOLOGII (fragment) Autor:...Dawid Pawlos Projekt okładki:...rafael Pawlos Korekta językowa:...agnieszka Sanetra Wydanie I Copyright by Dawid Pawlos 2015 Wszelkie prawa zastrzeżone. Kopiowanie
TESTY Z BIOLOGII (fragment) Autor:...Dawid Pawlos Projekt okładki:...rafael Pawlos Korekta językowa:...agnieszka Sanetra Wydanie I Copyright by Dawid Pawlos 2015 Wszelkie prawa zastrzeżone. Kopiowanie
Hybrydyzacja kwasów nukleinowych
 Hybrydyzacja kwasów nukleinowych Jaka jest lokalizacja tego genu na chromosomie? Jakie jest jego sąsiedztwo? Hybrydyzacja - powstawanie stabilnych struktur dwuniciowych z cząsteczek jednoniciowych o komplementarnych
Hybrydyzacja kwasów nukleinowych Jaka jest lokalizacja tego genu na chromosomie? Jakie jest jego sąsiedztwo? Hybrydyzacja - powstawanie stabilnych struktur dwuniciowych z cząsteczek jednoniciowych o komplementarnych
Novabeads Food DNA Kit
 Novabeads Food DNA Kit Novabeads Food DNA Kit jest nowej generacji narzędziem w technikach biologii molekularnej, umożliwiającym izolację DNA z produktów spożywczych wysoko przetworzonych. Metoda oparta
Novabeads Food DNA Kit Novabeads Food DNA Kit jest nowej generacji narzędziem w technikach biologii molekularnej, umożliwiającym izolację DNA z produktów spożywczych wysoko przetworzonych. Metoda oparta
Dominika Stelmach Gr. 10B2
 Dominika Stelmach Gr. 10B2 Czym jest DNA? Wielkocząsteczkowy organiczny związek chemiczny z grupy kwasów nukleinowych Zawiera kwas deoksyrybonukleoinowy U organizmów eukariotycznych zlokalizowany w jądrze
Dominika Stelmach Gr. 10B2 Czym jest DNA? Wielkocząsteczkowy organiczny związek chemiczny z grupy kwasów nukleinowych Zawiera kwas deoksyrybonukleoinowy U organizmów eukariotycznych zlokalizowany w jądrze
Konstrukcja wektora plazmidowego DNA do klonowania genów i/lub wektora plazmidowego do sekrecji w bakteriach mlekowych
 Konstrukcja wektora plazmidowego DNA do klonowania genów i/lub wektora plazmidowego do sekrecji w bakteriach mlekowych Łukasz Tranda Promotor: doc. dr hab. Jacek Bardowski, IBB Promotor: dr hab. Edward
Konstrukcja wektora plazmidowego DNA do klonowania genów i/lub wektora plazmidowego do sekrecji w bakteriach mlekowych Łukasz Tranda Promotor: doc. dr hab. Jacek Bardowski, IBB Promotor: dr hab. Edward
Najważniejsze z nich to: enzymy restrykcyjne wektory DNA inne enzymy np. ligazy, fosfatazy, polimerazy, nukleazy
 Aby manipulować genami niezbędne są odpowiednie narzędzia molekularne, które pozwalają uzyskać tzw. zrekombinowane DNA (umożliwiają rekombinację materiału genetycznego in vitro czyli w próbówce) Najważniejsze
Aby manipulować genami niezbędne są odpowiednie narzędzia molekularne, które pozwalają uzyskać tzw. zrekombinowane DNA (umożliwiają rekombinację materiału genetycznego in vitro czyli w próbówce) Najważniejsze
Proteomika: umożliwia badanie zestawu wszystkich lub prawie wszystkich białek komórkowych
 Proteomika: umożliwia badanie zestawu wszystkich lub prawie wszystkich białek komórkowych Zalety w porównaniu z analizą trankryptomu: analiza transkryptomu komórki identyfikacja mrna nie musi jeszcze oznaczać
Proteomika: umożliwia badanie zestawu wszystkich lub prawie wszystkich białek komórkowych Zalety w porównaniu z analizą trankryptomu: analiza transkryptomu komórki identyfikacja mrna nie musi jeszcze oznaczać
ZAKŁAD WIRUSOLOGII Instytut Mikrobiologii
 ZAKŁAD WIRUSOLOGII Instytut Mikrobiologii DNI OTWARTE 11-15.05.2015 w GODZ. 10-14 www.biol.uw.edu.pl/wirusologia Zespół Dr Monika Radlińska kierownik (+22) 554 14 19 e-mail: m.radlinska@biol.uw.edu.pl
ZAKŁAD WIRUSOLOGII Instytut Mikrobiologii DNI OTWARTE 11-15.05.2015 w GODZ. 10-14 www.biol.uw.edu.pl/wirusologia Zespół Dr Monika Radlińska kierownik (+22) 554 14 19 e-mail: m.radlinska@biol.uw.edu.pl
TECHNIKI ANALIZY RNA TECHNIKI ANALIZY RNA TECHNIKI ANALIZY RNA
 DNA 28SRNA 18/16S RNA 5SRNA mrna Ilościowa analiza mrna aktywność genów w zależności od wybranych czynników: o rodzaju tkanki o rodzaju czynnika zewnętrznego o rodzaju upośledzenia szlaku metabolicznego
DNA 28SRNA 18/16S RNA 5SRNA mrna Ilościowa analiza mrna aktywność genów w zależności od wybranych czynników: o rodzaju tkanki o rodzaju czynnika zewnętrznego o rodzaju upośledzenia szlaku metabolicznego
Ryc 1. Budowa operonu laktozowego.
 Uniwersytet Gdański, Wydział Biologii Biologia i Biologia medyczna II rok Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią =================================================================
Uniwersytet Gdański, Wydział Biologii Biologia i Biologia medyczna II rok Katedra Biologii Molekularnej Przedmiot: Biologia Molekularna z Biotechnologią =================================================================
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych???
 Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Wybrane techniki badania białek -proteomika funkcjonalna
 Wybrane techniki badania białek -proteomika funkcjonalna Proteomika: umożliwia badanie zestawu wszystkich (lub prawie wszystkich) białek komórkowych Zalety analizy proteomu np. w porównaniu z analizą trankryptomu:
Wybrane techniki badania białek -proteomika funkcjonalna Proteomika: umożliwia badanie zestawu wszystkich (lub prawie wszystkich) białek komórkowych Zalety analizy proteomu np. w porównaniu z analizą trankryptomu:
INICJACJA ELONGACJA TERMINACJA
 INICJACJA ELONGACJA TERMINACJA 2007 by National Academy of Sciences Kornberg R D PNAS 2007;104:12955-12961 Struktura chromatyny pozwala na różny sposób odczytania informacji zawartej w DNA. Możliwe staje
INICJACJA ELONGACJA TERMINACJA 2007 by National Academy of Sciences Kornberg R D PNAS 2007;104:12955-12961 Struktura chromatyny pozwala na różny sposób odczytania informacji zawartej w DNA. Możliwe staje
BUDOWA I FUNKCJA GENOMU LUDZKIEGO
 BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
Endonukleazy restrykcyjne molekularne nożyczki
 Endonukleazy restrykcyjne molekularne nożyczki Inżynieria genetyczna nauka o technikach badawczych pozwalających na ingerencję w materiał genetyczny organizmów w celu wyizolowania i charakterystyki określonych
Endonukleazy restrykcyjne molekularne nożyczki Inżynieria genetyczna nauka o technikach badawczych pozwalających na ingerencję w materiał genetyczny organizmów w celu wyizolowania i charakterystyki określonych
Geny i działania na nich
 Metody bioinformatyki Geny i działania na nich prof. dr hab. Jan Mulawka Trzy królestwa w biologii Prokaryota organizmy, których komórki nie zawierają jądra, np. bakterie Eukaryota - organizmy, których
Metody bioinformatyki Geny i działania na nich prof. dr hab. Jan Mulawka Trzy królestwa w biologii Prokaryota organizmy, których komórki nie zawierają jądra, np. bakterie Eukaryota - organizmy, których
Ćwiczenie 7 Klonowanie DNA w wektorach plazmidowych
 Ćwiczenie 7 Klonowanie DNA w wektorach plazmidowych Celem ćwiczenia jest sklonowanie fragmentu DNA (powielonego techniką PCR i wyizolowanego z żelu agarozowego na poprzednich zajęciach) do wektora plazmidowego
Ćwiczenie 7 Klonowanie DNA w wektorach plazmidowych Celem ćwiczenia jest sklonowanie fragmentu DNA (powielonego techniką PCR i wyizolowanego z żelu agarozowego na poprzednich zajęciach) do wektora plazmidowego
Antywirulentny potencjał białek fagowych
 Antywirulentny potencjał białek fagowych Grażyna Majkowska-Skrobek Zakład Biologii Patogenów i Immunologii Instytut Genetyki i Mikrobiologii UWr IMMUNOGENNOŚĆ BAKTERII PATOGENNOŚĆ BAKTERII BAKTERIOFAGI
Antywirulentny potencjał białek fagowych Grażyna Majkowska-Skrobek Zakład Biologii Patogenów i Immunologii Instytut Genetyki i Mikrobiologii UWr IMMUNOGENNOŚĆ BAKTERII PATOGENNOŚĆ BAKTERII BAKTERIOFAGI
Laboratoria.net Innowacje Nauka Technologie
 Akceptuję W ramach naszej witryny stosujemy pliki cookies w celu świadczenia państwu usług na najwyższym poziomie, w tym w sposób dostosowany do indywidualnych potrzeb. Korzystanie z witryny bez zmiany
Akceptuję W ramach naszej witryny stosujemy pliki cookies w celu świadczenia państwu usług na najwyższym poziomie, w tym w sposób dostosowany do indywidualnych potrzeb. Korzystanie z witryny bez zmiany
The Role of Maf1 Protein in trna Processing and Stabilization / Rola białka Maf1 w dojrzewaniu i kontroli stabilności trna
 Streszczenie rozprawy doktorskiej pt. The Role of Maf1 Protein in trna Processing and Stabilization / Rola białka Maf1 w dojrzewaniu i kontroli stabilności trna mgr Tomasz Turowski, promotor prof. dr hab.
Streszczenie rozprawy doktorskiej pt. The Role of Maf1 Protein in trna Processing and Stabilization / Rola białka Maf1 w dojrzewaniu i kontroli stabilności trna mgr Tomasz Turowski, promotor prof. dr hab.
Zgodnie z tzw. modelem interpunkcji trna, cząsteczki mt-trna wyznaczają miejsca
 Tytuł pracy: Autor: Promotor rozprawy: Recenzenci: Funkcje białek ELAC2 i SUV3 u ssaków i ryb Danio rerio. Praca doktorska wykonana w Instytucie Genetyki i Biotechnologii, Wydział Biologii UW Lien Brzeźniak
Tytuł pracy: Autor: Promotor rozprawy: Recenzenci: Funkcje białek ELAC2 i SUV3 u ssaków i ryb Danio rerio. Praca doktorska wykonana w Instytucie Genetyki i Biotechnologii, Wydział Biologii UW Lien Brzeźniak
Making the impossible possible: the metamorphosis of Polish Biology Olympiad
 Making the impossible possible: the metamorphosis of Polish Biology Olympiad Takao Ishikawa Faculty of Biology, University of Warsaw, Poland Performance of Polish students at IBO Gold Silver Bronze Merit
Making the impossible possible: the metamorphosis of Polish Biology Olympiad Takao Ishikawa Faculty of Biology, University of Warsaw, Poland Performance of Polish students at IBO Gold Silver Bronze Merit
TaqNovaHS. Polimeraza DNA RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925
 TaqNovaHS RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 TaqNovaHS Polimeraza TaqNovaHS jest mieszaniną termostabilnej polimerazy DNA
TaqNovaHS RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 RP902A, RP905A, RP910A, RP925A RP902, RP905, RP910, RP925 TaqNovaHS Polimeraza TaqNovaHS jest mieszaniną termostabilnej polimerazy DNA
Laboratorium 7. Testy na genotoksyczność
 Test rewersji mutacji test Amesa Laboratorium 7 Testy na genotoksyczność Test Amesa jest testem bakteryjno-ssaczym. Organizmami testowymi są tutaj bakterie Salmonella typhimurium. W wyniku mutacji, celowo
Test rewersji mutacji test Amesa Laboratorium 7 Testy na genotoksyczność Test Amesa jest testem bakteryjno-ssaczym. Organizmami testowymi są tutaj bakterie Salmonella typhimurium. W wyniku mutacji, celowo
BUDOWA, REPLIKACJA, SYSTEMATYKA WIRUSÓW METODY HODOWLI WIRUSÓW. Katedra i Zakład Mikrobiologii Lekarskiej Anna Majewska Warszawa, 2014
 BUDOWA, REPLIKACJA, SYSTEMATYKA WIRUSÓW METODY HODOWLI WIRUSÓW Katedra i Zakład Mikrobiologii Lekarskiej Anna Majewska Warszawa, 2014 OGÓLNA CHARAKTERYSTYKA WIRUSÓW Małe rozmiary (20 nm-300 średnicy) Brak
BUDOWA, REPLIKACJA, SYSTEMATYKA WIRUSÓW METODY HODOWLI WIRUSÓW Katedra i Zakład Mikrobiologii Lekarskiej Anna Majewska Warszawa, 2014 OGÓLNA CHARAKTERYSTYKA WIRUSÓW Małe rozmiary (20 nm-300 średnicy) Brak
Powodzenie reakcji PCR wymaga właściwego doboru szeregu parametrów:
 Powodzenie reakcji PCR wymaga właściwego doboru szeregu parametrów: dobór warunków samej reakcji PCR (temperatury, czas trwania cykli, ilości cykli itp.) dobór odpowiednich starterów do reakcji amplifikacji
Powodzenie reakcji PCR wymaga właściwego doboru szeregu parametrów: dobór warunków samej reakcji PCR (temperatury, czas trwania cykli, ilości cykli itp.) dobór odpowiednich starterów do reakcji amplifikacji
Zagadnienia egzaminacyjne z przedmiotu Mikrobiologia kosmetologiczna dla studentów II roku kierunku Kosmetologia
 Zagadnienia egzaminacyjne z przedmiotu Mikrobiologia kosmetologiczna dla studentów II roku kierunku Kosmetologia I. Zagadnienia omawiane na wykładach. Uzupełnieniem zagadnień omawianych na wykładach są
Zagadnienia egzaminacyjne z przedmiotu Mikrobiologia kosmetologiczna dla studentów II roku kierunku Kosmetologia I. Zagadnienia omawiane na wykładach. Uzupełnieniem zagadnień omawianych na wykładach są
6. Z pięciowęglowego cukru prostego, zasady azotowej i reszty kwasu fosforowego, jest zbudowany A. nukleotyd. B. aminokwas. C. enzym. D. wielocukier.
 ID Testu: F5679R8 Imię i nazwisko ucznia Klasa Data 1. Na indywidualne cechy danego osobnika ma (maja) wpływ A. wyłacznie czynniki środowiskowe. B. czynniki środowiskowe i materiał genetyczny. C. wyłacznie
ID Testu: F5679R8 Imię i nazwisko ucznia Klasa Data 1. Na indywidualne cechy danego osobnika ma (maja) wpływ A. wyłacznie czynniki środowiskowe. B. czynniki środowiskowe i materiał genetyczny. C. wyłacznie
Wektory DNA - klonowanie molekularne
 Wektory DNA - klonowanie molekularne Fragment DNA (np. pojedynczy gen) można trwale wprowadzić do komórek gospodarza (tzn. zmusić go do powielania w tym gospodarzu) tylko wtedy, gdy zostanie on wbudowany
Wektory DNA - klonowanie molekularne Fragment DNA (np. pojedynczy gen) można trwale wprowadzić do komórek gospodarza (tzn. zmusić go do powielania w tym gospodarzu) tylko wtedy, gdy zostanie on wbudowany
Priony. co dobrego mówią nam drożdże? Takao Ishikawa Zakład Biologii Molekularnej Uniwersytet Warszawski
 Priony co dobrego mówią nam drożdże? Takao Ishikawa Zakład Biologii Molekularnej Uniwersytet Warszawski Choroba Kreutzfeldta-Jakoba Pierwsze opisy pochodzą z lat 30. XX wieku Zakaźna choroba, często rodzinna
Priony co dobrego mówią nam drożdże? Takao Ishikawa Zakład Biologii Molekularnej Uniwersytet Warszawski Choroba Kreutzfeldta-Jakoba Pierwsze opisy pochodzą z lat 30. XX wieku Zakaźna choroba, często rodzinna
Inżynieria genetyczna
 Inżynieria genetyczna i technologia rekombinowanego DNA Dr n. biol. Urszula Wasik Zakład Biologii Medycznej Inżynieria genetyczna świadoma, celowa, kontrolowana ingerencja w materiał genetyczny organizmów
Inżynieria genetyczna i technologia rekombinowanego DNA Dr n. biol. Urszula Wasik Zakład Biologii Medycznej Inżynieria genetyczna świadoma, celowa, kontrolowana ingerencja w materiał genetyczny organizmów
Endonukleazy restrykcyjne molekularne nożyczki
 Endonukleazy restrykcyjne molekularne nożyczki Inżynieria genetyczna nauka o technikach (narzędziach) biologii molekularnej pozwalających na świadomą i zamierzoną ingerencję w materiał genetyczny organizmów
Endonukleazy restrykcyjne molekularne nożyczki Inżynieria genetyczna nauka o technikach (narzędziach) biologii molekularnej pozwalających na świadomą i zamierzoną ingerencję w materiał genetyczny organizmów
Endonukleazy restrykcyjne molekularne nożyczki
 Endonukleazy restrykcyjne molekularne nożyczki Inżynieria genetyczna nauka o technikach (narzędziach) biologii molekularnej pozwalających na świadomą i zamierzoną ingerencję w materiał genetyczny organizmów
Endonukleazy restrykcyjne molekularne nożyczki Inżynieria genetyczna nauka o technikach (narzędziach) biologii molekularnej pozwalających na świadomą i zamierzoną ingerencję w materiał genetyczny organizmów
Badanie dynamiki białek jądrowych w żywych komórkach metodą mikroskopii konfokalnej
 Badanie dynamiki białek jądrowych w żywych komórkach metodą mikroskopii konfokalnej PRAKTIKUM Z BIOLOGII KOMÓRKI () ćwiczenie prowadzone we współpracy z Pracownią Biofizyki Komórki Badanie dynamiki białek
Badanie dynamiki białek jądrowych w żywych komórkach metodą mikroskopii konfokalnej PRAKTIKUM Z BIOLOGII KOMÓRKI () ćwiczenie prowadzone we współpracy z Pracownią Biofizyki Komórki Badanie dynamiki białek
Adam Dawidowski Klasa III b. mgr Beata Ulczyńska
 Adam Dawidowski Klasa III b (autor) mgr Beata Ulczyńska (opiekun) BADANIE WPŁYWU GENU orf654 Z OPERONU mboii NA ZJAWISKO PRZESUNIĘCIA RAMKI ODCZYTU TRANSLACJI -1 W ZMUTOWANYM GENIE mboiim2δ I Akademickie
Adam Dawidowski Klasa III b (autor) mgr Beata Ulczyńska (opiekun) BADANIE WPŁYWU GENU orf654 Z OPERONU mboii NA ZJAWISKO PRZESUNIĘCIA RAMKI ODCZYTU TRANSLACJI -1 W ZMUTOWANYM GENIE mboiim2δ I Akademickie
Sesja sponsorowana przez Polską Sieć Biologii Molekularnej SESJA 1 ORGANIZACJA MATERIAŁU GENETYCZNEGO WYKŁADY
 Sesja sponsorowana przez Polską Sieć Biologii Molekularnej SESJA 1 ORGANIZACJA MATERIAŁU GENETYCZNEGO WYKŁADY 12 SESJA 1 WYKŁADY W01-01 ORGANIZACJA GENOMU BAKTERIOFAGA P1, IMPLIKACJE DLA PROCESU PROPAGACJI
Sesja sponsorowana przez Polską Sieć Biologii Molekularnej SESJA 1 ORGANIZACJA MATERIAŁU GENETYCZNEGO WYKŁADY 12 SESJA 1 WYKŁADY W01-01 ORGANIZACJA GENOMU BAKTERIOFAGA P1, IMPLIKACJE DLA PROCESU PROPAGACJI
JĄDRO KOMÓRKOWE I ORGANIZACJA CHROMATYNY
 Wykład: 2 JĄDRO KOMÓRKOWE I ORGANIZACJA CHROMATYNY Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej Jądro komórkowe 1 Jądro komórkowe Otoczka jądrowa zewnętrzna membrana jądrowa wewnętrzna
Wykład: 2 JĄDRO KOMÓRKOWE I ORGANIZACJA CHROMATYNY Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej Jądro komórkowe 1 Jądro komórkowe Otoczka jądrowa zewnętrzna membrana jądrowa wewnętrzna
Instytut Mikrobiologii
 Instytut Mikrobiologii Warto zostać mikrobiologiem! Zrób licencjat w Instytucie Mikrobiologii UW (a potem pracę magisterską i doktorat) Badamy biologię oraz genetyczne podstawy funkcjonowania bakterii
Instytut Mikrobiologii Warto zostać mikrobiologiem! Zrób licencjat w Instytucie Mikrobiologii UW (a potem pracę magisterską i doktorat) Badamy biologię oraz genetyczne podstawy funkcjonowania bakterii
Wybrane techniki badania białek -proteomika funkcjonalna
 Wybrane techniki badania białek -proteomika funkcjonalna Proteomika: umożliwia badanie zestawu wszystkich (lub prawie wszystkich) białek komórkowych Zalety analizy proteomu w porównaniu z analizą trankryptomu:
Wybrane techniki badania białek -proteomika funkcjonalna Proteomika: umożliwia badanie zestawu wszystkich (lub prawie wszystkich) białek komórkowych Zalety analizy proteomu w porównaniu z analizą trankryptomu:
Wykład: 2 JĄDRO KOMÓRKOWE I ORGANIZACJA CHROMATYNY. Jądro komórkowe. Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej.
 Wykład: 2 JĄDRO KOMÓRKOWE I ORGANIZACJA CHROMATYNY Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej Jądro komórkowe Jądro komórkowe Otoczka jądrowa zewnętrzna membrana jądrowa wewnętrzna
Wykład: 2 JĄDRO KOMÓRKOWE I ORGANIZACJA CHROMATYNY Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej Jądro komórkowe Jądro komórkowe Otoczka jądrowa zewnętrzna membrana jądrowa wewnętrzna
Replikacja DNA. Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego.
 Replikacja DNA Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Replikacja DNA jest bardzo złożonym procesem, w którym biorą udział setki
Replikacja DNA Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Replikacja DNA jest bardzo złożonym procesem, w którym biorą udział setki
Mikrosatelitarne sekwencje DNA
 Mikrosatelitarne sekwencje DNA Małgorzata Pałucka Wykorzystanie sekwencji mikrosatelitarnych w jądrowym DNA drzew leśnych do udowodnienia pochodzenia materiału dowodowego w postępowaniu sądowym 27.09.2012
Mikrosatelitarne sekwencje DNA Małgorzata Pałucka Wykorzystanie sekwencji mikrosatelitarnych w jądrowym DNA drzew leśnych do udowodnienia pochodzenia materiału dowodowego w postępowaniu sądowym 27.09.2012
Przeglądanie bibliotek
 Przeglądanie bibliotek Czyli jak złapać (i sklonować) ciekawy gen? Klonowanie genów w oparciu o identyczność lub podobieństwo ich sekwencji do znanego już DNA Sonda homologiczna (komplementarna w 100%)
Przeglądanie bibliotek Czyli jak złapać (i sklonować) ciekawy gen? Klonowanie genów w oparciu o identyczność lub podobieństwo ich sekwencji do znanego już DNA Sonda homologiczna (komplementarna w 100%)
Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej
 Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Inżynieria Genetyczna ćw. 1
 Materiały do ćwiczeń z przedmiotu Genetyka z inżynierią genetyczną D - blok Inżynieria Genetyczna ćw. 1 Instytut Genetyki i Biotechnologii, Wydział Biologii, Uniwersytet Warszawski, rok akad. 2018/2019
Materiały do ćwiczeń z przedmiotu Genetyka z inżynierią genetyczną D - blok Inżynieria Genetyczna ćw. 1 Instytut Genetyki i Biotechnologii, Wydział Biologii, Uniwersytet Warszawski, rok akad. 2018/2019
