Komputerowe wspomaganie projektowanie leków
|
|
|
- Edward Sobczak
- 9 lat temu
- Przeglądów:
Transkrypt
1 Komputerowe wspomaganie projektowanie leków wykład V Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III Strategie projektowania leków Dopasowanie ligandów do miejsca aktywnego receptora (dokowanie) znana Budowa nowych ligandów ab-initio (de-novo) Struktura receptora Dynamika kompleksu receptor-ligand Budowanie modelu miejsc aktywnych liganda (farmakofor) nieznana Przeszukiwanie baz danych (screening) 1D i 3D QSAR (pseudoreceptory i pola molekularne) 2 1
2 Modyfikacje liganda w celu zwiększenia oddziaływania z celem molekularnym Struktura kompleksu nie jest znana Poszukiwanie miejsca aktywnego (informacja biochemiczna lub wnęka/rowek na powierzchni receptora Dokowanie znanych ligandów tego białka lub przeszukiwanie całych bibliotek różnych ligandów Struktura kompleksu jest znana Analiza istniejących oddziaływań lek-białko Maksymalizacja liczby i siły oddziaływań Ocena efektów entropowych (zmniejszenie liczby konformacji, schowanie części hydrofobowych) 3 Analiza oddziaływań w kompleksie biotyna-streptoawidyna Silne wiązanie biotyny (witamina B7) K D mol/dm 3 (0.01 pm) Streptoawidyna jest uzyskiwana z bakterii Streptomyces avidinii. Znalazła zastosowanie w biotechnologii do oczyszczania białek (pi 7 więc nie wiąże się niespecyficznie z innymi białkami). Biotyna-awidyna nawet pm ale dla awidyny pi 10. Awidyna (antywitamina) - składnik jaj ptaków unieczynniana przez gotowanie. 4 2
3 Analiza oddziaływań w kompleksie biotyna-streptoawidyna 5 8 reguł projektowania leków dla receptor-based drug design 1. Znaleźć kluczowe miejsca oddziaływania z receptorem 2. Wykorzystać oddziaływania hydrofobowe (głównie efekt entropowy, dużo wypartej wody z receptora); oddziaływania bezkierunkowe 3. Wykorzystać wiązania wodorowe (kąt C=O H jest 120, ale mogą się także zbliżać do 180 w -kartkach; dla N-H O jest pomiędzy 140 a Wykorzystać oddziaływania jonowe 6 3
4 8 reguł projektowania leków dla receptor-based drug design 5. Konformacja bioaktywna zbliżona do konformacji o najniższej energii (mała energia naprężeń) 6. Zoptymalizować kontakty van der Waalsa: dopasować kształty liganda i receptora, unikać nakładania powierzchni molekularnych (bumps). 7. Usuwać strukturalną wodę 8. Zmniejszać niekorzystane efekty entropowe poprzez usztywnianie liganda. 7 Analiza oddziaływań w kompleksie oseltamivir (tamiflu) - neuramidaza wirusa grypy H5N1 8 4
5 Analiza oddziaływań w kompleksie oseltamivir (tamiflu) - neuramidaza wirusa grypy H5N1 Tamiflu Kwas sialowy - substrat 9 Działanie proteazy wirusa HIV 10 5
6 Struktura proteazy wirusa HIV-1 Pierwsza struktura krystaliczna w 1989 r. 11 Schemat inhibitora stanu przejściowego dla proteazy 12 6
7 Topografia miejsca aktywnego proteazy 13 Inhibitor MVT-101 Heksapeptyd, K D = 780 nm 14 7
8 Oddziaływania MVT-101 z proteazą HIV-1 Oddziaływania poprzez cząsteczkę wody Wiązania wodorowe do inhibitora 15 Projektowanie dodatkowych miejsc hydrofobowych inhibitor A K D = 0.15 nm, 5000 x silniejsze wiązanie niż MVT
9 Analiza wiązania inhibitora A Oddziaływania także poprzez cząsteczkę wody Wiązania wodorowe do obu Asp25 17 Zbudowanie farmakofora na podstawie zadokowanego liganda Prosty farmakofor: dwie symetryczne grupy hydrofobowe i jedno miejsce HB donor/akceptor 18 9
10 Jeden ze znalezionych w bazie danych związków 19 Analiza zadokowania Grupa metoksylowa -O-CH 3 zastępuje strukturalną wodę 20 10
11 Analiza dopasowania Usunięcie strukturalnej cząsteczki wody jest korzystne entropowo 21 Analiza dopasowania Usztywnienie liganda jest także korzystne entropowo 22 11
12 Modyfikacje środkowego pierścienia cykloheksanon Pierścień 7-członowy (korzystniejsze wiązania do obu Asp25 23 Dodanie pierścieni naftylowych Nowa seria związków: cykliczne pochodne mocznika 24 12
13 Analiza wiązania XK Dynamika molekularna kompleksu ligand-cel molekularny Pobranie struktury z bazy PDB (Protein Data Bank) Usunięcie dodatkowych molekuł i jonów użytych do krystalizacji Uzupełnienie brakujących elementów struktury Niewidoczne w krysztale aminokwasy (np. w labilnych pętlach) Atomy wodoru (zbyt niska rozdzielczość) + optymalizacja ich położeń Ustalenie warunków prowadzenia symulacji Wybór pola siłowego Ustalenie temperatury i ciśnienia (zwykle standardowe) Parametryzacja liganda: ładunki cząstkowe (dla wszystkich atomów liganda) i parametry pola siłowego (dla nowych typów atomów liganda) 13
14 Dynamika molekularna kompleksu ligand-cel mol. - stopniowe usuwanie więzów (czasy symulacji dla enzymów ok aminokwasów) Dodanie cząsteczek wody w pudle periodycznym Dynamika samej wody kompleks zamrożony (1 ns) Odmrożenie liganda (100 ps) Odmrożenie łańcuchów bocznych aminokwasów (1 ns) Odmrożenie łańcucha głównego (20 ns) Właściwa symulacja (100 ns lub więcej dla większych białek) MD biotyna-streptoawidyna 14
Komputerowe wspomaganie projektowanie leków
 Komputerowe wspomaganie projektowanie leków wykład VI Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Komputerowe wspomaganie projektowanie leków wykład VI Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Komputerowe wspomaganie projektowanie leków
 Komputerowe wspomaganie projektowanie leków wykład IV Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Komputerowe wspomaganie projektowanie leków wykład IV Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Wykład 5-6. leki przeciwwirusowe i przeciwnowotworowe
 Wykład 5-6 leki przeciwwirusowe i przeciwnowotworowe Budowa i działanie wirusów Wirusy RNA: grypa, rotawirus, SARS, wir. zap. wątroby A i C, Ebola, HIV Wirusy DNA: - ospa prawdziwa (smallpox), - ospa wietrzna
Wykład 5-6 leki przeciwwirusowe i przeciwnowotworowe Budowa i działanie wirusów Wirusy RNA: grypa, rotawirus, SARS, wir. zap. wątroby A i C, Ebola, HIV Wirusy DNA: - ospa prawdziwa (smallpox), - ospa wietrzna
cz. VII Metody projektowania leków i modelowanie molekularne
 Oddziaływanie leków z celami molekularnymi cz. VII Metody projektowania leków i modelowanie molekularne Prof. dr hab. Sławomir Filipek Uniwersytet Warszawski, Wydział Chemii 1 Strategie projektowania leków
Oddziaływanie leków z celami molekularnymi cz. VII Metody projektowania leków i modelowanie molekularne Prof. dr hab. Sławomir Filipek Uniwersytet Warszawski, Wydział Chemii 1 Strategie projektowania leków
Podstawy projektowania leków wykład 12
 Podstawy projektowania leków wykład 12 Łukasz Berlicki Projektowanie wspomagane komputerowo Ligand-based design QSAR i 3D-QSAR Structure-based design projektowanie oparte na strukturze celu molekularnego
Podstawy projektowania leków wykład 12 Łukasz Berlicki Projektowanie wspomagane komputerowo Ligand-based design QSAR i 3D-QSAR Structure-based design projektowanie oparte na strukturze celu molekularnego
QSAR i związki z innymi metodami. Karol Kamel Uniwersytet Warszawski
 QSAR i związki z innymi metodami Wstęp QSAR Quantitative Structure-Activity Relationship. Jest to metoda polegająca na znalezieniu (i analizie) zależności pomiędzy strukturą chemiczną (geometria cząsteczki,
QSAR i związki z innymi metodami Wstęp QSAR Quantitative Structure-Activity Relationship. Jest to metoda polegająca na znalezieniu (i analizie) zależności pomiędzy strukturą chemiczną (geometria cząsteczki,
Komputerowe wspomaganie projektowania leków
 Komputerowe wspomaganie projektowania leków MECHANIKA MOLEKULARNA I KWANTOWA W MM korzysta się z równań wynikających z praw fizyki klasycznej i stosuje się je do jader atomów z pominięciem elektronów,
Komputerowe wspomaganie projektowania leków MECHANIKA MOLEKULARNA I KWANTOWA W MM korzysta się z równań wynikających z praw fizyki klasycznej i stosuje się je do jader atomów z pominięciem elektronów,
Structure and Charge Density Studies of Pharmaceutical Substances in the Solid State
 Maura Malińska Wydział Chemii, Uniwersytet Warszawski Promotorzy: prof. dr hab. Krzysztof Woźniak, prof. dr hab. Andrzej Kutner Structure and Charge Density Studies of Pharmaceutical Substances in the
Maura Malińska Wydział Chemii, Uniwersytet Warszawski Promotorzy: prof. dr hab. Krzysztof Woźniak, prof. dr hab. Andrzej Kutner Structure and Charge Density Studies of Pharmaceutical Substances in the
Oddziaływanie leków z celami molekularnymi i projektowanie leków
 Oddziaływanie leków z celami molekularnymi i projektowanie leków Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania (biomodellab.eu) Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych
Oddziaływanie leków z celami molekularnymi i projektowanie leków Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania (biomodellab.eu) Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych
Projektowanie Nowych Chemoterapeutyków
 Jan Mazerski Katedra Technologii Leków i Biochemii Wydział Chemiczny Projektowanie Nowych Chemoterapeutyków XV. QSAR 3D QSAR w przestrzeni Rozwój metod ustalania struktury 3D dla białek i ich kompleksów.
Jan Mazerski Katedra Technologii Leków i Biochemii Wydział Chemiczny Projektowanie Nowych Chemoterapeutyków XV. QSAR 3D QSAR w przestrzeni Rozwój metod ustalania struktury 3D dla białek i ich kompleksów.
Podstawy projektowania leków wykład 13
 odstawy projektowania leków wykład 13 Łukasz Berlicki rojektowanie wspomagane komputerowo rojektowanie leków oparte na strukturze często wykorzystuje metody komputerowe, aby: rzeanalizować duŝą liczbę
odstawy projektowania leków wykład 13 Łukasz Berlicki rojektowanie wspomagane komputerowo rojektowanie leków oparte na strukturze często wykorzystuje metody komputerowe, aby: rzeanalizować duŝą liczbę
Komputerowe wspomaganie projektowanie leków
 Komputerowe wspomaganie projektowanie leków wykład II Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Komputerowe wspomaganie projektowanie leków wykład II Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Dokowanie molekularne. Karol Kamel Uniwersytet Warszawski
 molekularne Wstęp Dokowanie metoda modelowania molekularnego, pozwalająca na znalezienie położenia (i konformacji) liganda w miejscu wiążącym receptora. Informacja ta pozwala na ocenę energii swobodnej
molekularne Wstęp Dokowanie metoda modelowania molekularnego, pozwalająca na znalezienie położenia (i konformacji) liganda w miejscu wiążącym receptora. Informacja ta pozwala na ocenę energii swobodnej
Model wiązania kowalencyjnego cząsteczka H 2
 Model wiązania kowalencyjnego cząsteczka H 2 + Współrzędne elektronu i protonów Orbitale wiążący i antywiążący otrzymane jako kombinacje orbitali atomowych Orbital wiążący duża gęstość ładunku między jądrami
Model wiązania kowalencyjnego cząsteczka H 2 + Współrzędne elektronu i protonów Orbitale wiążący i antywiążący otrzymane jako kombinacje orbitali atomowych Orbital wiążący duża gęstość ładunku między jądrami
Badanie długości czynników sieciujących metodami symulacji komputerowych
 Badanie długości czynników sieciujących metodami symulacji komputerowych Agnieszka Obarska-Kosińska Prof. dr hab. Bogdan Lesyng Promotorzy: Dr hab. Janusz Bujnicki Zakład Biofizyki, Instytut Fizyki Doświadczalnej,
Badanie długości czynników sieciujących metodami symulacji komputerowych Agnieszka Obarska-Kosińska Prof. dr hab. Bogdan Lesyng Promotorzy: Dr hab. Janusz Bujnicki Zakład Biofizyki, Instytut Fizyki Doświadczalnej,
Metody dokowania ligandów
 Metody dokowania ligandów Strategie projektowania leków Ligand-based drug design nieznana Budowanie modelu miejsc aktywnych liganda (farmakofor) Przeszukiwanie baz danych (screening) Struktura celu molekularnego
Metody dokowania ligandów Strategie projektowania leków Ligand-based drug design nieznana Budowanie modelu miejsc aktywnych liganda (farmakofor) Przeszukiwanie baz danych (screening) Struktura celu molekularnego
Oddziaływanie leków z celami molekularnymi i projektowanie leków
 Oddziaływanie leków z celami molekularnymi i projektowanie leków Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania (biomodellab.eu) Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych
Oddziaływanie leków z celami molekularnymi i projektowanie leków Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania (biomodellab.eu) Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych
- parametry geometryczne badanego związku: współrzędne i typy atomów, ich masy, ładunki, prędkości początkowe itp. (w NAMD plik.
 Avogadro Tworzenie i manipulacja modelami związków chemicznych. W symulacjach dynamiki molekularnej kluczowych elementem jest przygotowanie układu do symulacji tzn. stworzyć pliki wejściowe zawierające
Avogadro Tworzenie i manipulacja modelami związków chemicznych. W symulacjach dynamiki molekularnej kluczowych elementem jest przygotowanie układu do symulacji tzn. stworzyć pliki wejściowe zawierające
Zasady obsadzania poziomów
 Zasady obsadzania poziomów Model atomu Bohra Model kwantowy atomu Fala stojąca Liczby kwantowe -główna liczba kwantowa (n = 1,2,3...) kwantuje energię elektronu (numer orbity) -poboczna liczba kwantowa
Zasady obsadzania poziomów Model atomu Bohra Model kwantowy atomu Fala stojąca Liczby kwantowe -główna liczba kwantowa (n = 1,2,3...) kwantuje energię elektronu (numer orbity) -poboczna liczba kwantowa
Farmakofory. metody QSAR
 Farmakofory metody QSAR Strategie projektowania leków Ligand-based drug design nieznana Budowanie modelu miejsc aktywnych liganda (farmakofor) Przeszukiwanie baz danych (screening) Struktura celu molekularnego
Farmakofory metody QSAR Strategie projektowania leków Ligand-based drug design nieznana Budowanie modelu miejsc aktywnych liganda (farmakofor) Przeszukiwanie baz danych (screening) Struktura celu molekularnego
Bioinformatyka wykład 9
 Bioinformatyka wykład 9 14.XII.21 białkowa bioinformatyka strukturalna krzysztof_pawlowski@sggw.pl 211-1-17 1 Plan wykładu struktury białek dlaczego? struktury białek geometria i fizyka modyfikacje kowalencyjne
Bioinformatyka wykład 9 14.XII.21 białkowa bioinformatyka strukturalna krzysztof_pawlowski@sggw.pl 211-1-17 1 Plan wykładu struktury białek dlaczego? struktury białek geometria i fizyka modyfikacje kowalencyjne
Modelowanie rozpoznawania w układzie lek-receptor
 Modelowanie rozpoznawania w układzie lek-receptor rok model autor 1890 1958 2003 klucza i zamka (lock-and-key) komplementarność indukowanego dopasowania (induced fit) tolerancja zespołu konformacji (ensemble
Modelowanie rozpoznawania w układzie lek-receptor rok model autor 1890 1958 2003 klucza i zamka (lock-and-key) komplementarność indukowanego dopasowania (induced fit) tolerancja zespołu konformacji (ensemble
Strategie projektowania leków
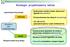 Strategie projektowania leków Ligand-based drug design nieznana Budowanie modelu miejsc aktywnych liganda (farmakofor) Przeszukiwanie baz danych (screening) Struktura celu molekularnego znana 1D i 3D QSAR
Strategie projektowania leków Ligand-based drug design nieznana Budowanie modelu miejsc aktywnych liganda (farmakofor) Przeszukiwanie baz danych (screening) Struktura celu molekularnego znana 1D i 3D QSAR
Recenzja rozprawy doktorskiej mgr. Tomasza Makarewicza, pt.
 Prof. dr hab. Sławomir Filipek, Wydział Chemii, Centrum Nauk Biologiczno-Chemicznych, Uniwersytet Warszawski, ul. Pasteura 1, 02-093 Warszawa Tel. 22-55-26405, E-mail: sfilipek@chem.uw.edu.pl Warszawa,
Prof. dr hab. Sławomir Filipek, Wydział Chemii, Centrum Nauk Biologiczno-Chemicznych, Uniwersytet Warszawski, ul. Pasteura 1, 02-093 Warszawa Tel. 22-55-26405, E-mail: sfilipek@chem.uw.edu.pl Warszawa,
Chemiczne składniki komórek
 Chemiczne składniki komórek Pierwiastki chemiczne w komórkach: - makroelementy (pierwiastki biogenne) H, O, C, N, S, P Ca, Mg, K, Na, Cl >1% suchej masy - mikroelementy Fe, Cu, Mn, Mo, B, Zn, Co, J, F
Chemiczne składniki komórek Pierwiastki chemiczne w komórkach: - makroelementy (pierwiastki biogenne) H, O, C, N, S, P Ca, Mg, K, Na, Cl >1% suchej masy - mikroelementy Fe, Cu, Mn, Mo, B, Zn, Co, J, F
Różne typy wiązań mają ta sama przyczynę: energia powstającej stabilnej cząsteczki jest mniejsza niż sumaryczna energia tworzących ją, oddalonych
 Wiązania atomowe Atomy wieloelektronowe, obsadzanie stanów elektronowych, układ poziomów energii. Przykładowe konfiguracje elektronów, gazy szlachetne, litowce, chlorowce, układ okresowy pierwiastków,
Wiązania atomowe Atomy wieloelektronowe, obsadzanie stanów elektronowych, układ poziomów energii. Przykładowe konfiguracje elektronów, gazy szlachetne, litowce, chlorowce, układ okresowy pierwiastków,
Wykład 3-4. Materiały z wykładów (plik PDF): http://www.biomodellab.eu/lecture/
 Wykład 3-4 Materiały z wykładów (plik PDF): http://www.biomodellab.eu/lecture/ Porównanie zakresów modelowania układów biologicznych Symulacje mezoskopowe, jedno ziarno = kilkanaście aminokwasów, ziarna
Wykład 3-4 Materiały z wykładów (plik PDF): http://www.biomodellab.eu/lecture/ Porównanie zakresów modelowania układów biologicznych Symulacje mezoskopowe, jedno ziarno = kilkanaście aminokwasów, ziarna
Część A wprowadzenie do programu Mercury
 Część A wprowadzenie do programu Mercury Mercury program graficzny do wizualizacji i analizy geometrii cząsteczek i kryształów. Zbiory wejściowe do tego programu o rozszerzeniu res, cif (mol lub pdb) zawierają
Część A wprowadzenie do programu Mercury Mercury program graficzny do wizualizacji i analizy geometrii cząsteczek i kryształów. Zbiory wejściowe do tego programu o rozszerzeniu res, cif (mol lub pdb) zawierają
Zakład Chemii Teoretycznej i Strukturalnej
 Badania struktury i aktywności nietypowego enzymu dekapującego ze Świdrowca nagany Białko TbALPH1 zostało zidentyfikowane jako enzym dekapujący w pasożytniczym pierwotniaku, świdrowcu nagany Trypasonoma
Badania struktury i aktywności nietypowego enzymu dekapującego ze Świdrowca nagany Białko TbALPH1 zostało zidentyfikowane jako enzym dekapujący w pasożytniczym pierwotniaku, świdrowcu nagany Trypasonoma
Wykład z Chemii Ogólnej
 Wykład z Chemii Ogólnej Część 2 Budowa materii: od atomów do układów molekularnych 2.3. WIĄZANIA CHEMICZNE i ODDZIAŁYWANIA Katedra i Zakład Chemii Fizycznej Collegium Medicum w Bydgoszczy Uniwersytet Mikołaja
Wykład z Chemii Ogólnej Część 2 Budowa materii: od atomów do układów molekularnych 2.3. WIĄZANIA CHEMICZNE i ODDZIAŁYWANIA Katedra i Zakład Chemii Fizycznej Collegium Medicum w Bydgoszczy Uniwersytet Mikołaja
Stany skupienia materii
 Stany skupienia materii Ciała stałe - ustalony kształt i objętość - uporządkowanie dalekiego zasięgu - oddziaływania harmoniczne Ciecze -słabo ściśliwe - uporządkowanie bliskiego zasięgu -tworzą powierzchnię
Stany skupienia materii Ciała stałe - ustalony kształt i objętość - uporządkowanie dalekiego zasięgu - oddziaływania harmoniczne Ciecze -słabo ściśliwe - uporządkowanie bliskiego zasięgu -tworzą powierzchnię
Wydział Chemiczny Wybrzeże Wyspiańskiego 27, Wrocław. Prof. dr hab. Ilona Turowska-Tyrk Wrocław, r.
 Wydział Chemiczny Wybrzeże Wyspiańskiego 27, 50-370 Wrocław Prof. dr hab. Ilona Turowska-Tyrk Wrocław, 18.01.2016 r. Recenzja rozprawy doktorskiej mgr Pauliny Klimentowskiej pt. Krystalochemia wybranych
Wydział Chemiczny Wybrzeże Wyspiańskiego 27, 50-370 Wrocław Prof. dr hab. Ilona Turowska-Tyrk Wrocław, 18.01.2016 r. Recenzja rozprawy doktorskiej mgr Pauliny Klimentowskiej pt. Krystalochemia wybranych
października 2013: Elementarz biologii molekularnej. Wykład nr 2 BIOINFORMATYKA rok II
 10 października 2013: Elementarz biologii molekularnej www.bioalgorithms.info Wykład nr 2 BIOINFORMATYKA rok II Komórka: strukturalna i funkcjonalne jednostka organizmu żywego Jądro komórkowe: chroniona
10 października 2013: Elementarz biologii molekularnej www.bioalgorithms.info Wykład nr 2 BIOINFORMATYKA rok II Komórka: strukturalna i funkcjonalne jednostka organizmu żywego Jądro komórkowe: chroniona
Przegląd budowy i funkcji białek
 Przegląd budowy i funkcji białek Co piszą o białkach? Wyraz wprowadzony przez Jönsa J. Berzeliusa w 1883 r. w celu podkreślenia znaczenia tej grupy związków. Termin pochodzi od greckiego słowa proteios,
Przegląd budowy i funkcji białek Co piszą o białkach? Wyraz wprowadzony przez Jönsa J. Berzeliusa w 1883 r. w celu podkreślenia znaczenia tej grupy związków. Termin pochodzi od greckiego słowa proteios,
WIĄZANIA. Co sprawia, że ciała stałe istnieją i są stabilne? PRZYCIĄGANIE ODPYCHANIE
 WIĄZANIA Co sprawia, że ciała stałe istnieją i są stabilne? PRZYCIĄGANIE ODPYCHANIE Przyciąganie Wynika z elektrostatycznego oddziaływania między elektronami a dodatnimi jądrami atomowymi. Może to być
WIĄZANIA Co sprawia, że ciała stałe istnieją i są stabilne? PRZYCIĄGANIE ODPYCHANIE Przyciąganie Wynika z elektrostatycznego oddziaływania między elektronami a dodatnimi jądrami atomowymi. Może to być
Lek od pomysłu do wdrożenia
 Lek od pomysłu do wdrożenia Lek od pomysłu do wdrożenia KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU
Lek od pomysłu do wdrożenia Lek od pomysłu do wdrożenia KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU KRÓTKA HISTORIA LEKU
Komputerowe wspomaganie projektowanie leków
 Komputerowe wspomaganie projektowanie leków wykład VII Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Komputerowe wspomaganie projektowanie leków wykład VII Prof. dr hab. Sławomir Filipek Grupa BIOmodelowania Uniwersytet Warszawski, Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Cent-III www.biomodellab.eu
Uniwersytet Śląski w Katowicach str. 1 Wydział
 Uniwersytet Śląski w Katowicach str. 1 Kierunek i poziom studiów: Chemia II stopień Sylabus modułu: : Przedmiot C związany ze specjalnością (0310-CH-S2-003) Nazwa wariantu modułu (opcjonalnie): Chemoinformatyka
Uniwersytet Śląski w Katowicach str. 1 Kierunek i poziom studiów: Chemia II stopień Sylabus modułu: : Przedmiot C związany ze specjalnością (0310-CH-S2-003) Nazwa wariantu modułu (opcjonalnie): Chemoinformatyka
Informacje. W sprawach organizacyjnych Slajdy z wykładów
 Biochemia Informacje W sprawach organizacyjnych malgorzata.dutkiewicz@wum.edu.pl Slajdy z wykładów www.takao.pl W sprawach merytorycznych Takao Ishikawa (takao@biol.uw.edu.pl) Kiedy? Co? Kto? 24 lutego
Biochemia Informacje W sprawach organizacyjnych malgorzata.dutkiewicz@wum.edu.pl Slajdy z wykładów www.takao.pl W sprawach merytorycznych Takao Ishikawa (takao@biol.uw.edu.pl) Kiedy? Co? Kto? 24 lutego
Atomy wieloelektronowe
 Wiązania atomowe Atomy wieloelektronowe, obsadzanie stanów elektronowych, układ poziomów energii. Przykładowe konfiguracje elektronów, gazy szlachetne, litowce, chlorowce, układ okresowy pierwiastków,
Wiązania atomowe Atomy wieloelektronowe, obsadzanie stanów elektronowych, układ poziomów energii. Przykładowe konfiguracje elektronów, gazy szlachetne, litowce, chlorowce, układ okresowy pierwiastków,
( ) ρ ( ) ( ) ( ) ( ) ( ) Rozkład ładunku i momenty dipolowe cząsteczek. woda H 2 O. aceton (CH 3 ) 2 CO
 Rozkład ładunku i momenty dipolowe cząsteczek W fizyce klasycznej moment dipolowy układu ładunków oblicza się tak: i i i ( ) ρ ( ) µ = q r lub µ = ρ x, y, z r dxdydz = x, y, z r dv W teorii kwantowej moment
Rozkład ładunku i momenty dipolowe cząsteczek W fizyce klasycznej moment dipolowy układu ładunków oblicza się tak: i i i ( ) ρ ( ) µ = q r lub µ = ρ x, y, z r dxdydz = x, y, z r dv W teorii kwantowej moment
Bioinformatyka wykład 3.I.2008
 Bioinformatyka wykład 3.I.2008 Białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2008-01-03 1 Plan wykładu analiza i porównywanie struktur białek. doświadczalne metody badania struktur
Bioinformatyka wykład 3.I.2008 Białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2008-01-03 1 Plan wykładu analiza i porównywanie struktur białek. doświadczalne metody badania struktur
Dotyczy to zarówno istniejących już związków, jak i związków, których jeszcze dotąd nie otrzymano.
 Chemia teoretyczna to dział chemii zaliczany do chemii fizycznej, zajmujący się zagadnieniami związanymi z wiedzą chemiczną od strony teoretycznej, tj. bez wykonywania eksperymentów na stole laboratoryjnym.
Chemia teoretyczna to dział chemii zaliczany do chemii fizycznej, zajmujący się zagadnieniami związanymi z wiedzą chemiczną od strony teoretycznej, tj. bez wykonywania eksperymentów na stole laboratoryjnym.
Podstawy projektowania leków wykład 11
 Podstawy projektowania leków wykład 11 Łukasz Berlicki QSAR i 3D QSAR 1964, C. Hansch i T. Fujita: QSAR 1984, P. Andrews: wkład grup funkcyjnych do oddziaływania z celem molekularnym 1985, P. Goodford:
Podstawy projektowania leków wykład 11 Łukasz Berlicki QSAR i 3D QSAR 1964, C. Hansch i T. Fujita: QSAR 1984, P. Andrews: wkład grup funkcyjnych do oddziaływania z celem molekularnym 1985, P. Goodford:
Atomy wieloelektronowe i cząsteczki
 Atomy wieloelektronowe i cząsteczki 1 Atomy wieloelektronowe Wodór ma liczbę atomową Z=1 i jest prostym atomem. Zawiera tylko jeden elektron i jeden proton stąd potencjał opisuje oddziaływanie kulombowskie
Atomy wieloelektronowe i cząsteczki 1 Atomy wieloelektronowe Wodór ma liczbę atomową Z=1 i jest prostym atomem. Zawiera tylko jeden elektron i jeden proton stąd potencjał opisuje oddziaływanie kulombowskie
Dokowanie molekularne. Andrzej Bąk Instytut Chemii UŚ chemoinformatyka wykład 1
 Dokowanie molekularne Andrzej Bąk Instytut Chemii UŚ chemoinformatyka wykład 1 Zarys Oddziaływanie ligand-receptor Modelowanie struktury receptora Reprezentacja makromolekuł Opis energii oddziaływań ligand-receptor
Dokowanie molekularne Andrzej Bąk Instytut Chemii UŚ chemoinformatyka wykład 1 Zarys Oddziaływanie ligand-receptor Modelowanie struktury receptora Reprezentacja makromolekuł Opis energii oddziaływań ligand-receptor
Żwirki i Wigury 93, Warszawa TEL.: , FAX: , E- MAIL: Dr hab. Joanna T
 Żwirki i Wigury 93, 02-089 Warszawa TEL.: + 48 22 55 40 800, FAX: +48 22 55 40 801, E- MAIL: sekretariat@uw.edu.pl www.cent.uw.edu.pl Dr hab. Joanna Trylska, prof. UW tel. (22) 5540843 e- mail: joanna@cent.uw.edu.pl
Żwirki i Wigury 93, 02-089 Warszawa TEL.: + 48 22 55 40 800, FAX: +48 22 55 40 801, E- MAIL: sekretariat@uw.edu.pl www.cent.uw.edu.pl Dr hab. Joanna Trylska, prof. UW tel. (22) 5540843 e- mail: joanna@cent.uw.edu.pl
ogromna liczba małych cząsteczek, doskonale elastycznych, poruszających się we wszystkich kierunkach, tory prostoliniowe, kierunek ruchu zmienia się
 CHEMIA NIEORGANICZNA Dr hab. Andrzej Kotarba Zakład Chemii Nieorganicznej Wydział Chemii I pietro p. 138 WYKŁAD - STAN GAZOWY i CHEMIA GAZÓW kinetyczna teoria gazów ogromna liczba małych cząsteczek, doskonale
CHEMIA NIEORGANICZNA Dr hab. Andrzej Kotarba Zakład Chemii Nieorganicznej Wydział Chemii I pietro p. 138 WYKŁAD - STAN GAZOWY i CHEMIA GAZÓW kinetyczna teoria gazów ogromna liczba małych cząsteczek, doskonale
O C E N A rozprawy doktorskiej mgr Pauliny Fortuny pt. Projektowanie i synteza inhibitorów oddziaływania białko-białko dla układów
 Wrocław, 2017-05-18 Dr hab. Piotr Stefanowicz tel. 71 375 7213 piotr.stefanowicz@chem.uni.wroc.pl O C E N A rozprawy doktorskiej mgr Pauliny Fortuny pt. Projektowanie i synteza inhibitorów oddziaływania
Wrocław, 2017-05-18 Dr hab. Piotr Stefanowicz tel. 71 375 7213 piotr.stefanowicz@chem.uni.wroc.pl O C E N A rozprawy doktorskiej mgr Pauliny Fortuny pt. Projektowanie i synteza inhibitorów oddziaływania
Podział ciał stałych ze względu na strukturę atomowo-cząsteczkową
 Podział ciał stałych ze względu na strukturę atomowo-cząsteczkową Kryształy Atomy w krysztale ułożone są w pewien powtarzający się regularny wzór zwany siecią krystaliczną. Struktura kryształu NaCl Polikryształy
Podział ciał stałych ze względu na strukturę atomowo-cząsteczkową Kryształy Atomy w krysztale ułożone są w pewien powtarzający się regularny wzór zwany siecią krystaliczną. Struktura kryształu NaCl Polikryształy
Zastosowanie banku asferycznych pseudoatomów w badaniach oddziaływań elektrostatycznych palców cynkowych z DNA
 Prof. dr hab. Sławomir Filipek, Wydział Chemii, Centrum Nauk Biologiczno-Chemicznych, Uniwersytet Warszawski, ul. Pasteura 1, 02-093 Warszawa Tel. 22-55-26405, E-mail: sfilipek@chem.uw.edu.pl Warszawa,
Prof. dr hab. Sławomir Filipek, Wydział Chemii, Centrum Nauk Biologiczno-Chemicznych, Uniwersytet Warszawski, ul. Pasteura 1, 02-093 Warszawa Tel. 22-55-26405, E-mail: sfilipek@chem.uw.edu.pl Warszawa,
prof. dr hab. Krzysztof Lewiński Kraków, Wydział Chemii Uniwersytetu Jagiellońskiego
 prof. dr hab. Krzysztof Lewiński Kraków, 12.06.2017 Wydział Chemii Uniwersytetu Jagiellońskiego Recenzja rozprawy doktorskiej mgr. Pawła Strzelczyka "Badania strukturalne kompleksów awidyny z ligandami"
prof. dr hab. Krzysztof Lewiński Kraków, 12.06.2017 Wydział Chemii Uniwersytetu Jagiellońskiego Recenzja rozprawy doktorskiej mgr. Pawła Strzelczyka "Badania strukturalne kompleksów awidyny z ligandami"
1. Kryształy jonowe omówić oddziaływania w kryształach jonowych oraz typy struktur jonowych.
 Tematy opisowe 1. Kryształy jonowe omówić oddziaływania w kryształach jonowych oraz typy struktur jonowych. 2. Dlaczego do kadłubów statków, doków, falochronów i filarów mostów przymocowuje się płyty z
Tematy opisowe 1. Kryształy jonowe omówić oddziaływania w kryształach jonowych oraz typy struktur jonowych. 2. Dlaczego do kadłubów statków, doków, falochronów i filarów mostów przymocowuje się płyty z
Wiązania chemiczne. Związek klasyfikacji ciał krystalicznych z charakterem wiązań atomowych. 5 typów wiązań
 Wiązania chemiczne Związek klasyfikacji ciał krystalicznych z charakterem wiązań atomowych 5 typów wiązań wodorowe A - H - A, jonowe ( np. KCl ) molekularne (pomiędzy atomami gazów szlachetnych i małymi
Wiązania chemiczne Związek klasyfikacji ciał krystalicznych z charakterem wiązań atomowych 5 typów wiązań wodorowe A - H - A, jonowe ( np. KCl ) molekularne (pomiędzy atomami gazów szlachetnych i małymi
Autoreferat rozprawy doktorskiej pt.: ROZSZERZENIE BANKU ASFERYCZNYCH PSEUDOATOMÓW W KIERUNKU
 Warszawa, 2.10.2012 Katarzyna Jarzembska Pracownia Krystalochemii Wydział Chemii Uniwersytet Warszawski Autoreferat rozprawy doktorskiej pt.: ROZSZERZENIE BANKU ASFERYCZNYCH PSEUDOATOMÓW W KIERUNKU KWASÓW
Warszawa, 2.10.2012 Katarzyna Jarzembska Pracownia Krystalochemii Wydział Chemii Uniwersytet Warszawski Autoreferat rozprawy doktorskiej pt.: ROZSZERZENIE BANKU ASFERYCZNYCH PSEUDOATOMÓW W KIERUNKU KWASÓW
Cz. I Materiał powtórzeniowy do sprawdzianu dla klas II LO - Wiązania chemiczne + przykładowe zadania i proponowane rozwiązania
 Cz. I Materiał powtórzeniowy do sprawdzianu dla klas II LO - Wiązania chemiczne + przykładowe zadania i proponowane rozwiązania I. Elektroujemność pierwiastków i elektronowa teoria wiązań Lewisa-Kossela
Cz. I Materiał powtórzeniowy do sprawdzianu dla klas II LO - Wiązania chemiczne + przykładowe zadania i proponowane rozwiązania I. Elektroujemność pierwiastków i elektronowa teoria wiązań Lewisa-Kossela
Przewidywanie struktur białek
 Łukasz Ołdziejewski Wydział Chemii UW Przewidywanie struktur białek czyli droga do projektowania indywidualnych leków Sprawozdanie studenckie 2007/2008 1 Indywidualność jednostki KaŜdy człowiek jest indywidualnym
Łukasz Ołdziejewski Wydział Chemii UW Przewidywanie struktur białek czyli droga do projektowania indywidualnych leków Sprawozdanie studenckie 2007/2008 1 Indywidualność jednostki KaŜdy człowiek jest indywidualnym
Bioinformatyka wykład 8, 27.XI.2012
 Bioinformatyka wykład 8, 27.XI.2012 białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2013-01-21 1 Plan wykładu regiony nieuporządkowane sposoby przedstawienia struktur białkowych powierzchnia
Bioinformatyka wykład 8, 27.XI.2012 białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2013-01-21 1 Plan wykładu regiony nieuporządkowane sposoby przedstawienia struktur białkowych powierzchnia
Spektrometria mas (1)
 pracował: Wojciech Augustyniak Spektrometria mas (1) Spektrometr masowy ma źródło jonów, które jonizuje próbkę Jony wędrują w polu elektromagnetycznym do detektora Metody jonizacji: - elektronowa (EI)
pracował: Wojciech Augustyniak Spektrometria mas (1) Spektrometr masowy ma źródło jonów, które jonizuje próbkę Jony wędrują w polu elektromagnetycznym do detektora Metody jonizacji: - elektronowa (EI)
Podstawowe prawa opisujące właściwości gazów zostały wyprowadzone dla gazu modelowego, nazywanego gazem doskonałym (idealnym).
 Spis treści 1 Stan gazowy 2 Gaz doskonały 21 Definicja mikroskopowa 22 Definicja makroskopowa (termodynamiczna) 3 Prawa gazowe 31 Prawo Boyle a-mariotte a 32 Prawo Gay-Lussaca 33 Prawo Charlesa 34 Prawo
Spis treści 1 Stan gazowy 2 Gaz doskonały 21 Definicja mikroskopowa 22 Definicja makroskopowa (termodynamiczna) 3 Prawa gazowe 31 Prawo Boyle a-mariotte a 32 Prawo Gay-Lussaca 33 Prawo Charlesa 34 Prawo
prof. dr hab. Krzysztof Lewiński Kraków, Wydział Chemii Uniwersytetu Jagiellońskiego
 prof. dr hab. Krzysztof Lewiński Kraków, 8.08.2014 Wydział Chemii Uniwersytetu Jagiellońskiego Recenzja rozprawy doktorskiej mgr. Przemysława Porębskiego "Improvement of model building and refinement of
prof. dr hab. Krzysztof Lewiński Kraków, 8.08.2014 Wydział Chemii Uniwersytetu Jagiellońskiego Recenzja rozprawy doktorskiej mgr. Przemysława Porębskiego "Improvement of model building and refinement of
cz. VI leki przeciwwirusowe i przeciwnowotworowe
 Oddziaływanie leków z celami molekularnymi cz. VI leki przeciwwirusowe i przeciwnowotworowe Prof. dr hab. Sławomir Filipek Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Uniwersytet Warszawski
Oddziaływanie leków z celami molekularnymi cz. VI leki przeciwwirusowe i przeciwnowotworowe Prof. dr hab. Sławomir Filipek Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Uniwersytet Warszawski
Bioinformatyka wykład 10
 Bioinformatyka wykład 10 21.XII.2010 białkowa bioinformatyka strukturalna, c.d. krzysztof_pawlowski@sggw.pl 2011-01-17 1 Regiony nieuporządkowane disordered regions trudna definicja trudne do przewidzenia
Bioinformatyka wykład 10 21.XII.2010 białkowa bioinformatyka strukturalna, c.d. krzysztof_pawlowski@sggw.pl 2011-01-17 1 Regiony nieuporządkowane disordered regions trudna definicja trudne do przewidzenia
Modelowanie molekularne w projektowaniu leków
 Modelowanie molekularne w projektowaniu leków Wykład I Wstęp (o czym będę a o czym nie będę mówić) Opis układu Solwent (woda z rozpuszczonymi jonami i innymi substancjami) Ligand (potencjalny lek) Makromolekuła
Modelowanie molekularne w projektowaniu leków Wykład I Wstęp (o czym będę a o czym nie będę mówić) Opis układu Solwent (woda z rozpuszczonymi jonami i innymi substancjami) Ligand (potencjalny lek) Makromolekuła
protos (gr.) pierwszy protein/proteins (ang.)
 Białka 1 protos (gr.) pierwszy protein/proteins (ang.) cząsteczki życia materiał budulcowy materii ożywionej oraz wirusów wielkocząsteczkowe biopolimery o masie od kilku tysięcy do kilku milionów jednostek
Białka 1 protos (gr.) pierwszy protein/proteins (ang.) cząsteczki życia materiał budulcowy materii ożywionej oraz wirusów wielkocząsteczkowe biopolimery o masie od kilku tysięcy do kilku milionów jednostek
Cz. I Materiał powtórzeniowy do sprawdzianu dla klas I LO - Wiązania chemiczne + przykładowe zadania i proponowane rozwiązania
 Cz. I Materiał powtórzeniowy do sprawdzianu dla klas I LO - Wiązania chemiczne + przykładowe zadania i proponowane rozwiązania I. Elektroujemność pierwiastków i elektronowa teoria wiązań Lewisa-Kossela
Cz. I Materiał powtórzeniowy do sprawdzianu dla klas I LO - Wiązania chemiczne + przykładowe zadania i proponowane rozwiązania I. Elektroujemność pierwiastków i elektronowa teoria wiązań Lewisa-Kossela
Część A wprowadzenie do programu Mercury
 Część A wprowadzenie do programu Mercury Mercury program graficzny do wizualizacji i analizy geometrii cząsteczek i kryształów. Program ten jest stale uzupełniany i modyfikowany, jednak jego podstawowe
Część A wprowadzenie do programu Mercury Mercury program graficzny do wizualizacji i analizy geometrii cząsteczek i kryształów. Program ten jest stale uzupełniany i modyfikowany, jednak jego podstawowe
Wiązania kowalencyjne
 Wiązania kowalencyjne (pierw. o dużej E + pierw. o dużej E), E < 1,8 TERIE WIĄZANIA KWALENCYJNEG Teoria hybrydyzacji orbitali atomowych Teoria orbitali molekularnych Teoria pola ligandów YBRYDYZACJA RBITALI
Wiązania kowalencyjne (pierw. o dużej E + pierw. o dużej E), E < 1,8 TERIE WIĄZANIA KWALENCYJNEG Teoria hybrydyzacji orbitali atomowych Teoria orbitali molekularnych Teoria pola ligandów YBRYDYZACJA RBITALI
SPECJALNE TECHNIKI ROZDZIELANIA W BIOTECHNOLOGII. Laboratorium nr1 CHROMATOGRAFIA ODDZIAŁYWAŃ HYDROFOBOWYCH
 SPECJALNE TECHNIKI ROZDZIELANIA W BIOTECHNOLOGII Laboratorium nr1 CHROMATOGRAFIA ODDZIAŁYWAŃ HYDROFOBOWYCH Opracowała: dr inż. Renata Muca I. WPROWADZENIE TEORETYCZNE Chromatografia oddziaływań hydrofobowych
SPECJALNE TECHNIKI ROZDZIELANIA W BIOTECHNOLOGII Laboratorium nr1 CHROMATOGRAFIA ODDZIAŁYWAŃ HYDROFOBOWYCH Opracowała: dr inż. Renata Muca I. WPROWADZENIE TEORETYCZNE Chromatografia oddziaływań hydrofobowych
Fizyka i Ŝycie cz. I impresje molekularne. śycie z molekularnego punktu widzenia. Cząsteczka chemiczna
 Fizyka i Ŝycie cz. I impresje molekularne Poznawanie zjawiska Ŝycia to fascynujące miejsce spotkań wielu dziedzin nauki: biologii biochemii chemii fizyki matematyki informatyki oraz 7 stycznia 2010 Maciej
Fizyka i Ŝycie cz. I impresje molekularne Poznawanie zjawiska Ŝycia to fascynujące miejsce spotkań wielu dziedzin nauki: biologii biochemii chemii fizyki matematyki informatyki oraz 7 stycznia 2010 Maciej
Komputerowe wspomaganie projektowania leków - QSAR
 Komputerowe wspomaganie projektowania leków - QSAR JAKIE OBIEKTY SĄ INTERESUJĄCE? Długość pojazdu wynosi 2 nm, szerokość 2nm. Pojazd porusza się na kołach złożonych z 60 atomów czystego węgla, uformowanych
Komputerowe wspomaganie projektowania leków - QSAR JAKIE OBIEKTY SĄ INTERESUJĄCE? Długość pojazdu wynosi 2 nm, szerokość 2nm. Pojazd porusza się na kołach złożonych z 60 atomów czystego węgla, uformowanych
Enzymy katalizatory biologiczne
 Enzymy katalizatory biologiczne Kataliza zjawisko polegające na obniżeniu energii aktywacji reakcji i zwiększeniu szybkości reakcji chemicznej i/lub skierowaniu reakcji na jedną z termodynamicznie możliwych
Enzymy katalizatory biologiczne Kataliza zjawisko polegające na obniżeniu energii aktywacji reakcji i zwiększeniu szybkości reakcji chemicznej i/lub skierowaniu reakcji na jedną z termodynamicznie możliwych
EWA PIĘTA. Streszczenie pracy doktorskiej
 EWA PIĘTA Spektroskopowa analiza struktur molekularnych i procesu adsorpcji fosfinowych pochodnych pirydyny, potencjalnych inhibitorów aminopeptydazy N Streszczenie pracy doktorskiej wykonanej na Wydziale
EWA PIĘTA Spektroskopowa analiza struktur molekularnych i procesu adsorpcji fosfinowych pochodnych pirydyny, potencjalnych inhibitorów aminopeptydazy N Streszczenie pracy doktorskiej wykonanej na Wydziale
Optymalizacja optymalizacji
 7 maja 2008 Wstęp Optymalizacja lokalna Optymalizacja globalna Algorytmy genetyczne Badane czasteczki Wykorzystane oprogramowanie (Algorytm genetyczny) 2 Sieć neuronowa Pochodne met-enkefaliny Optymalizacja
7 maja 2008 Wstęp Optymalizacja lokalna Optymalizacja globalna Algorytmy genetyczne Badane czasteczki Wykorzystane oprogramowanie (Algorytm genetyczny) 2 Sieć neuronowa Pochodne met-enkefaliny Optymalizacja
Konkurs chemiczny gimnazjum. 2018/2019. Etap wojewódzki. MODEL ODPOWIEDZI I SCHEMAT PUNKTOWANIA ZADAŃ III ETAPU KONKURSU CHEMICZNEGO
 MODEL ODPOWIEDZI I SCHEMAT PUNKTOWANIA ZADAŃ III ETAPU KONKURSU CHEMICZNEGO Zadania zamknięte: 1 pkt poprawnie zaznaczona odpowiedź 0 pkt błędnie zaznaczona odpowiedź Zad. 1 2 3 4 5 Odp. D D C A B Zadania
MODEL ODPOWIEDZI I SCHEMAT PUNKTOWANIA ZADAŃ III ETAPU KONKURSU CHEMICZNEGO Zadania zamknięte: 1 pkt poprawnie zaznaczona odpowiedź 0 pkt błędnie zaznaczona odpowiedź Zad. 1 2 3 4 5 Odp. D D C A B Zadania
Struktura i funkcja białek (I mgr)
 Struktura i funkcja białek (I mgr) Dr Filip Jeleń fj@protein.pl http://www.protein.pl/ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer Biochemia Carl Branden, John Tooze Introduction to Protein Structure
Struktura i funkcja białek (I mgr) Dr Filip Jeleń fj@protein.pl http://www.protein.pl/ Jeremy M. Berg, John L. Tymoczko, Lubert Stryer Biochemia Carl Branden, John Tooze Introduction to Protein Structure
Bioinformatyka II Modelowanie struktury białek
 Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania dla każdego z podanych przypadków? Dlaczego? Struktura krystaliczną czy NMR (to samo białko,
Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania dla każdego z podanych przypadków? Dlaczego? Struktura krystaliczną czy NMR (to samo białko,
WIĄZANIA. Co sprawia, że ciała stałe istnieją i są stabilne? PRZYCIĄGANIE ODPYCHANIE
 WIĄZANIA Co sprawia, że ciała stałe istnieją i są stabilne? PRZYCIĄGANIE ODPYCHANIE 1 Przyciąganie Wynika z elektrostatycznego oddziaływania między elektronami a dodatnimi jądrami atomowymi. Może to być
WIĄZANIA Co sprawia, że ciała stałe istnieją i są stabilne? PRZYCIĄGANIE ODPYCHANIE 1 Przyciąganie Wynika z elektrostatycznego oddziaływania między elektronami a dodatnimi jądrami atomowymi. Może to być
Bioinformatyka wykład 11, 11.I.2011 Białkowa bioinformatyka strukturalna c.d.
 Bioinformatyka wykład 11, 11.I.2011 Białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 11.01.11 1 Dopasowanie strukturalne (alignment) odległość: d ij = (x i -x J ) 2 + (y i -y J ) 2
Bioinformatyka wykład 11, 11.I.2011 Białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 11.01.11 1 Dopasowanie strukturalne (alignment) odległość: d ij = (x i -x J ) 2 + (y i -y J ) 2
Opracowała: mgr inż. Ewelina Nowak
 Materiały dydaktyczne na zajęcia wyrównawcze z chemii dla studentów pierwszego roku kierunku zamawianego Inżynieria Środowiska w ramach projektu Era inżyniera pewna lokata na przyszłość Opracowała: mgr
Materiały dydaktyczne na zajęcia wyrównawcze z chemii dla studentów pierwszego roku kierunku zamawianego Inżynieria Środowiska w ramach projektu Era inżyniera pewna lokata na przyszłość Opracowała: mgr
Leki przeciwwirusowe
 Leki przeciwwirusowe Budowa i działanie wirusów Wirusy RNA: grypa, rotawirus, SARS, wir. zap. wątroby A i C, Ebola, HIV Wirusy DNA: - ospa prawdziwa (smallpox), - ospa wietrzna (chickenpox), - opryszczka
Leki przeciwwirusowe Budowa i działanie wirusów Wirusy RNA: grypa, rotawirus, SARS, wir. zap. wątroby A i C, Ebola, HIV Wirusy DNA: - ospa prawdziwa (smallpox), - ospa wietrzna (chickenpox), - opryszczka
Warszawa, 25 sierpnia 2016
 ul. S. Banacha 2c, 02-097 Warszawa TEL.: + 48 22 55 43 600, FAX: +48 22 55 43 606, E-MAIL: sekretariat@cent.uw.edu.pl www.cent.uw.edu.pl Warszawa, 25 sierpnia 2016 Dr hab. Joanna Trylska, prof. UW e-mail:
ul. S. Banacha 2c, 02-097 Warszawa TEL.: + 48 22 55 43 600, FAX: +48 22 55 43 606, E-MAIL: sekretariat@cent.uw.edu.pl www.cent.uw.edu.pl Warszawa, 25 sierpnia 2016 Dr hab. Joanna Trylska, prof. UW e-mail:
Fizyka 2. Janusz Andrzejewski
 Fizyka 2 wykład 13 Janusz Andrzejewski Scaledlugości Janusz Andrzejewski 2 Scaledługości Simple molecules
Fizyka 2 wykład 13 Janusz Andrzejewski Scaledlugości Janusz Andrzejewski 2 Scaledługości Simple molecules
Kuratorium Oświaty w Lublinie
 Kuratorium Oświaty w Lublinie KOD UCZNIA ZESTAW ZADAŃ KONKURSOWYCH Z CHEMII DLA UCZNIÓW GIMNAZJÓW ROK SZKOLNY 2015/2016 ETAP WOJEWÓDZKI Instrukcja dla ucznia 1. Zestaw konkursowy zawiera 12 zadań. 2. Przed
Kuratorium Oświaty w Lublinie KOD UCZNIA ZESTAW ZADAŃ KONKURSOWYCH Z CHEMII DLA UCZNIÓW GIMNAZJÓW ROK SZKOLNY 2015/2016 ETAP WOJEWÓDZKI Instrukcja dla ucznia 1. Zestaw konkursowy zawiera 12 zadań. 2. Przed
Bioinformatyka wykład 10.I.2008
 Bioinformatyka wykład 10.I.2008 Przewidywanie struktur białek krzysztof_pawlowski@sggw.pl 2008-01-10 1 Plan wykładu pole siłowe - opis energetyczny struktur białka proces zwijania się białek przewidywanie
Bioinformatyka wykład 10.I.2008 Przewidywanie struktur białek krzysztof_pawlowski@sggw.pl 2008-01-10 1 Plan wykładu pole siłowe - opis energetyczny struktur białka proces zwijania się białek przewidywanie
Bioinformatyka II Modelowanie struktury białek
 Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania? Dlaczego? Struktura krystaliczną czy NMR (to samo białko, ta sama rozdzielczość)? Strukturę
Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania? Dlaczego? Struktura krystaliczną czy NMR (to samo białko, ta sama rozdzielczość)? Strukturę
Molecular dynamics investigation of the structure-function relationships in proteins with examples
 Molecular dynamics investigation of the structure-function relationships in proteins with examples from Hsp70 molecular chaperones, αa-crystallin, and sericin Badanie metodą dynamiki molekularnej zależności
Molecular dynamics investigation of the structure-function relationships in proteins with examples from Hsp70 molecular chaperones, αa-crystallin, and sericin Badanie metodą dynamiki molekularnej zależności
Rysunki pochodzą z książki Chemia medyczna, G.L. Patrick, WNT. Odwołanie do wykładu z STL
 Rysunki pochodzą z książki Chemia medyczna, G.L. Patrick, WNT Odwołanie do wykładu z STL Izolacja z surowców naturalnych jak np. rośliny, tkanki zwierzęce Lek naturalny Lek biotechnologiczny Lek syntetyczny
Rysunki pochodzą z książki Chemia medyczna, G.L. Patrick, WNT Odwołanie do wykładu z STL Izolacja z surowców naturalnych jak np. rośliny, tkanki zwierzęce Lek naturalny Lek biotechnologiczny Lek syntetyczny
Przegląd budowy i funkcji białek - od enzymów do prionów -
 Przegląd budowy i funkcji białek - od enzymów do prionów - Zakład Biologii Molekularnej Instytut Biochemii, Wydział Biologii UW Takao Ishikawa Kontakt Imię i nazwisko: Takao Ishikawa Mail: takao@biol.uw.edu.pl
Przegląd budowy i funkcji białek - od enzymów do prionów - Zakład Biologii Molekularnej Instytut Biochemii, Wydział Biologii UW Takao Ishikawa Kontakt Imię i nazwisko: Takao Ishikawa Mail: takao@biol.uw.edu.pl
Elementy chemii obliczeniowej i bioinformatyki Zagadnienia na egzamin
 Elementy chemii obliczeniowej i bioinformatyki Zagadnienia na egzamin 1. Zapisz konfigurację elektronową dla atomu helu (dwa elektrony) i wyjaśnij, dlaczego cząsteczka wodoru jest stabilna, a cząsteczka
Elementy chemii obliczeniowej i bioinformatyki Zagadnienia na egzamin 1. Zapisz konfigurację elektronową dla atomu helu (dwa elektrony) i wyjaśnij, dlaczego cząsteczka wodoru jest stabilna, a cząsteczka
Konkurs chemiczny szkoła podstawowa. 2018/2019. Etap wojewódzki. MODEL ODPOWIEDZI I SCHEMAT PUNKTOWANIA ZADAŃ III ETAPU KONKURSU CHEMICZNEGO
 MODEL ODPOWIEDZI I SCHEMAT PUNKTOWANIA ZADAŃ III ETAPU KONKURSU CHEMICZNEGO Zadania zamknięte: 1 pkt poprawnie zaznaczona odpowiedź 0 pkt błędnie zaznaczona odpowiedź Zad. 1 2 3 4 5 6 7 Odp. D C C B C
MODEL ODPOWIEDZI I SCHEMAT PUNKTOWANIA ZADAŃ III ETAPU KONKURSU CHEMICZNEGO Zadania zamknięte: 1 pkt poprawnie zaznaczona odpowiedź 0 pkt błędnie zaznaczona odpowiedź Zad. 1 2 3 4 5 6 7 Odp. D C C B C
Struktura biomakromolekuł chemia biologiczna III rok
 truktura biomakromolekuł chemia biologiczna III rok jak są zbudowane białka? dlaczego białka są tak zbudowane? co z tego wynika? 508 13 604 liczba struktur dostępnych w Protein Data Bank wynosi aktualnie
truktura biomakromolekuł chemia biologiczna III rok jak są zbudowane białka? dlaczego białka są tak zbudowane? co z tego wynika? 508 13 604 liczba struktur dostępnych w Protein Data Bank wynosi aktualnie
Konformacje cykloheksanu
 I O T E C N O L CEMI O G ORGNICZN I Konformacje cykloheksanu Konformacje cykloheksanu sposób rysowania pierścienia sześcioczłonowego Dekalina MONOPODSTWIONE pochodne cykloheksanu Konformacja krzesłowa
I O T E C N O L CEMI O G ORGNICZN I Konformacje cykloheksanu Konformacje cykloheksanu sposób rysowania pierścienia sześcioczłonowego Dekalina MONOPODSTWIONE pochodne cykloheksanu Konformacja krzesłowa
Bioinformatyka wykład 8
 Bioinformatyka wykład 8 2.XII.2008 białkowa bioinformatyka strukturalna krzysztof_pawlowski@sggw.pl 2009-01-08 1 Lecture outline, Dec. 6th protein structures why? protein structures geometry and physics
Bioinformatyka wykład 8 2.XII.2008 białkowa bioinformatyka strukturalna krzysztof_pawlowski@sggw.pl 2009-01-08 1 Lecture outline, Dec. 6th protein structures why? protein structures geometry and physics
cz. III leki przeciwzapalne
 Oddziaływanie leków z celami molekularnymi cz. III leki przeciwzapalne Prof. dr hab. Sławomir Filipek Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Uniwersytet Warszawski 1 Leki przeciwzapalne
Oddziaływanie leków z celami molekularnymi cz. III leki przeciwzapalne Prof. dr hab. Sławomir Filipek Wydział Chemii oraz Centrum Nauk Biologiczno-Chemicznych Uniwersytet Warszawski 1 Leki przeciwzapalne
Repetytorium z wybranych zagadnień z chemii
 Repetytorium z wybranych zagadnień z chemii Mol jest to liczebność materii występująca, gdy liczba cząstek (elementów) układu jest równa liczbie atomów zawartych w masie 12 g węgla 12 C (równa liczbie
Repetytorium z wybranych zagadnień z chemii Mol jest to liczebność materii występująca, gdy liczba cząstek (elementów) układu jest równa liczbie atomów zawartych w masie 12 g węgla 12 C (równa liczbie
Modelowanie białek ab initio / de novo
 Modelowanie białek ab initio / de novo Słowniczek de novo - od początku, na nowo ab initio - od początku Słowniczek de novo - kategoria metod przewidywania struktury, w których nie używa się wzorców homologicznych
Modelowanie białek ab initio / de novo Słowniczek de novo - od początku, na nowo ab initio - od początku Słowniczek de novo - kategoria metod przewidywania struktury, w których nie używa się wzorców homologicznych
Badanie oddziaływań związków biologicznie aktywnych z modelowymi membranami lipidowymi
 UNIWERSYTET JAGIELLOŃSKI W KRAKOWIE WYDZIAŁ CHEMII STRESZCZENIE ROZPRAWY DOKTORSKIEJ Badanie oddziaływań związków biologicznie aktywnych z modelowymi membranami lipidowymi Marcelina Gorczyca Promotorzy:
UNIWERSYTET JAGIELLOŃSKI W KRAKOWIE WYDZIAŁ CHEMII STRESZCZENIE ROZPRAWY DOKTORSKIEJ Badanie oddziaływań związków biologicznie aktywnych z modelowymi membranami lipidowymi Marcelina Gorczyca Promotorzy:
Właściwości błony komórkowej
 Właściwości błony komórkowej płynność asymetria selektywna przepuszczalność Transport przez błony Cząsteczki < 150Da Błony - selektywnie przepuszczalne RóŜnice składu jonowego między wnętrzem komórki ssaka
Właściwości błony komórkowej płynność asymetria selektywna przepuszczalność Transport przez błony Cząsteczki < 150Da Błony - selektywnie przepuszczalne RóŜnice składu jonowego między wnętrzem komórki ssaka
4.1 Hierarchiczna budowa białek
 Spis treści 4.1 ierarchiczna budowa białek... 51 4.1.1 Struktura pierwszorzędowa... 51 4.1.2 Struktura drugorzędowa... 53 4.1.3 Struktura trzeciorzędowa... 60 4.1.4 Rodzaje oddziaływań stabilizujących
Spis treści 4.1 ierarchiczna budowa białek... 51 4.1.1 Struktura pierwszorzędowa... 51 4.1.2 Struktura drugorzędowa... 53 4.1.3 Struktura trzeciorzędowa... 60 4.1.4 Rodzaje oddziaływań stabilizujących
