Motywacja. Do tej pory: Dzisiaj:
|
|
|
- Barbara Kołodziej
- 5 lat temu
- Przeglądów:
Transkrypt
1 GENE SET ENRICHMENT
2 Motywacja Do tej pory: Gen traktowany jest niezależnie Konieczna jest korekcja dla wielokrotnego testowania (FDR) Wynikowa lista interesujących genów jest zmienna Biologiczna interpretacja takiej listy genów jest trudna Dzisiaj: Predefiniowane grupy genów dostarczają większej wiedzy biologicznej Bardziej znacząca interpretacja w kontekście biologicznym Liczba zbiorów genów jest mniejsza niż liczba indywidualnych genów Przydatne, aby potwierdzić publicznie dostępne grupy genów. Np. Czy sygnatura genu ma wartość przewidującą?
3 Grupy genów po klastrowaniu Co zrobić z grupą genów, kiedy już ją znajdziemy? W jaki sposób sprawdzić czy znaleziona grupa niesie ze sobą pewną informację dla nas? Można porównać do predefiniowanych grup genów dostępnych w bazach danych.
4 Predefiniowane grupy genów Ścieżki: KEGG, Pathway Interaction Database (cmap, BioCarta) Gene Ontology Regiony w genomie Sygnatury dla klasyfikacji Grupy genów z opublikowanych wyników
5 KEGG Kyoto Encyclopedia of Genes and Genomes
6 KEGG pathway
7 KEGG fragment general pathway
8 Gene Ontology Konsorcjum ontologii genów ma na celu ujednolicenie i uspójnienie informacji z różnych baz danych o genach. Rozpoczęto od współpracy pomiędzy bazami 3 organizmów: Drosophila (FlyBase), Saccharomyces Genome Database (SGD) i Mouse Genome Database (MGD) w cellular component Środowisko komórkowe lub zewnątrzkomórkowe molecular function Podstawowe czynności produktów genowych na poziomie cząsteczkowym takim jak kataliza, czy wiązanie biological process Operacje albo zbiory zdarzeń ze zdefiniowanym początkiem i końcem dotyczący funkcjonowania żyjących jednostek: komórek, tkanek, organów i organizmów
9 Jak ocenić związek pomiędzy grupami genów? Zwiększona ekspresja genów w pewnej grupie w porównaniu do genów w innej grupie Przykład: Czy geny, które odpowiadają za cyklinę D1 pokazują niezwykły wzorzec ekspresji dla ludzkich komórek rakowych? Różnicowa ekspresja genów nie dla pojedynczego genu, ale dla specyficznej grupy genów Przykład: Czy ścieżka cyklu komórkowego zawiera (wiele) genów ulegających zróżnicowanej ekspresji dla komórek rakowych typu A i B? Dwie podstawowe strategie do analizy: Gene set enrichment (wzbogacenie grupy genów) i podejście holistyczne (całościowe)
10 Testowanie grupowe Gene set enrichment Sposób: sprawdzenie znaczenia biologicznego dla listy interesujących genów poprzez analizę nadreprezentacji Krok 1: analiza pojedynczych genów (np. różnicowa ekspresja) Krok 2: Ocena grupy genów poprzez wzbogacenie (zawsze w porównaniu do wszystkich genów) Cel: Znaleźć grupy genów, które się składają z wielu interesujących genów Fisher-test, GSEA Podejście holistyczne Sposób: Patrzenie bezpośrednio na grupy genów i pytanie czy są znaczące biologicznie pod względem różnicowej ekspresji Globalna analiza różnicowej ekspresji dla grupy genów (bez brania jako referencji wszystkich genów) Cel: Znalezienie grupy genów które zawierają przynajmniej jeden interesujący gen, lub wiele genów o umiarkowanej różnorodności Kategoria, globaltest, GlobalAncova
11 Test hipergeometryczny, czyli test Fishera Krok 1 Wyznacz różnicową ekspresję dla genu (np. p-value dla statystyki t) Dostosuj p-value do wielokrotnego testowania (FDR) i wybierz odcięcie, które zdefiniuje listę interesujących genów Krok 2 K np. klastrowanie x N N wszystkie geny na mikromacierzy M wybrana grupa genów geny ze zróżnicowaną ekspresją (DE) geny bez zróżnicowanej ekspresji M Jakie jest prawdopodobieństwo że nasza grupa genów będzie wzbogacona w geny ze zróżnicowaną ekspresją?
12 Test hipergeometryczny, czyli test Fishera Krok 1 Wyznacz różnicową ekspresję dla genu (np. p-value dla statystyki t) Dostosuj p-value do wielokrotnego testowania (FDR) i wybierz odcięcie, które zdefiniuje listę interesujących genów Krok 2 Mając N genów na mikromacierzy i M genów w grupie, jakie jest prawdopodobieństwo posiadania x z K interesujących genów w tej grupie? P X = x N, M, K = M K N M K x N K K x p-value dla grupy genów odpowiada P(X>=x N,M,K) N M
13 Test hipergeometryczny, czyli test Fishera Hipergeometryczny test jest równoważny testowi Fishera z grupy genów poza grupą genów geny z DE x K x K geny bez DE M x (N M) (K x) N K M N M N Test Fishera jest podobny do testów opartych na zliczaniu genów bardzo często używanych w analizie ontologii genów Jaką można postawić hipotezę H 0?
14 Test hipergeometryczny, czyli test Fishera Przykład: N = genów na mikromacierzy, M = 100 genów w grupie, która nas interesuje, K = 300 genów ze zróżnicowaną ekspresją z grupy poza grupą z DE bez DE p-value = 0.19 Takie rozłożenie genów w grupie może być przez przypadek z grupy poza grupą z DE bez DE p-value = p-value wskazuje, że grupa ta jest wzbogacona w geny ze zróżnicowaną ekspresją
15 Test hipergeometryczny, czyli test Fishera Zalety Nie jest uzależnione tylko od analizy różnicowej ekspresji (np. badanie wzbogacenia w ścieżki z bazy KEGG) Dla wybranej w jakiś sposób listy interesujących nas genów, ocena biologicznego tła poprzez test oparty na liczności genów jest jedynym sposobem Problemy Utrata informacji poprzez dwa oddzielne kroki Niewielka różnica w ekspresji nie jest widoczna Podział na geny znaczące (z DE) i nieznaczące jest sztuczny (jaki próg p- value przyjąć?)
16 Gene Set Enrichment Analysis GSEA, analiza wzbogacenia grupy genów Krok 1 Wyznacz różnicową ekspresję dla genu (np. p-value dla statystyki t) Utwórz ranking genów zgodnie do wyznaczonej miary (np. p-value) Krok 2 Przypisz etykietę A genom należącym do interesującej nas grupy genów, a etykietę B pozostałym genom Jeśli grupa A jest wzbogacona (enriched) w znaczące geny (DE), wiele z jej genów będzie miało wysoką ocenę w rankingu i będziemy obserwowali je najczęściej na początku rankingu BABAAABBABBAABBABAAABAABBBBBABBABBBBABBBBBBABABABBBBBBBBABABBB.. ABBABBBBBBBBBABABBABBBBAABBBBABBABAABBBBBABBBBBABBAABAAABBBBAB..
17 Gene Set Enrichment Analysis GSEA Dla genów z klasy A dodaj n A punktów a dla genów z klasy B odejmij n B punktów Narysuj skumulowaną sumę tych punktów Czy maksimum M ze skumulowanej sumy jest niezwykle wysokie? test Kolmogorova-Smirnova
18 GSEA Jak bardzo powinna odskoczyć od 0 suma skumulowana (enrichment score), aby uznać wynik za znaczący? Można przeprowadzić test permutacji. Jing Shi, Michael G. Walker, Gene Set Enrichment Analysis (GSEA) for Interpreting Gene Expression Profiles Current Bioinformatics vol.2 no.2, 2007.
19 GSEA test permutacji Permutacja genów Przestaw etykiety A i B w uporządkowanej liście k razy Wyznacz maksimum M * ze skumulowanych sum dla każdej permutacji Wyznacz p-value: ile razy M * było większe od M (dla oryginalnego zbioru), podzielone przez liczbę testów p = # (M * > M)/k Permutacja obiektów Przestaw etykiety próbek k razy Jeśli były 2 rodzaje próbek (po kilka powtórzeń), przydziel każdemu z powtórzeń losowo etykiety z próbki nr. 1 lub 2 Dla każdej z k permutacji: Wyznacz różnicową ekspresję genów (np. t-test) i utwórz ranking jak w Kroku 1 algorytmu GSEA Wyznacz skumulowaną sumę M * Wyznacz p-value jak wyżej
20 GSEA Zalety Nie jest uzależnione tylko od analizy różnicowej ekspresji (np. badanie wzbogacenia w ścieżki z bazy KEGG) Wprowadzony ranking genów powoduje, iż nie ma sztucznego odcięcia na 2 grupy Problemy Utrata informacji poprzez dwa oddzielne kroki Niewielka różnica w ekspresji nie jest widoczna Można polepszyć tę metodę poprzez wykonanie testy permutacji (dla genów lub próbek)
21 Cytoscape i Bingo - prezentacja
22
23
24
25
26 Co dalej można zrobić z genami z interesującej nas grupy? Można przyjrzeć się adnotacji genów i szukać dalszych informacji
27 Dostępne bazy danych do adnotacji Bazy literaturowe PubMed, Medline, OMIM Bazy rzeczywiste Sekwencje nukleotydowe: GenBank Sekwencje aminokwasowe: SwissProt Struktury cząsteczek 3D: PDB Bazy wiedzy Klasyfikacja genów: UniGene Ontologia genów: GO Biblioteki motywów: Prosite Ścieżki: KEGG, WIT Czynniki transkrypcyjne: Transfac
28 Przykład Name Interleukin 13 Gene Symbol Il13 GenBank accession NM_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc
29
30 Przykład Name Interleukin 13 Gene Symbol Il13 GenBank accession NM_ UniGene NP_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc
31
32 Przykład Name Interleukin 13 Gene Symbol Il13 GenBank accession NM_ UniGene NP_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains
33
34 Przykład Name Interleukin 13 Gene Symbol Il13 Chromosome 11 31,98 cm MGI (Mouse Genome Informatics) GenBank accession NM_ UniGene NP_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains
35
36 Przykład Name Interleukin 13 GenBank accession NM_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc Gene Symbol Il13 Chromosome 11 31,98 cm MGI UniGene NP_ PubMed ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains
37
38 Przykład Name Interleukin 13 GenBank accession NM_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc Gene Symbol Il13 Chromosome 11 31,98 cm MGI UniGene NP_ PubMed GEO Profiles (Gene Expression Omnibus) ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains
39
40 Przykład Name Interleukin 13 GenBank accession NM_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc Gene Symbol Il13 Chromosome 11 31,98 cm MGI UniGene NP_ PubMed GEO Profiles (Gene Expression Omnibus) UniProtKB P20109 ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains
41
42 Przykład Name Interleukin 13 GenBank accession NM_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc Gene Symbol Il13 Chromosome 11 31,98 cm MGI UniGene NP_ PubMed GEO Profiles (Gene Expression Omnibus) UniProtKB P20109 ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains AmiGO Il13
43
44 Przykład Name Interleukin 13 GenBank accession NM_ Nucleotide sequence acaagccagcagcctaggccagcccacagttctacagctccctggttctctcactg gctctgggcttcatggcgctctgggtgactgcagtcctggctcttgcttgccttggt ggtctcgccgccccagggccggtgccaagatctgtgtctctccctctgacccttaa ggagcttattgaggagctgagcaacatcacacaagaccagactcccctgtgcaa cggcagcatggtatggagtgtggacctggccgctggcgggttctgtgtagccct ggattccctgaccaacatctccaattgcaatgccatctacaggacccagaggatat tgcatggcctctgtaaccgcaaggcccccactacggtctccagcctcccc Gene Symbol Il13 Chromosome 11 31,98 cm MGI UniGene NP_ PubMed GEO Profiles (Gene Expression Omnibus) UniProtKB P20109 ProtEST Protein/EST matches BLink Protein/protein matches CDD Conserved domains AmiGO Il13 KEGG pathway 16163
45
46
47 MIAME Minimum Information About a Microarray Experiment
48
49 Sześć najważniejszych elementów wg MIAME 1. Dane surowe z hybrydyzacji (np. CEL, GPR) 2. Końcowo przetworzone dane (znormalizowane) dla całego zbioru hybrydyzacji w eksperymencie (macierz ekspresji genów) 3. Niezbędna adnotacja próbki włączając w to eksperymentalny czynnik i jego dawka 4. Projekt eksperymentu włączając w to związki pomiędzy próbkami (które dane surowe odnoszą się do której próbki, które hybrydyzacje są czysto techniczne, które są biologicznymi powtórzeniami) 5. Wystarczająca adnotacja macierzy (identyfikatory genów, koordynaty genomiczne, sekwencje oligonukleotydowe) 6. Opis protokołu laboratoryjnego i przetworzenia danych (np. metoda normalizacji, itp.)
1. KEGG 2. GO. 3. Klastry
 ANALIZA DANYCH 1. Wykład wstępny 2. Charakterystyka danych 3. Analiza wstępna genomiczna charakterystyka cech 4. Prezentacje grup roboczych analiza wstępna 5. Prezentacje grup roboczych analiza wstępna
ANALIZA DANYCH 1. Wykład wstępny 2. Charakterystyka danych 3. Analiza wstępna genomiczna charakterystyka cech 4. Prezentacje grup roboczych analiza wstępna 5. Prezentacje grup roboczych analiza wstępna
Bioinformatyka. Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl
 Bioinformatyka Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Bazy danych biologicznych Bazy danych sekwencji nukleotydowych Pierwotne bazy danych (ang. primary database) Wykorzystywane do zbierania
Bioinformatyka Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Bazy danych biologicznych Bazy danych sekwencji nukleotydowych Pierwotne bazy danych (ang. primary database) Wykorzystywane do zbierania
Wstęp do Biologii Obliczeniowej
 Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
MIKROMACIERZE. dr inż. Aleksandra Świercz dr Agnieszka Żmieńko
 MIKROMACIERZE dr inż. Aleksandra Świercz dr Agnieszka Żmieńko Informacje ogólne Wykłady będą częściowo dostępne w formie elektronicznej http://cs.put.poznan.pl/aswiercz aswiercz@cs.put.poznan.pl Godziny
MIKROMACIERZE dr inż. Aleksandra Świercz dr Agnieszka Żmieńko Informacje ogólne Wykłady będą częściowo dostępne w formie elektronicznej http://cs.put.poznan.pl/aswiercz aswiercz@cs.put.poznan.pl Godziny
Bioinformatyka. Michał Bereta
 Bioinformatyka Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Bazy danych biologicznych Bazy danych sekwencji nukleotydowych Pierwotne bazy danych (ang. primary database) Wykorzystywane do zbierania
Bioinformatyka Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Bazy danych biologicznych Bazy danych sekwencji nukleotydowych Pierwotne bazy danych (ang. primary database) Wykorzystywane do zbierania
2014-03-26. Analiza sekwencji promotorów
 2014-03-26 Analiza sekwencji promotorów 1 2014-03-26 TFy tworzą zawiły układ regulacyjny, na który składają się różne oddziaływania białko białko poprzez wytworzenie PĘTLI Specyficzne TFy Ogólne TFy Benfey,
2014-03-26 Analiza sekwencji promotorów 1 2014-03-26 TFy tworzą zawiły układ regulacyjny, na który składają się różne oddziaływania białko białko poprzez wytworzenie PĘTLI Specyficzne TFy Ogólne TFy Benfey,
Dane mikromacierzowe. Mateusz Markowicz Marta Stańska
 Dane mikromacierzowe Mateusz Markowicz Marta Stańska Mikromacierz Mikromacierz DNA (ang. DNA microarray) to szklana lub plastikowa płytka (o maksymalnych wymiarach 2,5 cm x 7,5 cm) z naniesionymi w regularnych
Dane mikromacierzowe Mateusz Markowicz Marta Stańska Mikromacierz Mikromacierz DNA (ang. DNA microarray) to szklana lub plastikowa płytka (o maksymalnych wymiarach 2,5 cm x 7,5 cm) z naniesionymi w regularnych
Pytania i odpowiedzi
 Pytania i odpowiedzi PCA PCA a MDS - PCA bazuje na macierzy kowariancji, MDS bazuje na macierzy dystansów genetycznych Będą identyczne jeśli kowariancja będzie równa odległości euklidesowej. W badaniach
Pytania i odpowiedzi PCA PCA a MDS - PCA bazuje na macierzy kowariancji, MDS bazuje na macierzy dystansów genetycznych Będą identyczne jeśli kowariancja będzie równa odległości euklidesowej. W badaniach
Database resources of the National Center for Biotechnology Information. Magdalena Malczyk
 Database resources of the National Center for Biotechnology Information Magdalena Malczyk NCBI NCBI = National Center for Biotechnology Information Założone w 1998 r. Cel: rozwijanie systemów informatycznych
Database resources of the National Center for Biotechnology Information Magdalena Malczyk NCBI NCBI = National Center for Biotechnology Information Założone w 1998 r. Cel: rozwijanie systemów informatycznych
Przykładowa analiza danych
 Przykładowa analiza danych W analizie wykorzystano dane pochodzące z publicznego repozytorium ArrayExpress udostępnionego na stronach Europejskiego Instytutu Bioinformatyki (http://www.ebi.ac.uk/). Zbiór
Przykładowa analiza danych W analizie wykorzystano dane pochodzące z publicznego repozytorium ArrayExpress udostępnionego na stronach Europejskiego Instytutu Bioinformatyki (http://www.ebi.ac.uk/). Zbiór
BIOINFORMATYKA BIOLOGICZNE BAZY DANYCH
 http://theta.edu.pl/ Podstawy Bioinformatyki II BIOINFORMATYKA BIOLOGICZNE BAZY DANYCH 1 Czym jest bioinformatyka? 2 Bioinformatyka Bioinformatyka jest interdyscyplinarną dziedziną nauki obejmującą wykorzystanie
http://theta.edu.pl/ Podstawy Bioinformatyki II BIOINFORMATYKA BIOLOGICZNE BAZY DANYCH 1 Czym jest bioinformatyka? 2 Bioinformatyka Bioinformatyka jest interdyscyplinarną dziedziną nauki obejmującą wykorzystanie
PODSTAWY BIOINFORMATYKI WYKŁAD 3 BIOLOGICZNE BAZY DANYCH (1)
 PODSTAWY BIOINFORMATYKI WYKŁAD 3 BIOLOGICZNE BAZY DANYCH (1) BIOINFORMATYKA HISTORIA 1. 1982 utworzenie bazy danych GenBank (NIH) dane ogólnodostępne sekwencje nukleotydów 2. Wprowadzenie sekwencji z projektu
PODSTAWY BIOINFORMATYKI WYKŁAD 3 BIOLOGICZNE BAZY DANYCH (1) BIOINFORMATYKA HISTORIA 1. 1982 utworzenie bazy danych GenBank (NIH) dane ogólnodostępne sekwencje nukleotydów 2. Wprowadzenie sekwencji z projektu
Bioinformatyka. Bazy danych. Wykład 3. E. Banachowicz. Wykład monograficzny Bioinformatyka. Wykład 3, Zakład Biofizyki Molekularnej IF UAM
 Bioinformatyka Wykład 3. E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Bazy danych Wykład 3, 2008 1 Niesekwencyjne BazyDanych bibliograficzne kliniczne ścieżek metabolicznych
Bioinformatyka Wykład 3. E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Bazy danych Wykład 3, 2008 1 Niesekwencyjne BazyDanych bibliograficzne kliniczne ścieżek metabolicznych
Podstawy bioinformatyki - biologiczne bazy danych
 Podstawy bioinformatyki - biologiczne bazy danych Czym jest bioinformatyka? Bioinformatyka Bioinformatyka jest interdyscyplinarną dziedziną nauki obejmującą wykorzystanie metod obliczeniowych do badania
Podstawy bioinformatyki - biologiczne bazy danych Czym jest bioinformatyka? Bioinformatyka Bioinformatyka jest interdyscyplinarną dziedziną nauki obejmującą wykorzystanie metod obliczeniowych do badania
Public gene expression data repositoris
 Public gene expression data repositoris GEO [Jan 2011]: 520 k samples 21 k experiments Homo, mus, rattus Bos, sus Arabidopsis, oryza, Salmonella, Mycobacterium et al. 17.01.11 14 17.01.11 15 17.01.11 16
Public gene expression data repositoris GEO [Jan 2011]: 520 k samples 21 k experiments Homo, mus, rattus Bos, sus Arabidopsis, oryza, Salmonella, Mycobacterium et al. 17.01.11 14 17.01.11 15 17.01.11 16
Bioinformatyczne bazy danych
 Bioinformatyczne bazy danych Czym jest bioinformatyka? Bioinformatyka jest nauką integrującą różne dziedziny wiedzy Gruca (2010) Czym jest bioinformatyka? Bioinformatyka obejmuje technologie wykorzystujące
Bioinformatyczne bazy danych Czym jest bioinformatyka? Bioinformatyka jest nauką integrującą różne dziedziny wiedzy Gruca (2010) Czym jest bioinformatyka? Bioinformatyka obejmuje technologie wykorzystujące
Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2
 ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
PODSTAWY BIOINFORMATYKI WYKŁAD 3 BIOLOGICZNE BAZY DANYCH (2)
 PODSTAWY BIOINFORMATYKI WYKŁAD 3 BIOLOGICZNE BAZY DANYCH (2) BIOINFORMATYKA HISTORIA 1. 1982 utworzenie bazy danych GenBank (NIH) dane ogólnodostępne sekwencje nukleotydów 2. Wprowadzenie sekwencji z projektu
PODSTAWY BIOINFORMATYKI WYKŁAD 3 BIOLOGICZNE BAZY DANYCH (2) BIOINFORMATYKA HISTORIA 1. 1982 utworzenie bazy danych GenBank (NIH) dane ogólnodostępne sekwencje nukleotydów 2. Wprowadzenie sekwencji z projektu
Bioinformatyczne bazy danych
 Bioinformatyczne bazy danych Czym jest bioinformatyka? Bioinformatyka jest nauką integrującą różne dziedziny wiedzy Gruca (2010) http://bioinformaticsonline.com/file/view/4482/bioinformatics-definitions-and-applications
Bioinformatyczne bazy danych Czym jest bioinformatyka? Bioinformatyka jest nauką integrującą różne dziedziny wiedzy Gruca (2010) http://bioinformaticsonline.com/file/view/4482/bioinformatics-definitions-and-applications
Analiza zmienności czasowej danych mikromacierzowych
 Systemy Inteligencji Obliczeniowej Analiza zmienności czasowej danych mikromacierzowych Kornel Chromiński Instytut Informatyki Uniwersytet Śląski Plan prezentacji Dane mikromacierzowe Cel badań Prezentacja
Systemy Inteligencji Obliczeniowej Analiza zmienności czasowej danych mikromacierzowych Kornel Chromiński Instytut Informatyki Uniwersytet Śląski Plan prezentacji Dane mikromacierzowe Cel badań Prezentacja
Bioinformatyka 2 (BT172) Struktura i organizacja kursu
 Bioinformatyka 2 (BT172) Wykład 1 Struktura i organizacja kursu dr Krzysztof Murzyn adiunkt w Zakładzie Biofizyki WBtUJ pok. B028, tel. 664-6379 10.X.2005 PODSTAWOWE INFORMACJE 9 godz. wykładów (45 min,
Bioinformatyka 2 (BT172) Wykład 1 Struktura i organizacja kursu dr Krzysztof Murzyn adiunkt w Zakładzie Biofizyki WBtUJ pok. B028, tel. 664-6379 10.X.2005 PODSTAWOWE INFORMACJE 9 godz. wykładów (45 min,
Bioinformatyka wykład I.2009
 Bioinformatyka wykład 13 20.I.2009 biologia systemów biologiczne dane wielowymiarowe Krzysztof Pawłowski Krzysztof_Pawlowski@sggw.pl 2009-01-22 1 Plan wykładu Biologia systemów Bazy danych ekspresji genów
Bioinformatyka wykład 13 20.I.2009 biologia systemów biologiczne dane wielowymiarowe Krzysztof Pawłowski Krzysztof_Pawlowski@sggw.pl 2009-01-22 1 Plan wykładu Biologia systemów Bazy danych ekspresji genów
Bioinformatyka, edycja 2016/2017, laboratorium
 Instytut Informatyki i Matematyki Komputerowej UJ, opracowanie: dr Jacek Śmietański Mikromacierze 1. Mikromacierze wprowadzenie Mikromacierze to technologia pozwalająca na pomiar aktywności genów w komórce.
Instytut Informatyki i Matematyki Komputerowej UJ, opracowanie: dr Jacek Śmietański Mikromacierze 1. Mikromacierze wprowadzenie Mikromacierze to technologia pozwalająca na pomiar aktywności genów w komórce.
Bioinformatyczne bazy danych - część 2. -przeszukiwanie baz danych -pobieranie danych
 Bioinformatyczne bazy danych - część 2 -przeszukiwanie baz danych -pobieranie danych Numery dostępowe baz danych (accession number) to ciąg liter i cyfr służących jako etykieta identyfikująca sekwencję
Bioinformatyczne bazy danych - część 2 -przeszukiwanie baz danych -pobieranie danych Numery dostępowe baz danych (accession number) to ciąg liter i cyfr służących jako etykieta identyfikująca sekwencję
Testowanie hipotez statystycznych
 9 października 2008 ...czyli definicje na rozgrzewkę n-elementowa próba losowa - wektor n zmiennych losowych (X 1,..., X n ); intuicyjnie: wynik n eksperymentów realizacja próby (X 1,..., X n ) w ω Ω :
9 października 2008 ...czyli definicje na rozgrzewkę n-elementowa próba losowa - wektor n zmiennych losowych (X 1,..., X n ); intuicyjnie: wynik n eksperymentów realizacja próby (X 1,..., X n ) w ω Ω :
Analizy wielkoskalowe w badaniach chromatyny
 Analizy wielkoskalowe w badaniach chromatyny Analizy wielkoskalowe wykorzystujące mikromacierze DNA Genotypowanie: zróżnicowane wewnątrz genów RNA Komórka eukariotyczna Ekspresja genów: Które geny? Poziom
Analizy wielkoskalowe w badaniach chromatyny Analizy wielkoskalowe wykorzystujące mikromacierze DNA Genotypowanie: zróżnicowane wewnątrz genów RNA Komórka eukariotyczna Ekspresja genów: Które geny? Poziom
SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO
 SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO. Rzeczywistość (istniejąca lub projektowana).. Model fizyczny. 3. Model matematyczny (optymalizacyjny): a. Zmienne projektowania
SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO. Rzeczywistość (istniejąca lub projektowana).. Model fizyczny. 3. Model matematyczny (optymalizacyjny): a. Zmienne projektowania
Nowoczesne systemy ekspresji genów
 Nowoczesne systemy ekspresji genów Ekspresja genów w organizmach żywych GEN - pojęcia podstawowe promotor sekwencja kodująca RNA terminator gen Gen - odcinek DNA zawierający zakodowaną informację wystarczającą
Nowoczesne systemy ekspresji genów Ekspresja genów w organizmach żywych GEN - pojęcia podstawowe promotor sekwencja kodująca RNA terminator gen Gen - odcinek DNA zawierający zakodowaną informację wystarczającą
Ocena jakości modeli strukturalnych białek w oparciu o podobieństwo strukturalne i semantyczny opis funkcji w ontologii GO
 Ocena jakości modeli strukturalnych białek w oparciu o podobieństwo strukturalne i semantyczny opis funkcji w ontologii GO Bogumil Konopka 1, Jean-Christophe Nebel 2, Malgorzata Kotulska 1 * 1 Politechnika
Ocena jakości modeli strukturalnych białek w oparciu o podobieństwo strukturalne i semantyczny opis funkcji w ontologii GO Bogumil Konopka 1, Jean-Christophe Nebel 2, Malgorzata Kotulska 1 * 1 Politechnika
Podobieństwo semantyczne w ontologiach biomedycznych
 Podobieństwo semantyczne w ontologiach biomedycznych Bogumił Konopka Politechnika Wrocławska Wydział Podstawowych Problemów Techniki Instytut Inżynierii Biomedycznej i Pomiarowej KN Bio Nanopor Plan prezentacji
Podobieństwo semantyczne w ontologiach biomedycznych Bogumił Konopka Politechnika Wrocławska Wydział Podstawowych Problemów Techniki Instytut Inżynierii Biomedycznej i Pomiarowej KN Bio Nanopor Plan prezentacji
Przybliżone algorytmy analizy ekspresji genów.
 Przybliżone algorytmy analizy ekspresji genów. Opracowanie i implementacja algorytmu analizy danych uzyskanych z eksperymentu biologicznego. 20.06.04 Seminarium - SKISR 1 Wstęp. Dane wejściowe dla programu
Przybliżone algorytmy analizy ekspresji genów. Opracowanie i implementacja algorytmu analizy danych uzyskanych z eksperymentu biologicznego. 20.06.04 Seminarium - SKISR 1 Wstęp. Dane wejściowe dla programu
Kontakt.
 BIOINFORMATYKA i BIOLOGIA OBLICZENIOWA Kontakt Małgorzata.Kotulska@pwr.edu.pl Katedra Inżynierii Biomedycznej WPPT http://www.kotulska-lab.pwr.wroc.pl/forstudents/ D1 pok. 115 Konsultacje : czwartek 17.05-18.55
BIOINFORMATYKA i BIOLOGIA OBLICZENIOWA Kontakt Małgorzata.Kotulska@pwr.edu.pl Katedra Inżynierii Biomedycznej WPPT http://www.kotulska-lab.pwr.wroc.pl/forstudents/ D1 pok. 115 Konsultacje : czwartek 17.05-18.55
PODSTAWY BIOINFORMATYKI
 PODSTAWY BIOINFORMATYKI Prowadzący: JOANNA SZYDA ADRIAN DROśDś WSTĘP 1. Katedra Genetyki badania bioinformatyczne 2. Tematyka przedmiotu 3. Charakterystyka wykładów 4. Charakterystyka ćwiczeń 5. Informacje
PODSTAWY BIOINFORMATYKI Prowadzący: JOANNA SZYDA ADRIAN DROśDś WSTĘP 1. Katedra Genetyki badania bioinformatyczne 2. Tematyka przedmiotu 3. Charakterystyka wykładów 4. Charakterystyka ćwiczeń 5. Informacje
ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH
 1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
Przykład 1. (A. Łomnicki)
 Plan wykładu: 1. Wariancje wewnątrz grup i między grupami do czego prowadzi ich ocena 2. Rozkład F 3. Analiza wariancji jako metoda badań założenia, etapy postępowania 4. Dwie klasyfikacje a dwa modele
Plan wykładu: 1. Wariancje wewnątrz grup i między grupami do czego prowadzi ich ocena 2. Rozkład F 3. Analiza wariancji jako metoda badań założenia, etapy postępowania 4. Dwie klasyfikacje a dwa modele
PODSTAWY BIOINFORMATYKI 12 MIKROMACIERZE
 PODSTAWY BIOINFORMATYKI 12 MIKROMACIERZE WSTĘP 1. Mikromacierze ekspresyjne tworzenie macierzy przykłady zastosowań 2. Mikromacierze SNP tworzenie macierzy przykłady zastosowań MIKROMACIERZE EKSPRESYJNE
PODSTAWY BIOINFORMATYKI 12 MIKROMACIERZE WSTĘP 1. Mikromacierze ekspresyjne tworzenie macierzy przykłady zastosowań 2. Mikromacierze SNP tworzenie macierzy przykłady zastosowań MIKROMACIERZE EKSPRESYJNE
Statystyka matematyczna i ekonometria
 Statystyka matematyczna i ekonometria prof. dr hab. inż. Jacek Mercik B4 pok. 55 jacek.mercik@pwr.wroc.pl (tylko z konta studenckiego z serwera PWr) Konsultacje, kontakt itp. Strona WWW Elementy wykładu.
Statystyka matematyczna i ekonometria prof. dr hab. inż. Jacek Mercik B4 pok. 55 jacek.mercik@pwr.wroc.pl (tylko z konta studenckiego z serwera PWr) Konsultacje, kontakt itp. Strona WWW Elementy wykładu.
Jest to dziedzina biologiczna wywodząca się z biotechnologii. Bioinformatyka
 Wstęp do obsługi biologicznych baz danych i analizy porównawczej białek i genów Katedra Fizjologii i Biotechnologii Roślin Pok. 113 CB jan.jastrzebski@uwm.edu.pl bioinformatyka@gmail.com www.ebiology.net
Wstęp do obsługi biologicznych baz danych i analizy porównawczej białek i genów Katedra Fizjologii i Biotechnologii Roślin Pok. 113 CB jan.jastrzebski@uwm.edu.pl bioinformatyka@gmail.com www.ebiology.net
Indeksy w bazach danych. Motywacje. Techniki indeksowania w eksploracji danych. Plan prezentacji. Dotychczasowe prace badawcze skupiały się na
 Techniki indeksowania w eksploracji danych Maciej Zakrzewicz Instytut Informatyki Politechnika Poznańska Plan prezentacji Zastosowania indeksów w systemach baz danych Wprowadzenie do metod eksploracji
Techniki indeksowania w eksploracji danych Maciej Zakrzewicz Instytut Informatyki Politechnika Poznańska Plan prezentacji Zastosowania indeksów w systemach baz danych Wprowadzenie do metod eksploracji
Wykład 8 Dane kategoryczne
 Wykład 8 Dane kategoryczne Wrocław, 19.04.2017r Zmienne kategoryczne 1 Przykłady zmiennych kategorycznych 2 Zmienne nominalne, zmienne ordynalne (porządkowe) 3 Zmienne dychotomiczne kodowanie zmiennych
Wykład 8 Dane kategoryczne Wrocław, 19.04.2017r Zmienne kategoryczne 1 Przykłady zmiennych kategorycznych 2 Zmienne nominalne, zmienne ordynalne (porządkowe) 3 Zmienne dychotomiczne kodowanie zmiennych
BUDOWA I FUNKCJA GENOMU LUDZKIEGO
 BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
Statystyczna analiza Danych
 Statystyczna analiza Danych Dla bioinformatyków Wykład pierwszy: O testowaniu hipotez Plan na dziś Quiz! Cele wykładu Plan na semestr Kryteria zaliczenia Sprawy organizacyjne Quiz (15 minut) Jakie znasz
Statystyczna analiza Danych Dla bioinformatyków Wykład pierwszy: O testowaniu hipotez Plan na dziś Quiz! Cele wykładu Plan na semestr Kryteria zaliczenia Sprawy organizacyjne Quiz (15 minut) Jakie znasz
Jak wybrać 45 najlepszych prezentacji na FORUM?
 Być data driven company w erze Big Data. Jak wybrać 45 najlepszych prezentacji na FORUM? Doświadczenia z budowy platformy do prowadzenia na dużą skalę eksperymentów A/B Joanna Radosław Komuda, Kita, IAB
Być data driven company w erze Big Data. Jak wybrać 45 najlepszych prezentacji na FORUM? Doświadczenia z budowy platformy do prowadzenia na dużą skalę eksperymentów A/B Joanna Radosław Komuda, Kita, IAB
BIOLOGICZNE BAZY DANYCH SYLABUS
 BIOLOGICZNE BAZY DANYCH SYLABUS Elementy składowe sylabusu Nazwa jednostki prowadzącej kierunek Nazwa kierunku studiów Poziom kształcenia Profil studiów Forma studiów Kod Język Rodzaj Rok studiów /semestr
BIOLOGICZNE BAZY DANYCH SYLABUS Elementy składowe sylabusu Nazwa jednostki prowadzącej kierunek Nazwa kierunku studiów Poziom kształcenia Profil studiów Forma studiów Kod Język Rodzaj Rok studiów /semestr
Bioinformatyka. Rodzaje Mutacji
 Bioinformatyka (wykład monograficzny) wykład 3. E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Rodzaje Mutacji zmienność sekwencji (sequence variation) mutacje polimorfizm
Bioinformatyka (wykład monograficzny) wykład 3. E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Rodzaje Mutacji zmienność sekwencji (sequence variation) mutacje polimorfizm
Elementy statystyki wielowymiarowej
 Wnioskowanie_Statystyczne_-_wykład Spis treści 1 Elementy statystyki wielowymiarowej 1.1 Kowariancja i współczynnik korelacji 1.2 Macierz kowariancji 1.3 Dwumianowy rozkład normalny 1.4 Analiza składowych
Wnioskowanie_Statystyczne_-_wykład Spis treści 1 Elementy statystyki wielowymiarowej 1.1 Kowariancja i współczynnik korelacji 1.2 Macierz kowariancji 1.3 Dwumianowy rozkład normalny 1.4 Analiza składowych
BIOLOGICZNE BAZY DANYCH (1) GENOMY I ICH ADNOTACJE
 BIOLOGICZNE BAZY DANYCH (1) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 2 PODSTAWY BIOINFORMATYKI 2018/2019 MAGDA MIELCZAREK 1 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI
BIOLOGICZNE BAZY DANYCH (1) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 2 PODSTAWY BIOINFORMATYKI 2018/2019 MAGDA MIELCZAREK 1 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI
KARTA PRZEDMIOTU. (pieczęć wydziału)
 (pieczęć wydziału) KARTA PRZEDMIOTU 1. Nazwa przedmiotu: Eksploracja danych w bioinformatyce 2. Kod przedmiotu: EksDaBio 3. Karta przedmiotu ważna od roku akademickiego: 2017/2018 4. Forma kształcenia:
(pieczęć wydziału) KARTA PRZEDMIOTU 1. Nazwa przedmiotu: Eksploracja danych w bioinformatyce 2. Kod przedmiotu: EksDaBio 3. Karta przedmiotu ważna od roku akademickiego: 2017/2018 4. Forma kształcenia:
4.1. Wprowadzenie...70 4.2. Podstawowe definicje...71 4.3. Algorytm określania wartości parametrów w regresji logistycznej...74
 3 Wykaz najważniejszych skrótów...8 Przedmowa... 10 1. Podstawowe pojęcia data mining...11 1.1. Wprowadzenie...12 1.2. Podstawowe zadania eksploracji danych...13 1.3. Główne etapy eksploracji danych...15
3 Wykaz najważniejszych skrótów...8 Przedmowa... 10 1. Podstawowe pojęcia data mining...11 1.1. Wprowadzenie...12 1.2. Podstawowe zadania eksploracji danych...13 1.3. Główne etapy eksploracji danych...15
Modelowanie motywów łańcuchami Markowa wyższego rzędu
 Modelowanie motywów łańcuchami Markowa wyższego rzędu Uniwersytet Warszawski Wydział Matematyki, Informatyki i Mechaniki 23 października 2008 roku Plan prezentacji 1 Źródła 2 Motywy i ich znaczenie Łańcuchy
Modelowanie motywów łańcuchami Markowa wyższego rzędu Uniwersytet Warszawski Wydział Matematyki, Informatyki i Mechaniki 23 października 2008 roku Plan prezentacji 1 Źródła 2 Motywy i ich znaczenie Łańcuchy
Kolokwium ze statystyki matematycznej
 Kolokwium ze statystyki matematycznej 28.05.2011 Zadanie 1 Niech X będzie zmienną losową z rozkładu o gęstości dla, gdzie 0 jest nieznanym parametrem. Na podstawie pojedynczej obserwacji weryfikujemy hipotezę
Kolokwium ze statystyki matematycznej 28.05.2011 Zadanie 1 Niech X będzie zmienną losową z rozkładu o gęstości dla, gdzie 0 jest nieznanym parametrem. Na podstawie pojedynczej obserwacji weryfikujemy hipotezę
BIOLOGICZNE BAZY DANYCH (2) GENOMY I ICH ADNOTACJE. Podstawy Bioinformatyki wykład 4
 BIOLOGICZNE BAZY DANYCH (2) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 4 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI 2017/2018 MAGDA MIELCZAREK 2 GENOMY I ICH ADNOTACJE
BIOLOGICZNE BAZY DANYCH (2) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 4 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI 2017/2018 MAGDA MIELCZAREK 2 GENOMY I ICH ADNOTACJE
CZĘŚĆ III OPISPRZEDMIOTU ZAMÓWIENIA (OPZ)
 INSTYTUT IMMUNOLOGII I TERAPII DOŚWIADCZALNEJ im. Ludwika Hirszfelda Polska Akademia Nauk ul. Rudolfa Weigla 12, 53-114 Wrocław tel. / fax. (4871) 37-09-997, http://www.iitd.pan.wroc.pl NIP: 896-000-56-96;
INSTYTUT IMMUNOLOGII I TERAPII DOŚWIADCZALNEJ im. Ludwika Hirszfelda Polska Akademia Nauk ul. Rudolfa Weigla 12, 53-114 Wrocław tel. / fax. (4871) 37-09-997, http://www.iitd.pan.wroc.pl NIP: 896-000-56-96;
Analiza metod wykrywania przekazów steganograficznych. Magdalena Pejas Wydział EiTI PW magdap7@gazeta.pl
 Analiza metod wykrywania przekazów steganograficznych Magdalena Pejas Wydział EiTI PW magdap7@gazeta.pl Plan prezentacji Wprowadzenie Cel pracy Tezy pracy Koncepcja systemu Typy i wyniki testów Optymalizacja
Analiza metod wykrywania przekazów steganograficznych Magdalena Pejas Wydział EiTI PW magdap7@gazeta.pl Plan prezentacji Wprowadzenie Cel pracy Tezy pracy Koncepcja systemu Typy i wyniki testów Optymalizacja
Spis treści. Przedmowa... XI. Rozdział 1. Pomiar: jednostki miar... 1. Rozdział 2. Pomiar: liczby i obliczenia liczbowe... 16
 Spis treści Przedmowa.......................... XI Rozdział 1. Pomiar: jednostki miar................. 1 1.1. Wielkości fizyczne i pozafizyczne.................. 1 1.2. Spójne układy miar. Układ SI i jego
Spis treści Przedmowa.......................... XI Rozdział 1. Pomiar: jednostki miar................. 1 1.1. Wielkości fizyczne i pozafizyczne.................. 1 1.2. Spójne układy miar. Układ SI i jego
Wykład 10 2008-04-30. Bioinformatyka. Wykład 9. E. Banachowicz. Zakład Biofizyki Molekularnej IF UAM
 Bioinformatyka Wykład 9 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Konsekwencje zestawieo wielu sekwencji - rodziny białkowe, domeny, motywy i wzorce 2 Bioinformatyka,
Bioinformatyka Wykład 9 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Konsekwencje zestawieo wielu sekwencji - rodziny białkowe, domeny, motywy i wzorce 2 Bioinformatyka,
Ćwiczenie 5/6. Informacja genetyczna i geny u różnych grup organizmów. Porównywanie sekwencji nukleotydowych w bazie NCBI z wykorzystaniem BLAST.
 Ćwiczenie 5/6 Informacja genetyczna i geny u różnych grup organizmów. Porównywanie sekwencji nukleotydowych w bazie NCBI z wykorzystaniem BLAST. Prof. dr hab. Roman Zieliński 1. Informacja genetyczna u
Ćwiczenie 5/6 Informacja genetyczna i geny u różnych grup organizmów. Porównywanie sekwencji nukleotydowych w bazie NCBI z wykorzystaniem BLAST. Prof. dr hab. Roman Zieliński 1. Informacja genetyczna u
Bazy danych i biologia
 Bazy danych i biologia Biologiczne Aplikacje Baz Danych Politechnika Poznańska dr inż. Anna Leśniewska alesniewska@cs.put.poznan.pl Biological databases play a central role in bioinformatics. They offer
Bazy danych i biologia Biologiczne Aplikacje Baz Danych Politechnika Poznańska dr inż. Anna Leśniewska alesniewska@cs.put.poznan.pl Biological databases play a central role in bioinformatics. They offer
BIOLOGICZNE BAZY DANYCH GENOMY I ICH ADNOTACJE. Pracownia Informatyczna 2
 BIOLOGICZNE BAZY DANYCH GENOMY I ICH ADNOTACJE Pracownia Informatyczna 2 WYBRANE BIOLOGICZNE BAZY DANYCH GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC NATIONAL CENTER FOR BIOTECHNOLOGY INFORMATION NCBI Utworzone
BIOLOGICZNE BAZY DANYCH GENOMY I ICH ADNOTACJE Pracownia Informatyczna 2 WYBRANE BIOLOGICZNE BAZY DANYCH GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC NATIONAL CENTER FOR BIOTECHNOLOGY INFORMATION NCBI Utworzone
Naszym zadaniem jest rozpatrzenie związków między wierszami macierzy reprezentującej poziomy ekspresji poszczególnych genów.
 ANALIZA SKUPIEŃ Metoda k-means I. Cel zadania Zadaniem jest analiza zbioru danych, gdzie zmiennymi są poziomy ekspresji genów. Podczas badań pobrano próbki DNA od 36 różnych pacjentów z chorobą nowotworową.
ANALIZA SKUPIEŃ Metoda k-means I. Cel zadania Zadaniem jest analiza zbioru danych, gdzie zmiennymi są poziomy ekspresji genów. Podczas badań pobrano próbki DNA od 36 różnych pacjentów z chorobą nowotworową.
Zadanie 1 Przygotuj algorytm programu - sortowanie przez wstawianie.
 Sortowanie Dane wejściowe: ciąg n-liczb (kluczy) (a 1, a 2, a 3,..., a n 1, a n ) Dane wyjściowe: permutacja ciągu wejściowego (a 1, a 2, a 3,..., a n 1, a n) taka, że a 1 a 2 a 3... a n 1 a n. Będziemy
Sortowanie Dane wejściowe: ciąg n-liczb (kluczy) (a 1, a 2, a 3,..., a n 1, a n ) Dane wyjściowe: permutacja ciągu wejściowego (a 1, a 2, a 3,..., a n 1, a n) taka, że a 1 a 2 a 3... a n 1 a n. Będziemy
Przykład 2. Na podstawie książki J. Kowal: Metody statystyczne w badaniach sondażowych rynku
 Przykład 2 Na podstawie książki J. Kowal: Metody statystyczne w badaniach sondażowych rynku Sondaż sieciowy analiza wyników badania sondażowego dotyczącego motywacji w drodze do sukcesu Cel badania: uzyskanie
Przykład 2 Na podstawie książki J. Kowal: Metody statystyczne w badaniach sondażowych rynku Sondaż sieciowy analiza wyników badania sondażowego dotyczącego motywacji w drodze do sukcesu Cel badania: uzyskanie
Bioinformatyczne bazy danych
 Bioinformatyczne bazy danych Czym jest bioinformatyka? Bioinformatyka jest nauką integrującą różne dziedziny wiedzy Gruca (2010) Czym jest bioinformatyka? Bioinformatyka obejmuje technologie wykorzystujące
Bioinformatyczne bazy danych Czym jest bioinformatyka? Bioinformatyka jest nauką integrującą różne dziedziny wiedzy Gruca (2010) Czym jest bioinformatyka? Bioinformatyka obejmuje technologie wykorzystujące
Pytanie: Kiedy do testowania hipotezy stosujemy rozkład normalny?
 Pytanie: Kiedy do testowania hipotezy stosujemy rozkład normalny? Gdy: badana cecha jest mierzalna (tzn. posiada rozkład ciągły); badana cecha posiada rozkład normalny; dysponujemy pojedynczym wynikiem;
Pytanie: Kiedy do testowania hipotezy stosujemy rozkład normalny? Gdy: badana cecha jest mierzalna (tzn. posiada rozkład ciągły); badana cecha posiada rozkład normalny; dysponujemy pojedynczym wynikiem;
weryfikacja hipotez dotyczących parametrów populacji (średnia, wariancja) założenie: znany rozkład populacji (wykorzystuje się dystrybuantę)
 PODSTAWY STATYSTYKI 1. Teoria prawdopodobieństwa i elementy kombinatoryki. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5. Testy parametryczne (na
PODSTAWY STATYSTYKI 1. Teoria prawdopodobieństwa i elementy kombinatoryki. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5. Testy parametryczne (na
Politechnika Wrocławska. BIOINFORMATYKA i BIOLOGIA OBLICZENIOWA
 BIOINFORMATYKA i BIOLOGIA OBLICZENIOWA Kontakt Katedra Inżynierii Biomedycznej Konsultacje D1 pok. 115 Poniedziałek 10.00-12.00 Poniedziałek 15.00-17.00 Małgorzata.Kotulska@pwr.edu.pl Materiały: http://www.kotulska-lab.pwr.wroc.pl/forstudents/
BIOINFORMATYKA i BIOLOGIA OBLICZENIOWA Kontakt Katedra Inżynierii Biomedycznej Konsultacje D1 pok. 115 Poniedziałek 10.00-12.00 Poniedziałek 15.00-17.00 Małgorzata.Kotulska@pwr.edu.pl Materiały: http://www.kotulska-lab.pwr.wroc.pl/forstudents/
PODSTAWY BIOINFORMATYKI 6 BAZA DANYCH NCBI - II
 PODSTAWY BIOINFORMATYKI 6 BAZA DANYCH NCBI - II BAZA DANYCH NCBI 1. NCBI 2. Dane gromadzone przez NCBI 3. Przegląd baz danych NCBI: Publikacje naukowe Projekty analizy genomów OMIM: fenotypy człowieka
PODSTAWY BIOINFORMATYKI 6 BAZA DANYCH NCBI - II BAZA DANYCH NCBI 1. NCBI 2. Dane gromadzone przez NCBI 3. Przegląd baz danych NCBI: Publikacje naukowe Projekty analizy genomów OMIM: fenotypy człowieka
TEORETYCZNE PODSTAWY INFORMATYKI
 1 TEORETYCZNE PODSTAWY INFORMATYKI WFAiS UJ, Informatyka Stosowana I rok studiów, I stopień Wykład 16 2 Data Science: Uczenie maszynowe Uczenie maszynowe: co to znaczy? Metody Regresja Klasyfikacja Klastering
1 TEORETYCZNE PODSTAWY INFORMATYKI WFAiS UJ, Informatyka Stosowana I rok studiów, I stopień Wykład 16 2 Data Science: Uczenie maszynowe Uczenie maszynowe: co to znaczy? Metody Regresja Klasyfikacja Klastering
Analiza korespondencji
 Analiza korespondencji Kiedy stosujemy? 2 W wielu badaniach mamy do czynienia ze zmiennymi jakościowymi (nominalne i porządkowe) typu np.: płeć, wykształcenie, status palenia. Punktem wyjścia do analizy
Analiza korespondencji Kiedy stosujemy? 2 W wielu badaniach mamy do czynienia ze zmiennymi jakościowymi (nominalne i porządkowe) typu np.: płeć, wykształcenie, status palenia. Punktem wyjścia do analizy
( x) Równanie regresji liniowej ma postać. By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : Gdzie:
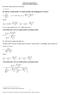 ma postać y = ax + b Równanie regresji liniowej By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : xy b = a = b lub x Gdzie: xy = też a = x = ( b ) i to dane empiryczne, a ilość
ma postać y = ax + b Równanie regresji liniowej By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : xy b = a = b lub x Gdzie: xy = też a = x = ( b ) i to dane empiryczne, a ilość
Statystyczna analiza danych (molekularnych) analiza wariancji ANOVA
 Statystyczna analiza danych (molekularnych) analiza wariancji ANOVA Anna Gambin 19 maja 2013 Spis treści 1 Przykład: Model liniowy dla ekspresji genów 1 2 Jednoczynnikowa analiza wariancji 3 2.1 Testy
Statystyczna analiza danych (molekularnych) analiza wariancji ANOVA Anna Gambin 19 maja 2013 Spis treści 1 Przykład: Model liniowy dla ekspresji genów 1 2 Jednoczynnikowa analiza wariancji 3 2.1 Testy
Globalne zależności w klastrowaniu hierarchicznym
 23 listopada 2008 Plan prezentacji 1 Praca źródłowa Metody Bottom-Up i Top-Down 2 Schemat algorytmu TDQC Preprocessing Algorytm Quantum Clustering 3 Zbiory danych Kryteria porównywania wyników Eksperymenty
23 listopada 2008 Plan prezentacji 1 Praca źródłowa Metody Bottom-Up i Top-Down 2 Schemat algorytmu TDQC Preprocessing Algorytm Quantum Clustering 3 Zbiory danych Kryteria porównywania wyników Eksperymenty
Grafy i sieci wybrane zagadnienia wykład 3: modele służące porównywaniu sieci
 Grafy i sieci wybrane zagadnienia wykład 3: modele służące porównywaniu sieci prof. dr hab. inż. Marta Kasprzak Instytut Informatyki, Politechnika Poznańska Plan wykładu 1. Sieci jako modele interakcji
Grafy i sieci wybrane zagadnienia wykład 3: modele służące porównywaniu sieci prof. dr hab. inż. Marta Kasprzak Instytut Informatyki, Politechnika Poznańska Plan wykładu 1. Sieci jako modele interakcji
Karta (sylabus) modułu/przedmiotu Inżynieria Materiałowa Studia II stopnia Specjalność: Inżynieria Powierzchni
 Karta (sylabus) modułu/przedmiotu Inżynieria Materiałowa Studia II stopnia Specjalność: Inżynieria Powierzchni Przedmiot: Statystyczne Sterowanie Procesami Rodzaj przedmiotu: Obowiązkowy Kod przedmiotu:
Karta (sylabus) modułu/przedmiotu Inżynieria Materiałowa Studia II stopnia Specjalność: Inżynieria Powierzchni Przedmiot: Statystyczne Sterowanie Procesami Rodzaj przedmiotu: Obowiązkowy Kod przedmiotu:
Mutacje jako źródło różnorodności wewnątrzgatunkowej
 Mutacje jako źródło różnorodności wewnątrzgatunkowej Zajęcia terenowe: Zajęcia w klasie: Poziom nauczania oraz odniesienie do podstawy programowej: Liceum IV etap edukacyjny zakres rozszerzony: Różnorodność
Mutacje jako źródło różnorodności wewnątrzgatunkowej Zajęcia terenowe: Zajęcia w klasie: Poziom nauczania oraz odniesienie do podstawy programowej: Liceum IV etap edukacyjny zakres rozszerzony: Różnorodność
Metody Statystyczne. Metody Statystyczne
 #7 1 Czy straszenie jest bardziej skuteczne niż zachęcanie? Przykład 5.2. s.197 Grupa straszona: 8,5,8,7 M 1 =7 Grupa zachęcana: 1, 1, 2,4 M 2 =2 Średnia ogólna M=(M1+M2)/2= 4,5 Wnioskowanie statystyczne
#7 1 Czy straszenie jest bardziej skuteczne niż zachęcanie? Przykład 5.2. s.197 Grupa straszona: 8,5,8,7 M 1 =7 Grupa zachęcana: 1, 1, 2,4 M 2 =2 Średnia ogólna M=(M1+M2)/2= 4,5 Wnioskowanie statystyczne
Wzorcowe efekty kształcenia dla kierunku studiów biotechnologia studia pierwszego stopnia profil ogólnoakademicki
 Załącznik nr 2 do Uchwały Rady Wydziału Biochemii, Biofizyki i Biotechnologii UJ z dnia 19 czerwca 2018 r. w sprawie programu i planu studiów na kierunku BIOTECHNOLOGIA na poziomie studiów pierwszego stopnia
Załącznik nr 2 do Uchwały Rady Wydziału Biochemii, Biofizyki i Biotechnologii UJ z dnia 19 czerwca 2018 r. w sprawie programu i planu studiów na kierunku BIOTECHNOLOGIA na poziomie studiów pierwszego stopnia
Algorytm grupowania danych typu kwantyzacji wektorów
 Algorytm grupowania danych typu kwantyzacji wektorów Wstęp Definicja problemu: Typowe, problemem często spotykanym w zagadnieniach eksploracji danych (ang. data mining) jest zagadnienie grupowania danych
Algorytm grupowania danych typu kwantyzacji wektorów Wstęp Definicja problemu: Typowe, problemem często spotykanym w zagadnieniach eksploracji danych (ang. data mining) jest zagadnienie grupowania danych
KARTA PRZEDMIOTU. WYMAGANIA WSTĘPNE W ZAKRESIE WIEDZY, UMIEJĘTNOŚCI I INNYCH KOMPETENCJI 1. Brak
 WYDZIAŁ PODSTAWOWYCH PROBLEMÓW TECHNIKI Zał. nr 4 do ZW KARTA PRZEDMIOTU Nazwa w języku polskim: Metody statystyczne w badaniu wzroku Nazwa w języku angielskim: Statistical methods in eye research Kierunek
WYDZIAŁ PODSTAWOWYCH PROBLEMÓW TECHNIKI Zał. nr 4 do ZW KARTA PRZEDMIOTU Nazwa w języku polskim: Metody statystyczne w badaniu wzroku Nazwa w języku angielskim: Statistical methods in eye research Kierunek
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
Kamila Muraszkowska Znaczenie wąskich gardeł w sieciach białkowych. źródło: (3)
 Kamila Muraszkowska Znaczenie wąskich gardeł w sieciach białkowych źródło: (3) Interakcje białko-białko Ze względu na zadanie: strukturalne lub funkcjonalne. Ze względu na właściwości fizyczne: stałe lub
Kamila Muraszkowska Znaczenie wąskich gardeł w sieciach białkowych źródło: (3) Interakcje białko-białko Ze względu na zadanie: strukturalne lub funkcjonalne. Ze względu na właściwości fizyczne: stałe lub
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZE W BIOLOGII 1. Wykład wstępny. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. ajczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZE W BIOLOGII 1. Wykład wstępny. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. ajczęściej wykorzystywane testy statystyczne
Druga zasada termodynamiki a modelowanie sieci.
 13 października 2009 O czym będzie mowa? Eksperyment biologiczny eksperyment biologiczny: mikromacierze modelowanie sieci interakcji: II zasada termodynamiki cel: weryfikacja metody metoda symulowania
13 października 2009 O czym będzie mowa? Eksperyment biologiczny eksperyment biologiczny: mikromacierze modelowanie sieci interakcji: II zasada termodynamiki cel: weryfikacja metody metoda symulowania
Statystyczna analiza danych
 Statystyczna analiza danych Testowanie wielu hipotez statystycznych na raz Ewa Szczurek szczurek@mimuw.edu.pl Instytut Informatyki Uniwersytet Warszawski 1/31 Zdechła ryba i emocje http://www.wired.com/2009/09/fmrisalmon/
Statystyczna analiza danych Testowanie wielu hipotez statystycznych na raz Ewa Szczurek szczurek@mimuw.edu.pl Instytut Informatyki Uniwersytet Warszawski 1/31 Zdechła ryba i emocje http://www.wired.com/2009/09/fmrisalmon/
Wykład 9 Testy rangowe w problemie dwóch prób
 Wykład 9 Testy rangowe w problemie dwóch prób Wrocław, 18 kwietnia 2018 Test rangowy Testem rangowym nazywamy test, w którym statystyka testowa jest konstruowana w oparciu o rangi współrzędnych wektora
Wykład 9 Testy rangowe w problemie dwóch prób Wrocław, 18 kwietnia 2018 Test rangowy Testem rangowym nazywamy test, w którym statystyka testowa jest konstruowana w oparciu o rangi współrzędnych wektora
Bioinformatyka. Wykład 2 (12.X.2010) I r. studiów magisterskich, biologia (SGGW) Krzysztof Pawłowski
 Bioinformatyka Wykład 2 (12.X.2010) I r. studiów magisterskich, biologia (SGGW) Krzysztof Pawłowski tydzień temu Co to jest bioinformatyka Sekwencjonowanie genomów historia Metagenomika Wykład 2 spis treści
Bioinformatyka Wykład 2 (12.X.2010) I r. studiów magisterskich, biologia (SGGW) Krzysztof Pawłowski tydzień temu Co to jest bioinformatyka Sekwencjonowanie genomów historia Metagenomika Wykład 2 spis treści
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii
 Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Laboratorium nr 5. Temat: Funkcje agregujące, klauzule GROUP BY, HAVING
 Laboratorium nr 5 Temat: Funkcje agregujące, klauzule GROUP BY, HAVING Celem ćwiczenia jest zaprezentowanie zagadnień dotyczących stosowania w zapytaniach języka SQL predefiniowanych funkcji agregujących.
Laboratorium nr 5 Temat: Funkcje agregujące, klauzule GROUP BY, HAVING Celem ćwiczenia jest zaprezentowanie zagadnień dotyczących stosowania w zapytaniach języka SQL predefiniowanych funkcji agregujących.
Zastosowanie kompresji w kryptografii Piotr Piotrowski
 Zastosowanie kompresji w kryptografii Piotr Piotrowski 1 Plan prezentacji I. Wstęp II. Kryteria oceny algorytmów III. Główne klasy algorytmów IV. Przykłady algorytmów selektywnego szyfrowania V. Podsumowanie
Zastosowanie kompresji w kryptografii Piotr Piotrowski 1 Plan prezentacji I. Wstęp II. Kryteria oceny algorytmów III. Główne klasy algorytmów IV. Przykłady algorytmów selektywnego szyfrowania V. Podsumowanie
Copyright by Wydawnictwo Naukowe Scholar, Warszawa 2000, 2008
 Redaktor: Alicja Zagrodzka Korekta: Krystyna Chludzińska Projekt okładki: Katarzyna Juras Copyright by Wydawnictwo Naukowe Scholar, Warszawa 2000, 2008 ISBN 978-83-7383-296-1 Wydawnictwo Naukowe Scholar
Redaktor: Alicja Zagrodzka Korekta: Krystyna Chludzińska Projekt okładki: Katarzyna Juras Copyright by Wydawnictwo Naukowe Scholar, Warszawa 2000, 2008 ISBN 978-83-7383-296-1 Wydawnictwo Naukowe Scholar
Testy nieparametryczne
 Testy nieparametryczne Testy nieparametryczne możemy stosować, gdy nie są spełnione założenia wymagane dla testów parametrycznych. Stosujemy je również, gdy dane można uporządkować według określonych kryteriów
Testy nieparametryczne Testy nieparametryczne możemy stosować, gdy nie są spełnione założenia wymagane dla testów parametrycznych. Stosujemy je również, gdy dane można uporządkować według określonych kryteriów
Szanowni Organizatorzy Olimpiad Przedmiotowych,
 33757 Warszawa, 11.05.2016 r. Szanowni Organizatorzy Olimpiad Przedmiotowych, Serdecznie dziękujemy za współpracę i Państwa zaangażowanie w przeprowadzenie olimpiad przedmiotowych wśród uczniów szkół podstawowych
33757 Warszawa, 11.05.2016 r. Szanowni Organizatorzy Olimpiad Przedmiotowych, Serdecznie dziękujemy za współpracę i Państwa zaangażowanie w przeprowadzenie olimpiad przedmiotowych wśród uczniów szkół podstawowych
Recenzja rozprawy doktorskiej mgr Łukasza Gadomera pt. Rozmyte lasy losowe oparte na modelach klastrowych drzew decyzyjnych w zadaniach klasyfikacji
 Prof. dr hab. inż. Eulalia Szmidt Instytut Badań Systemowych Polskiej Akademii Nauk ul. Newelska 6 01-447 Warszawa E-mail: szmidt@ibspan.waw.pl Warszawa, 30.04.2019r. Recenzja rozprawy doktorskiej mgr
Prof. dr hab. inż. Eulalia Szmidt Instytut Badań Systemowych Polskiej Akademii Nauk ul. Newelska 6 01-447 Warszawa E-mail: szmidt@ibspan.waw.pl Warszawa, 30.04.2019r. Recenzja rozprawy doktorskiej mgr
Analiza wariancji - ANOVA
 Analiza wariancji - ANOVA Analiza wariancji jest metodą pozwalającą na podział zmienności zaobserwowanej wśród wyników eksperymentalnych na oddzielne części. Każdą z tych części możemy przypisać oddzielnemu
Analiza wariancji - ANOVA Analiza wariancji jest metodą pozwalającą na podział zmienności zaobserwowanej wśród wyników eksperymentalnych na oddzielne części. Każdą z tych części możemy przypisać oddzielnemu
WNIOSEK O WYDANIE ZGODY NA ZAMKNIĘTE UŻYCIE GMO
 WNIOSEK O WYDANIE ZGODY NA ZAMKNIĘTE UŻYCIE GMO 1. Informacje o użytkowniku GMO i osobach odpowiedzialnych za realizację planowanego zamkniętego użycia GMO 1.1 (*) Nazwa i siedziba użytkownika lub imię,
WNIOSEK O WYDANIE ZGODY NA ZAMKNIĘTE UŻYCIE GMO 1. Informacje o użytkowniku GMO i osobach odpowiedzialnych za realizację planowanego zamkniętego użycia GMO 1.1 (*) Nazwa i siedziba użytkownika lub imię,
Politechnika Śląska. Wydział, Automatyki, Elektroniki i Informatyki
 Politechnika Śląska Wydział, Automatyki, Elektroniki i Informatyki Wybrane bazy danych adnotacji funkcjonalnych charakterystyka, opis, analiza jako struktury i powiązań. Analiza jakości oraz popularności
Politechnika Śląska Wydział, Automatyki, Elektroniki i Informatyki Wybrane bazy danych adnotacji funkcjonalnych charakterystyka, opis, analiza jako struktury i powiązań. Analiza jakości oraz popularności
Analizy wariancji ANOVA (analysis of variance)
 ANOVA Analizy wariancji ANOVA (analysis of variance) jest to metoda równoczesnego badania istotności różnic między wieloma średnimi z prób pochodzących z wielu populacji (grup). Model jednoczynnikowy analiza
ANOVA Analizy wariancji ANOVA (analysis of variance) jest to metoda równoczesnego badania istotności różnic między wieloma średnimi z prób pochodzących z wielu populacji (grup). Model jednoczynnikowy analiza
Metody Statystyczne. Metody Statystyczne. #8 Błąd I i II rodzaju powtórzenie. Dwuczynnikowa analiza wariancji
 gkrol@mail.wz.uw.edu.pl #8 Błąd I i II rodzaju powtórzenie. Dwuczynnikowa analiza wariancji 1 Ryzyko błędu - powtórzenie Statystyka niczego nie dowodzi, czyni tylko wszystko mniej lub bardziej prawdopodobnym
gkrol@mail.wz.uw.edu.pl #8 Błąd I i II rodzaju powtórzenie. Dwuczynnikowa analiza wariancji 1 Ryzyko błędu - powtórzenie Statystyka niczego nie dowodzi, czyni tylko wszystko mniej lub bardziej prawdopodobnym
Testy post-hoc. Wrocław, 6 czerwca 2016
 Testy post-hoc Wrocław, 6 czerwca 2016 Testy post-hoc 1 metoda LSD 2 metoda Duncana 3 metoda Dunneta 4 metoda kontrastów 5 matoda Newman-Keuls 6 metoda Tukeya Metoda LSD Metoda Least Significant Difference
Testy post-hoc Wrocław, 6 czerwca 2016 Testy post-hoc 1 metoda LSD 2 metoda Duncana 3 metoda Dunneta 4 metoda kontrastów 5 matoda Newman-Keuls 6 metoda Tukeya Metoda LSD Metoda Least Significant Difference
Synteza i eksploracja danych sekwencyjnych
 Synteza i eksploracja danych sekwencyjnych Definicja problemu i wstępne wyniki eksperymentalne Projekt finansowany z grantu nr DEC-2011/03/D/ST6/01621 otrzymanego z Narodowego Centrum Nauki Plan prezentacji
Synteza i eksploracja danych sekwencyjnych Definicja problemu i wstępne wyniki eksperymentalne Projekt finansowany z grantu nr DEC-2011/03/D/ST6/01621 otrzymanego z Narodowego Centrum Nauki Plan prezentacji
