Zastosowanie analizy filogeograficznej w interpretacji wyników sekwencjonowania mitochondrialnego DNA dla celów sądowych
|
|
|
- Wacława Kaczmarek
- 7 lat temu
- Przeglądów:
Transkrypt
1 ARCH. MED. SĄD. KRYM., 2006, LVI, PRACE ORYGINALNE Tomasz Grzybowski 1, Boris A. Malyarchuk 2, Jarosław Bednarek 1, Marcin Woźniak 1, Marta Papuga 1, Katarzyna Stopińska 1, Sylwia Łuczak 1 Zastosowanie analizy filogeograficznej w interpretacji wyników sekwencjonowania mitochondrialnego DNA dla celów sądowych Phylogeographic approach in the interpretation of mitochondrial DNA sequencing results in forensics 1 Z Zakładu Genetyki Molekularnej i Sądowej Katedry Medycyny Sądowej Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Uniwersytetu Mikołaja Kopernika w Toruniu Kierownik: prof. dr hab. Karol Śliwka 2 Z Laboratorium Genetyki Instytutu Biologicznych Problemów Północy Rosyjskiej Akademii Nauk, Magadan, Rosja W ostatnim czasie badaniom sekwencji mitochondrialnego DNA dla celów sądowych towarzyszy wyraźna perspektywa ewolucyjna. Szczególnie przydatne w interpretacji wyników sekwencjonowania mtdna w sprawach identyfikacyjnych okazuje się podejście filogeograficzne, które obejmuje filogenetyczną analizę przestrzennego rozkładu haplotypów i haplogrup w populacjach. W ramach niniejszej pracy wykazano przydatność podejścia filogeograficznego w interpretacji trudniejszych przypadków identyfikacyjnych, w których obserwowano jednonukleotydowe, homoplazmatyczne różnice sekwencji mtdna pomiędzy haplotypami dowodowymi i porównawczymi. Dodatkowo, w celu ustalenia przynależności haplogrupowej wybranych haplotypów przeprowadzono analizę pełnych sekwencji dwóch genomów mitochondrialnych należących do podkladu J1b. In recent years, forensic mitochondrial DNA analysis has been undertaken from an evolutionary perspective. In particular, the phylogeographic approach based on a phylogenetic analysis of the spatial distribution of mitochondrial haplotypes and haplogroups appears to be a useful tool in the interpretation of identification cases. In this study, the phylogeographic approach has been employed in the analysis of three difficult forensic cases, where single nucleotide, homoplasmic differences were found between the reference and evidentiary haplotypes. mtdna sequence variation has been examined by the control region (HVS I and HVS II) direct sequencing. Additionally, in order to clarify the subhaplogroup status of the selected haplotypes, DNA sequences of entire mitochondrial genomes obtained from two samples representing J1b subclade have been analyzed. WSTĘP Choć analiza sekwencji mitochondrialnego DNA (mtdna) stała się jedną z dobrze ugruntowanych technologii badania polimorfizmu DNA dla potrzeb wymiaru sprawiedliwości [1], niektóre zagadnienia dotyczące interpretacji wyników sekwencjonowania mtdna dla celów sądowych wciąż są przedmiotem ożywionych dyskusji. Do zagadnień tych należą m.in. wielkość, zasięg geograficzny oraz jakość populacyjnych baz danych mtdna wykorzystywanych dla oceny częstości haplotypów, zróżnicowane tempo mutacji dla różnych pozycji sekwencji regionu kontrolnego i związane z nim różne kryteria statystycznej oceny wyników potwierdzających lub wykluczających pochodzenie badanych próbek z tej samej linii matczynej, czy też ograniczona wartość dowodowa analizy sekwencji regionu kontrolnego, a stąd konieczność włączenia do badań wybranych wariantów regionu kodującego [2, 3, 4]. W ostatnim czasie analizom sekwencji mtdna dla celów sądowych towarzyszy wyraźna perspektywa ewolucyjna [2]. W szczególności, dzięki podejściu filogeograficznemu, które obejmuje analizę prze-
2 192 Tomasz Grzybowski, Boris A. Malyarchuk, Jarosław Bednarek, Marcin Woźniak Nr 3 strzennego, geograficznego rozkładu haplotypów w drzewie filogenetycznym mtdna, podjęto próby identyfikacji regionu geograficznego, z którego pochodzą badane haplotypy mtdna oraz a posteriori dokonano kontroli jakości danych zdeponowanych w niektórych sądowych populacyjnych bazach profili mtdna [2 oraz zawarte tam pozycje piśmiennictwa]. Ponieważ rozdzielczość analiz filogenetycznych osiągnęła w ostatnim czasie maksymalny poziom dzięki badaniom populacyjnym wykorzystującym sekwencjonowanie pełnych genomów mitochondrialnych [5 oraz zawarte tam pozycje piśmiennictwa], znacznie łatwiejsze niż dotychczas stały się poszukiwania dodatkowych markerów regionu kodującego, które mogą zwiększyć informatywność analiz mtdna dla celów sądowych. Ponadto, szczegółowa znajomość filogenezy mtdna pozwala na bardziej wiarygodną ocenę częstości haplotypów regionu kontrolnego w populacjach zamieszkujących różne regiony geograficzne. Dane przedstawione w niniejszej pracy ilustrują użyteczność podejścia filogeograficznego w interpretacji trudniejszych przypadków identyfikacyjnych pochodzących z praktyki Zakładu Genetyki Molekularnej i Sądowej, w których zachodziła konieczność analizy mtdna. W przypadkach tych zanotowano jednonukleotydowe, homoplazmatyczne różnice pomiędzy haplotypami uzyskanymi dla próbek dowodowych i porównawczych. Interpretacji wyników dokonano z wykorzystaniem własnych baz danych regionu kontrolnego mtdna szczegółowo scharakteryzowanych pod względem filogeograficznym. Dodatkowo, dla uaktualnienia filogenezy podhaplogrupy J1b, z której pochodziły haplotypy uzyskane w jednym z analizowanych przypadków oraz dla uprawdopodobnienia interpretacji, przeprowadzono analizę sekwencji pełnych genomów mitochondrialnych. MATERIAŁ I METODY Badany materiał biologiczny pochodził z trzech spośród około 225 przypadków, w których Zakład Genetyki Molekularnej i Sądowej w latach wydał ekspertyzy na podstawie badań sekwencji mtdna. Przypadek 1 W sprawie identyfikacyjnej z terenu Małopolski zabezpieczono pięć włosów dowodowych pozbawionych cebulek włosowych (oznaczone numerami 1-5). Materiałem porównawczym były włosy zabezpieczone z odciętej głowy odnalezionej w niedalekiej odległości od miejsca, w którym zabezpieczono włosy dowodowe. W wyniku wstępnej analizy morfologiczno porównawczej przeprowadzonej z wykorzystaniem komputerowego systemu analizy obrazu stwierdzono, że włosy dowodowe mają swe odpowiedniki morfologiczne wśród włosów porównawczych. Do badań mtdna zakwalifikowano wszystkie włosy dowodowe oraz trzy włosy porównawcze (oznaczone jako P1-P3). Przypadek 2 W sprawie zabójstwa mężczyzny dokonanego na terenie Pomorza i Kujaw zabezpieczono cztery włosy dowodowe pozbawione cebulek włosowych, ujawnione na ubraniu ofiary (oznaczone numerami 1-4). Materiałem porównawczym do badań identyfikacyjnych była krew zabezpieczona podczas sekcji zwłok denata (oznaczona jako SER_2). Przypadek 3 W sprawie identyfikacji zwłok nieznanego mężczyzny, który zginął podczas pożaru na terenie Pomorza i Kujaw, zabezpieczono materiał w postaci krwi denata (oznaczony jako NN). Materiałem porównawczym do badań identyfikacyjnych była krew uzyskana od matki osoby zaginionej (oznaczona jako SER_3). MOLEKULARNE ANALIZY mtdna Wszystkie próbki materiału biologicznego poddano badaniom regionu kontrolnego mtdna zgodnie z procedurą opisaną we wcześniejszej pracy [6]. Uzyskane sekwencje mtdna, obejmujące fragment regionu kontrolnego pomiędzy nukleotydem a dla HVS I oraz dla HVS II porównywano z sekwencją wzorcową CRS [7]. Analizie sekwencji pełnych genomów mitochondrialnych poddano próbki pochodzące z przypadku 3 (NN oraz SER_3). Pełne genomy amplifikowano metodą PCR w jedenastu nakładających się fragmentach, a następnie sekwencjonowano za pomocą 32 opisanych poprzednio wewnętrznych starterów, w warunkach podanych we wcześniejszej pracy [6]. Uzyskane sekwencje mtdna porównywano z poprawioną sekwencją wzorcową rcrs [8]. FILOGENETYCZNE I STATYSTYCZNE ANALIZY mtdna Drzewo filogenetyczne pełnych genomów mitochondrialnych zrekonstruowano manualnie metodą największej oszczędności i ukorzeniono za pomocą sekwencji rcrs [8] jako grupy zewnętrznej,
3 Nr 3 ANALIZA FILOGEOGRAFICZNA 193 a następnie sprawdzono z wykorzystaniem oprogramowania Network v (A. Röhl; Fluxus Technology; W rekonstrukcji drzewa wykorzystano sekwencję pełnego genomu mitochondrialnego należącego do podhaplogrupy J1b, opublikowanego przez Palanichamy i wsp. [9]. Interpretację statystyczną wyników analizy mtd- NA dla celów sądowych przedstawiano za pomocą ilorazu prawdopodobieństw warunkowych (LR) = P (E/H1) / P (E/H2), gdzie P (E/H1) oznacza prawdopodobieństwo uzyskania wyniku przy założeniu, że materiał dowodowy pochodzi z linii matczynej reprezentowanej przez próbkę referencyjną, zaś P (E/H2) jest prawdopodobieństwem uzyskania wyniku przy założeniu, że próbka dowodowa pochodzi z innej linii matczynej. W przypadku pełnej zgodności sekwencji pomiędzy próbką dowodową i porównawczą, P (E/H1) = 1. W przypadku zaistnienia jednonukleotydowej różnicy pomiędzy sekwencją próbki dowodowej i porównawczej, P (E/H1) ma postać iloczynu częstości mutacji w pozycji sekwencji różniącej oba haplotypy oraz tempa mutacji w regionie kontrolnym w czasie jednego pokolenia. Z uwagi na znaczną wewnątrzosobniczą niestabilność szlaków polipirymidynowych regionów HVS I i HVS II oraz związaną z tym ograniczoną wartość identyfikacyjną mutacji w tych regionach, w analizach statystycznych pomijano polimorfizmy długości notowane w pozycjach oraz Częstość substytucji w pozycjach 16357, oraz obliczano na podstawie ilorazu liczby mutacji obserwowanych w tych pozycjach w bazie danych i całkowitej liczby mutacji w tej samej bazie (7800). W obliczeniach wykorzystano własną bazę danych haplotypów HVS I i HVS II (n=1465) z obszaru zachodniej Eurazji (z populacji polskiej, rosyjskiej, bośniackiej i słoweńskiej) [6, 10, 11]. Wszystkie haplotypy umieszczone w bazie zaliczono do odpowiednich haplogrup i podhaplogrup mtdna na podstawie syntetycznego systemu klasyfikacji, uwzględniającego dotychczasowe dane uzyskane metodą PCR-RFLP oraz informacje pochodzące z sekwencjonowania regionów HVS I i HVS II. Uśrednione tempo mutacji w regionie kontrolnym rzędu w czasie jednego pokolenia (lub 0.32 na pozycję sekwencji w czasie miliona lat) przyjęto za Sigurðardóttir i wsp. [12]. P (E/H2) obliczano na podstawie liczby obserwacji haplotypu w bazie danych (w przypadku jednonukleotydowej różnicy pomiędzy próbką dowodową i porównawczą była to suma liczby obserwacji haplotypu dowodowego oraz haplotypu różniącego się od dowodowego jedną mutacją). W sytuacji braku haplotypu w bazie danych, w obliczeniach wykorzystywano częstość najmniejszej (najmłodszej ewolucyjnie, czyli znajdującej się na obrzeżach drzewa filogenetycznego) podhaplogrupy, do której można było zaliczyć uzyskany haplotyp, zgodnie z sugestią Forstera i wsp. [13]. W obliczeniach częstości rzadkich haplotypów na podstawie przeszukiwania bazy danych stosowano przedział ufności 95 %, z wykorzystaniem logarytmu naturalnego częstości, przybliżenia normalnego do rozkładu dwumianowego oraz antylogarytmu [1, 14]. Dla zachowania ostrożności, w obliczeniach LR wykorzystywano maksymalne częstości haplotypów (z górnej granicy przedziału ufności). WYNIKI I OMÓWIENIE Haplotypy regionu kontrolnego mtdna uzyskane dla próbek materiału dowodowego i porównawczego oraz ich przynależność haplogrupową przedstawiono w tabeli I. Z przedstawionych danych wynika, że pomiędzy sekwencjami mtdna uzyskanymi z włosa dowodowego oznaczonego numerem 4, a sekwencją otrzymaną dla włosów porównawczych analizowanych w Przypadku 1 wystąpiły różnice w postaci dodatkowej tranzycji T16357C oraz insercji 309.1C. Pozostałe haplotypy uzyskane dla włosów dowodowych wykazywały pełną zgodność wobec haplotypu porównawczego (tabela I). Uzyskane haplotypy należą do podhaplogrupy J1c zdefiniowanej poprzez tranzycje w pozycjach 228 i [9], najczęstszego podkladu w obrębie haplogrupy J w populacjach słowiańskich oraz w innych populacjach europejskich [6]. Haplotypów należących do J1c z tranzycjami w pozycjach i nie zanotowano jednak do tej pory w bazie danych [6, 10, 11]. Na podstawie znajomości topologii drzewa filogenetycznego haplogrupy J1 [6] można stwierdzić, że najmniejsza podgrupa w obrębie J1c, do której można zaliczyć haplotypy uzyskane w omawianym przypadku obejmuje sekwencje z motywem regionu kontrolnego , a jej częstość w bazie danych wynosi 3/1465, czyli x 10-3 (wartość będąca górną granicą przedziału ufności). Wartość LR dla obserwacji pełnej zgodności pomiędzy haplotypami dowodowymi i porównawczymi w przypadku pierwszym wynosi 486. Dla obserwacji jednonukleotydowej różnicy pomiędzy haplotypem uzyskanym z włosa numer 4 a haplotypem porównawczym (tabela I), dokonano oceny częstości substytucji T16357C z wykorzystaniem spektrum mutacji regionu kontrolnego obserwowanego w bazie danych. Zanotowano tutaj dwa przypadki substytucji w pozycji na
4 194 Tomasz Grzybowski, Boris A. Malyarchuk, Jarosław Bednarek, Marcin Woźniak Nr 3 Tabela I. Haplotypy regionu kontrolnego mtdna (HVS I i HVS II) oraz ich przynależność haplogrupowa w materiale dowodowym i porównawczym. Mutacje identyfikowano względem sekwencji wzorcowej CRS [7]. Brak zasady przy odpowiedniej pozycji sekwencji oznacza tranzycję. Insercje reszt cytozyny oznaczono przyrostkami.1c. Table I. mtdna control region haplotypes and their subhaplogroup assignment in the evidentiary and reference samples. Mutations are shown indicating positions relative to the mtdna Cambridge reference sequence (CRS) [7]. The nucleotide positions correspond to transitions; insertions of cytosines are referred to by.1c following the nucleotide position. Przypadek Nazwa próbki Podhaplogrupa Sekwencja regionu kontrolnego mtdna HVS I HVS II 1 1 J1c C C C C 315.1C C P C P C P C 2 1 H C 315.1C C 315.1C C 315.1C C 315.1C SER_ C 315.1C 3 NN J1b C SER_ C tle haplogrup R* oraz U5a [10, 11]. Warto jednak zwrócić uwagę, że pozycja uważana jest za gorące miejsce mutacji w regionie kontrolnym mtdna, co uzyskało potwierdzenie w co najmniej dwóch niezależnych analizach filogenetycznych [15, 16]. Oznacza to, że substytucja T16357C może być obserwowana na tle różnych haplogrup [16]. Prawdopodobieństwo pojawienia się mutacji w pozycji wynosi zatem 2/7800, czyli 2.5 x Przyjmując uśrednione tempo mutacji w czasie jednego pokolenia rzędu , P (E/H1) dla obserwacji różnicy jednonukleotydowej pomiędzy próbką dowodową i porównawczą wynosi 1 x 10-6, a wartość LR w omawianym przypadku równa jest tylko 4.9 x Innymi słowy, przy zaistnieniu różnicy jednonukleotydowej w pozycji 16357, pochodzenie próbki dowodowej z innej linii matczynej niż próbka porównawcza jest ok razy bardziej prawdopodobne niż pochodzenie tych próbek z tej samej linii. Zakładając, że mutacja T16304C ma charakter monofiletyczny w obrębie haplogrupy H [6] można uznać, że haplotypy obserwowane w Przypadku 2 należą do podkladu H5, zdefiniowanego poprzez tranzycje w pozycjach 456 i [17]. H jest drugim co do częstości motywem wśród haplotypów HVS I należących do haplogrupy H w Europie (ok. 4 %), choć występuje również na Bliskim Wschodzie, z częstościami rzędu 1-3 % [18]. W bazie danych odnaleziono jednak tylko jeden haplotyp z motywem H (częstość profilu w bazie 6.8 x 10-4 ). Mimo to pozycja uważana jest za jedno z najszybszych miejsc w regionie kontrolnym [15, 16, 19]. Przykładowo, w wyniku populacyjnych badań mtdna Polaków i Rosjan stwierdzono równoległe występowanie tranzycji G16129A w haplotypach należących do haplogrup H, U5, T*, T1, I, W oraz M* [10]. Na podstawie liczby obserwacji tej mutacji w bazie danych (22/7800) określono częstość substytucji w pozycji na 2.82 x Wartości LR dla obserwacji pełnej zgodności (próbki oznaczone jako 1 i 2, tabela I) oraz jednonukleotydowej różnicy (próbki oznaczone numerami 3 i 4, tabela I) pomiędzy haplotypami dowodowym i porównawczym wynoszą odpowiednio 1470 oraz x 10-4, a zatem w tym drugim przypadku dowód genetyczny wskazuje aż na ok krotnie większe prawdopodobieństwo pochodzenia haplotypów z różnych linii matczynych. W wyniku sekwencjonowania regionów HVS I i HVS II dwóch próbek badanych w ramach Przypadku 3 stwierdzono, że uzyskane haplotypy, z motywem należą najprawdopodobniej do niezwykle rzadkiego
5 Nr 3 ANALIZA FILOGEOGRAFICZNA 195 Ryc. 1. Drzewo filogenetyczne podhaplogrupy J1b utworzone metodą największej oszczędności, zrekonstruowane z wykorzystaniem sekwencji pełnych genomów mitochondrialnych i ukorzenione za pomocą poprawionej sekwencji referencyjnej (rcrs) [8] jako grupy zewnętrznej. Sekwencje uzyskane w ramach niniejszej pracy opisano jako NN i SER_3. Sekwencję mtdna uzyskaną przez Palanichamy i wsp. [9] z populacji Indii oznaczono jako MGP#R80. Gałęzie drzewa oznaczono za pomocą pozycji nukleotydowych sekwencji mtdna, według numeracji zastosowanej dla rcrs [8]. Brak zasady obok pozycji sekwencji oznacza tranzycję. Z analizy wyłączono polimorfizmy długości towarzyszące pozycjom 309 i 315 regionu HVS II. Fig. 1. The most parsimonious tree of the complete subhaplogroup J1b mtdna sequences, rooted with the revised Cambridge reference sequence (rcrs) [8] as an outgroup. The tree includes 3 mtdnas, out of which two are novel ( NN and SER_3] and one (from the Indian population, designated as MGP#R80 ) was reported by Palanichamy et al. [9]. Mutations are shown on the branches and are transitions. The hypervariable nucleotides (the indels) at nucleotides 309 and 315 in HVS II were excluded. w populacjach europejskich podkladu w obrębie J1b. Przypuszczenie takie oparte jest na obserwacji mutacji C271T w HVS II, przy jednoczesnym braku tranzycji w pozycji 242, diagnostycznej dla podhaplogrupy J1b1 [9]. Do chwili obecnej opublikowano tylko jedną pełną sekwencję genomu mitochondrialnego należącego do J1b z tranzycją w pozycji 271 (z populacji Indii) [9]. W celu dokładnego określenia statusu filogenetycznego haplotypów uzyskanych w Przypadku 3, przeprowadzono analizę sekwencji pełnych genomów mitochondrialnych próbek oznaczonych jako NN oraz SER_3, po czym dokonano rekonstrukcji filogenezy podhaplogrupy J1b w wykorzystaniem nowych danych (rycina 1). Analiza sekwencji wykazała, że pełne genomy mitochondrialne próbki dowodowej i porównawczej różnią się tylko w pozycji Z drzewa filogenetycznego przedstawionego na rycinie 1 wynika, że genomy analizowane w Przypadku 3 należą do podhaplogrupy J1b2, zdefiniowanej poprzez tranzycje w pozycjach 271 i Należy zwrócić uwagę, że taka definicja może mieć charakter tymczasowy, gdyż włączenie do analizy kolejnych danych o sekwencjach pełnych genomów może zmienić topologię drzewa filogenetycznego. W szczególności warto zauważyć, że pozycja uważana jest za kolejne gorące miejsce mutacji. Częstość podhaplogrupy J1b2 jest w chwili obecnej trudna do ustalenia, gdyż wiele zespołów badawczych nie uwzględnia w swych badaniach populacyjnych sekwencji regionu HVS II. We wcześniejszych badaniach własnych zidentyfikowano haplotypy pozbawione tranzycji w pozycji 242, a jednocześnie posiadające mutację C271T (lub jej pozbawione) w populacjach Bliskiego Wschodu (Persowie z Iranu) oraz południowo-zachodniej i południowo-wschodniej Syberii (Ałtajczycy, Buriaci) [6]. Najwyższe częstości J1b2 zanotowano u Persów z Iranu (ok. 3.7 %) [6], jednak w populacji tej wszystkie haplotypy należące do J1b2 były pozbawione tranzycji C1622T, zanotowanej w próbkach badanych w ramach Przypadku 3 (tabela I). Biorąc pod uwagę całokształt przytoczonych danych filogeograficznych należy się spodziewać, że podhaplogrupa J1b2 osiąga najwyższe częstości i wewnętrzne zróżnicowanie w populacjach północnozachodniego Iranu. Zważywszy jednak na niewielką liczebność baz danych sekwencji HVS I i HVS II z tego regionu, ich zastosowanie do obliczeń częstości haplotypów w sprawach sądowych nie wydaje się stosowne. Najmniejszym podkladem, do którego można zaliczyć haplotypy uzyskane w ramach Przypadku 3 jest zatem podhaplogrupa J1b. Częstość tego podkladu w bazie danych wynosi 17/ 1465, czyli 1.2 x Mutacja G16319A należy do
6 196 Tomasz Grzybowski, Boris A. Malyarchuk, Jarosław Bednarek, Marcin Woźniak Nr 3 gorących miejsc w regionie kontrolnym, a jej częstość oszacowano na 22/7800, czyli 2.8 x LR dla obserwacji poczynionych w ramach Przypadku 3 wynosi 0.001, czyli 1000-krotnie faworyzuje hipotezę wykluczającą pochodzenie próbek NN i SER_3 z tej samej linii matczynej. W przypadku zaistnienia pełnej zgodności pomiędzy haplotypami dowodowym i porównawczym, LR wynosiłby 83. W omawianym przypadku przeprowadzono dodatkowo badania piętnastu jądrowych mikrosatelitów. Wyniki tych badań nie pozwoliły na wykluczenie macierzyństwa (MI = , dane nie prezentowane). PODSUMOWANIE Przedstawione dane ilustrują przydatność podejścia filogeograficznego w interpretacji wyników sekwencjonowania mtdna dla celów sądowych. Podejście takie jest szczególnie użyteczne w ocenach częstości bardzo rzadkich haplotypów, których nie obserwowano do tej pory w bazach danych. W takich przypadkach ustalanie częstości najmniejszych podkladów obejmujących rzadkie haplotypy może stanowić alternatywę dla oceny maksymalnego, teoretycznego progu częstości danego haplotypu w populacji z wykorzystaniem tzw. przedziału ufności z proporcji zero [1]. Jak wykazano w niniejszej pracy, w niektórych przypadkach dla dokładnego ustalenia przynależności haplogrupowej uzyskanego haplotypu można się uciec do analizy sekwencji pełnych genomów, pełnych sekwencji regionu kodującego lub chociażby wybranych polimorfizmów regionu kodującego. We wszystkich analizowanych przypadkach niezgodności jednonukleotydowych pomiędzy haplotypami dowodowymi i porównawczymi uzyskiwano bardzo niskie wartości LR, faworyzujące hipotezę wykluczającą pochodzenie próbek z tej samej linii matczynej. Nie upoważnia to jednak do sformułowania ogólnej reguły interpretacji, według której obserwacja różnicy jednonukleotydowej prowadziłaby do jednoznacznie wykluczającej sentencji opinii. Warto zwrócić uwagę, że wszystkie pozycje sekwencji, w których zaobserwowano różnice należą do szybkich (16357) lub bardzo szybkich (16319, 16129) miejsc w regionie kontrolnym. Substytucje w pozycjach i powstają najprawdopodobniej na nici H na skutek przejściowego, nieprawidłowego połączenia startera i matrycy podczas replikacji mtdna przez mitochondrialną polimerazę gamma [16, 19]. Warto zwrócić uwagę, że ten sam mechanizm molekularny wyjaśnia powstawanie polimorfizmu długości w superzmiennych regionach polipirymidynowych HVS I i HVS II, a zmienność tego rodzaju zdaje się nie posiadać większego znaczenia identyfikacyjnego. Ponadto nie można wykluczyć, że przedstawione przypadki homoplazmatycznych różnic są w istocie przypadkami heteroplazmii, niemożliwych do wykrycia z uwagi na niską czułość metody bezpośredniego sekwencjonowania produktów PCR. Dokonane w ramach pracy obliczenia częstości mutacji właściwych dla poszczególnych pozycji regionu kontrolnego wymagają przyjęcia pewnych wstępnych założeń, które mogą być poddane dyskusji. Przykładowo, do tej pory nie ustalono w sposób definitywny przyczyn rozbieżności między częstościami mutacji w regionie kontrolnym obliczanymi na podstawie rekonstrukcji filogenetycznych oraz w wyniku kalkulacji bezpośrednich, poprzez badania mtdna w rodzinach [20]. Szczegółowe omówienie tego zagadnienia przekraczałoby ramy niniejszej pracy. Należy jednak zaznaczyć, że dla potrzeb interpretacji wyników w sprawach sądowych bardziej stosowne wydaje się przyjęcie założeń co do średniego tempa mutacji wyznaczonego na podstawie badań bezpośrednich. W badaniach takich preferencyjnie wykrywane są mutacje w gorących miejscach, które w rekonstrukcjach filogenetycznych mogą pozostać niezauważone bądź niedoszacowane z uwagi na możliwe przypadki rewersji, tj. powrotu danej pozycji sekwencji do stanu wyjściowego [20]. Dla potrzeb niniejszej pracy przyjęto wartość średniego tempa mutacji wyznaczonego w wyniku jednego z szerszych badań rodzinnych [12]. Wartość ta jest porównywalna z wynikami uzyskanymi w innych badaniach bezpośrednich [12]. Obliczenia częstości mutacji w konkretnych pozycjach regionu kontrolnego wymagają również wykorzystania baz danych o odpowiedniej liczebności oraz rozpoznania, czy dana mutacja nie występuje preferencyjnie w obrębie określonej haplogrupy. Substytucje T16357C, G16319A oraz G16129A zanotowano w obrębie wielu różnych haplogrup, toteż w obliczeniach wykorzystano liczbę mutacji obserwowanych w tych pozycjach w stosunku do całkowitej liczby mutacji w bazie danych. W przypadkach różnic jednonukleotydowych podejście takie ma charakter bardzo konserwatywny i prowadzi do znaczącego obniżenia wartości LR, przez co silnie faworyzuje hipotezę wykluczającą pochodzenie próbek z jednej linii matczynej. Należy się spodziewać, że wzrost liczebności baz danych mtdna oraz ich bardziej szczegółowa charakterystyka filogenetyczna pozwolą na dokonanie bardziej precyzyjnych obliczeń częstości mutacji w poszczególnych pozycjach regionu kontrolnego, a także bardziej wiarygodnych ocen częstości haplotypów. W szczególności wzrastająca ilość da-
7 Nr 3 ANALIZA FILOGEOGRAFICZNA 197 nych populacyjnych dotyczących sekwencji pełnych genomów przyczyni się do jeszcze szerszego wykorzystania podejścia filogeograficznego w genetyce sądowej, przede wszystkim w trudniejszych przypadkach identyfikacyjnych. PIŚMIENNICTWO 1. Holland M. M., Parsons T. J.: Mitochondrial DNA sequence analysis validation and use for forensic casework. Forensic Sci. Rev. 1999, 11, Salas A., Bandelt H. J., Macaulay V., Richards M.: Phylogeographic investigations: the role of trees in forensic genetics. Forensic Sci. Int. 2006, w druku. 3. Coble M. D., Vallone P. M., Just R. S., Diegoli T. M., Smith B. C., Parsons T. J.: Effective strategies for forensic analysis in the mitochondrial DNA coding region. Int. J. Legal Med. 2006, 120, Brandstätter A., Salas A., Niederstätter H., Gassner C., Carracedo A., Parson W.: Dissection of mitochondrial superhaplogroup H using coding region SNPs. Electrophoresis 2006, 27, w druku. 5. Torroni A., Achilli A., Macaulay V., Richards M., Bandelt H. J.: Harvesting the fruit of the human mtdna tree. Trends Genet. 2006, 22, Grzybowski T.: Zróżnicowanie sekwencji mitochondrialnego DNA w populacjach ludzkich północnej Eurazji. Wybrane aspekty filogeograficzne. Uniwersytet Mikołaja Kopernika w Toruniu, Collegium Medicum im. Ludwika Rydygiera, Bydgoszcz, Anderson S., Bankier A. T., Barell B. G., de Bruijn M. H., Coulson A. R., Drouin J., Eperon I. C., Nierlich D. P., Roe B. A., Sanger F. i wsp.: Sequence and organization of the human mitochondrial genome. Nature 1981, 290, Andrews R. M., Kubacka I., Chinnery P. F., Lightowlers R. N., Turnbull D. M., Howell N.: Reanalysis and revision of the Cambridge reference sequence for human mitochondrial DNA. Nat. Genet. 1999, 23, Palanichamy M. G., Sun C., Agrawal S., Bandelt H. J., Kong Q. P., Khan F., Wang C. Y., Chaudhuri T. K., Palla V., Zhang Y. P.: Phylogeny of mitochondrial DNA macrohaplogroup N in India, based on complete sequencing: implications for the peopling of South Asia. Am. J. Hum. Genet. 2004, 75, Malyarchuk B. A., Grzybowski T., Derenko M. V., Czarny J., Woźniak M., Miścicka-Śliwka D.: Mitochondrial DNA variability in Poles and Russians. Ann. Hum. Genet. 2002, 66, Malyarchuk B. A., Grzybowski T., Derenko M. V., Czarny J., Drobnic K., Miścicka-Śliwka D.: Mitochondrial DNA variability in Bosnians and Slovenians. Ann. Hum. Genet. 2003, 67, Sigurðardóttir S., Helgason A., Gulcher J. R., Stefansson K., Donnelly P.: The mutation rate in the human mtdna control region. Am. J. Hum. Genet. 2000, 66, Forster P., Cali F., Rohl A., Metspalu E., D Anna R., Mirisola M., De Leo G., Flugy A., Salerno A., Ayala G. i wsp.: Continental and subcontinental distributions of mtdna control region types. Int. J. Legal Med. 2002, 116, National Research Council: The Evaluation of Forensic DNA Evidence. National Academy Press, Washington D.C Bandelt H. J., Quintana-Murci L., Salas A., Macaulay V.: The fingerprint of phantom mutations in mitochondrial DNA data. Am. J. Hum. Genet. 2002, 71, Malyarchuk B. A., Rogozin I. B.: Mutagenesis by transient misalignment in the human mitochondrial DNA control region. Ann. Hum. Genet. 2004, 68, Loogväli E. L., Roostalu U., Malyarchuk B. A., Derenko M. V., Kivisild T., Metspalu E., Tambets K., Reidla M., Tolk H. V., Parik J. i wsp.: Disuniting uniformity: a pied cladistic canvas of mtdna haplogroup H in Eurasia. Mol. Bio.l Evol. 2004, 21, Richards M., Macaulay V., Hickey E., Vega E., Sykes B., Guida V., Rengo C., Sellito D., Cruciani F., Kivisild T. i wsp.: Tracing European founder lineages in the Near Eastern mtdna pool. Am. J. Hum. Genet. 2000, 67, Malyarchuk B. A., Rogozin I. B., Berikov V. B., Derenko M. V.: Analysis of phylogenetically reconstructed mutational spectra in human mitochondrial DNA control region. Hum. Genet. 2002, 111, Howell N., Smejkal C. B., Mackey D. A., Chinnery P. F., Turnbull D. M., Herrnstadt C.: The pedigree rate of sequence divergence in the human mitochondrial genome: there is a difference between phylogenetic and pedigree rates. Am. J. Hum. Genet. 2003, 72, Adres do korespondencji: Dr Tomasz Grzybowski Katedra Medycyny Sądowej Zakład Genetyki Molekularnej i Sądowej Collegium Medicum UMK ul. M. Curie-Skłodowskiej Bydgoszcz Tel: tgrzyb@cm.umk.pl
Standardowa analiza mtdna w badaniach genetyczno-sądowych
 ARCH. MED. SĄD. KRYMINOL., 2012, LXII, 213-218 PRACE KAZUISTYCZNE / CASE REPORTS Katarzyna Skonieczna, Jarosław Bednarek, Urszula Rogalla, Marcin Woźniak, Marta Gorzkiewicz, Katarzyna Linkowska, Anna Duleba,
ARCH. MED. SĄD. KRYMINOL., 2012, LXII, 213-218 PRACE KAZUISTYCZNE / CASE REPORTS Katarzyna Skonieczna, Jarosław Bednarek, Urszula Rogalla, Marcin Woźniak, Marta Gorzkiewicz, Katarzyna Linkowska, Anna Duleba,
Polimorfizm genu mitochondrialnej polimerazy gamma (pol γ) w populacjach ludzkich Europy
 Polimorfizm genu mitochondrialnej polimerazy gamma (pol γ) w populacjach ludzkich Europy Praca wykonana pod kierunkiem dr hab. Tomasza Grzybowskiego w Katedrze Medycyny Sądowej w Zakładzie Genetyki Molekularnej
Polimorfizm genu mitochondrialnej polimerazy gamma (pol γ) w populacjach ludzkich Europy Praca wykonana pod kierunkiem dr hab. Tomasza Grzybowskiego w Katedrze Medycyny Sądowej w Zakładzie Genetyki Molekularnej
Ocena stopnia wysycenia bazy danych mitochondrialnego DNA dla populacji Polski* Saturation of the Polish mitochondrial DNA database
 ARCH. MED. SĄD. KRYMINOL., 2010, LX, 263-269 PRACE ORYGINALNE / ORIGINALS Patrycja Daca, Marta Mielnik-Sikorska, Jarosław Bednarek, Tomasz Grzybowski Ocena stopnia wysycenia bazy danych mitochondrialnego
ARCH. MED. SĄD. KRYMINOL., 2010, LX, 263-269 PRACE ORYGINALNE / ORIGINALS Patrycja Daca, Marta Mielnik-Sikorska, Jarosław Bednarek, Tomasz Grzybowski Ocena stopnia wysycenia bazy danych mitochondrialnego
Zastosowanie reakcji minisekwencjonowania do oznaczania przynależności haplogrupowej mitochondrialnego DNA
 ARCH. MED. SĄD. KRYM., 2008, LVIII, 212-217 prace poglądowe Patrycja Daca, Marta Mielnik, Urszula Rogalla, Katarzyna Skonieczna, Katarzyna Linkowska, Tomasz Grzybowski Zastosowanie reakcji minisekwencjonowania
ARCH. MED. SĄD. KRYM., 2008, LVIII, 212-217 prace poglądowe Patrycja Daca, Marta Mielnik, Urszula Rogalla, Katarzyna Skonieczna, Katarzyna Linkowska, Tomasz Grzybowski Zastosowanie reakcji minisekwencjonowania
Mitochondrialna Ewa;
 Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Wprowadzenie do genetyki sądowej. Materiały biologiczne. Materiały biologiczne: prawidłowe zabezpieczanie śladów
 Wprowadzenie do genetyki sądowej 2013 Pracownia Genetyki Sądowej Katedra i Zakład Medycyny Sądowej Materiały biologiczne Inne: włosy z cebulkami, paznokcie możliwa degradacja - tkanki utrwalone w formalinie/parafinie,
Wprowadzenie do genetyki sądowej 2013 Pracownia Genetyki Sądowej Katedra i Zakład Medycyny Sądowej Materiały biologiczne Inne: włosy z cebulkami, paznokcie możliwa degradacja - tkanki utrwalone w formalinie/parafinie,
Polimorfizm. sekwencji mitochondrialnego DNA gatunku Canis familiaris - aspekty filogenetyczne i identyfikacyjne. Anna Czarnecka
 Uniwersytet Mikołaja Kopernika w Toruniu Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Polimorfizm sekwencji mitochondrialnego DNA gatunku Canis familiaris - aspekty filogenetyczne i identyfikacyjne
Uniwersytet Mikołaja Kopernika w Toruniu Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Polimorfizm sekwencji mitochondrialnego DNA gatunku Canis familiaris - aspekty filogenetyczne i identyfikacyjne
Wprowadzenie do genetyki medycznej i sądowej
 Genetyka medyczno-sądowa Wprowadzenie do genetyki medycznej i sądowej Kierownik Pracowni Genetyki Medycznej i Sądowej Ustalanie tożsamości zwłok Identyfikacja sprawców przestępstw Identyfikacja śladów
Genetyka medyczno-sądowa Wprowadzenie do genetyki medycznej i sądowej Kierownik Pracowni Genetyki Medycznej i Sądowej Ustalanie tożsamości zwłok Identyfikacja sprawców przestępstw Identyfikacja śladów
prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie
 prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie Sekwencyjność występowania zaburzeń molekularnych w niedrobnokomórkowym raku płuca
prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie Sekwencyjność występowania zaburzeń molekularnych w niedrobnokomórkowym raku płuca
Genetyka sądowa. Wydział Lekarski III, IV, V, VI. fakultatywny. Dr n. med. Magdalena Konarzewska
 Genetyka sądowa 1. Metryczka Nazwa Wydziału: Program kształcenia: Wydział Lekarski Lekarski, jednolite magisterskie, profil praktyczny, stacjonarne Rok akademicki: 2018/2019 Nazwa modułu/przedmiotu: Genetyka
Genetyka sądowa 1. Metryczka Nazwa Wydziału: Program kształcenia: Wydział Lekarski Lekarski, jednolite magisterskie, profil praktyczny, stacjonarne Rok akademicki: 2018/2019 Nazwa modułu/przedmiotu: Genetyka
Zasady atestacji laboratoriów genetycznych przy Polskim Towarzystwie Medycyny Sądowej i Kryminologii na lata
 ARCH. MED. SĄD. KRYM., 2007, LVII, 368-379 różne Komisja Genetyki Sądowej Polskiego Towarzystwa Medycyny Sądowej i Kryminologii Przewodnicząca: prof. dr hab. n. med. Zofia Szczerkowska (Gdańsk) członkowie:
ARCH. MED. SĄD. KRYM., 2007, LVII, 368-379 różne Komisja Genetyki Sądowej Polskiego Towarzystwa Medycyny Sądowej i Kryminologii Przewodnicząca: prof. dr hab. n. med. Zofia Szczerkowska (Gdańsk) członkowie:
Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi.
 Ryciny 193 Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi. Na fioletowo zaznaczone zostały populacje (nr 1 14)
Ryciny 193 Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi. Na fioletowo zaznaczone zostały populacje (nr 1 14)
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI
 ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI Joanna Szyda Magdalena Frąszczak Magda Mielczarek WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI Joanna Szyda Magdalena Frąszczak Magda Mielczarek WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
Zabezpieczanie próbek biologicznych i rejestracja profili w Bazie Danych DNA
 Zabezpieczanie próbek biologicznych i rejestracja profili w Bazie Danych DNA w przypadku: PROWADZENIA POSZUKIWAŃ OSOBY ZAGINIONEJ I. PRZEPISY OKREŚLAJĄCE OBOWIĄZKI POLICJANTÓW ORAZ ZASADY POSTĘPOWANIA
Zabezpieczanie próbek biologicznych i rejestracja profili w Bazie Danych DNA w przypadku: PROWADZENIA POSZUKIWAŃ OSOBY ZAGINIONEJ I. PRZEPISY OKREŚLAJĄCE OBOWIĄZKI POLICJANTÓW ORAZ ZASADY POSTĘPOWANIA
Polskie Towarzystwo Medycyny Sądowej i Kryminologii
 KOMUNIKAT Komisja Genetyki Sądowej Polskiego Towarzystwa Medycyny Sądowej i Kryminologii informuje, że został rozpoczęty proces atestacji na badania DNA w Polsce dla celów sądowych. Przesłanie próbek do
KOMUNIKAT Komisja Genetyki Sądowej Polskiego Towarzystwa Medycyny Sądowej i Kryminologii informuje, że został rozpoczęty proces atestacji na badania DNA w Polsce dla celów sądowych. Przesłanie próbek do
Porównywanie i dopasowywanie sekwencji
 Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI
 ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI JOANNA SZYDA MAGDALENA FRĄSZCZAK MAGDA MIELCZAREK WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI JOANNA SZYDA MAGDALENA FRĄSZCZAK MAGDA MIELCZAREK WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
LABORATORIUM KRYMINALISTYCZNE KSP SEKCJA VI - BIOLOGII I OSMOLOGII. Strona znajduje się w archiwum.
 LABORATORIUM KRYMINALISTYCZNE KSP Źródło: http://laboratorium.policja.waw.pl/lk/sekcje/sekcja-vi-biologii-i-os/2367,sekcja-vi-biologii-i-osmologii.html Wygenerowano: Piątek, 6 stycznia 2017, 20:57 Strona
LABORATORIUM KRYMINALISTYCZNE KSP Źródło: http://laboratorium.policja.waw.pl/lk/sekcje/sekcja-vi-biologii-i-os/2367,sekcja-vi-biologii-i-osmologii.html Wygenerowano: Piątek, 6 stycznia 2017, 20:57 Strona
Konstruowanie drzew filogenetycznych. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Wnioskowanie bayesowskie
 Wnioskowanie bayesowskie W podejściu klasycznym wnioskowanie statystyczne oparte jest wyłącznie na podstawie pobranej próby losowej. Możemy np. estymować punktowo lub przedziałowo nieznane parametry rozkładów,
Wnioskowanie bayesowskie W podejściu klasycznym wnioskowanie statystyczne oparte jest wyłącznie na podstawie pobranej próby losowej. Możemy np. estymować punktowo lub przedziałowo nieznane parametry rozkładów,
Zastosowanie analizy mitochondrialnego DNA. w badaniach kryminalistycznych. Katarzyna Gawêda-Walerych Ireneusz So³tyszewski
 Katarzyna Gawêda-Walerych Ireneusz So³tyszewski Zastosowanie analizy mitochondrialnego DNA w badaniach kryminalistycznych perspektywy Analiza mitochondrialnego DNA (mtdna) jest rutynow¹ metod¹ stosowan¹
Katarzyna Gawêda-Walerych Ireneusz So³tyszewski Zastosowanie analizy mitochondrialnego DNA w badaniach kryminalistycznych perspektywy Analiza mitochondrialnego DNA (mtdna) jest rutynow¹ metod¹ stosowan¹
OCENA ROZPRAWY NA STOPIEŃ NAUKOWY DOKTORA NAUK MEDYCZNYCH LEKARZA KRZYSZTOFA WIKTORA. pt. Analiza sekwencji mitochondrialnego DNA u
 Prof. dr hab. n. med. Paweł Kamiński Klinika Ginekologii i Ginekologii Onkologicznej Wojskowego Instytutu Medycznego OCENA ROZPRAWY NA STOPIEŃ NAUKOWY DOKTORA NAUK MEDYCZNYCH LEKARZA KRZYSZTOFA WIKTORA
Prof. dr hab. n. med. Paweł Kamiński Klinika Ginekologii i Ginekologii Onkologicznej Wojskowego Instytutu Medycznego OCENA ROZPRAWY NA STOPIEŃ NAUKOWY DOKTORA NAUK MEDYCZNYCH LEKARZA KRZYSZTOFA WIKTORA
BioTe21, Pracownia Kryminalistyki i Badań Ojcostwa.
 Bio Kraków, dnia... EKSPERTYZA Z BADAŃ GENETYCZNYCH POKREWIEŃSTWA Nr ekspertyzy:... Badania wykonano w: Bio, Ojcostwa. Na zlecenie:... Typ wybranego testu: TIG3-16 Zlecenie z dnia:... Data otrzymania mat.
Bio Kraków, dnia... EKSPERTYZA Z BADAŃ GENETYCZNYCH POKREWIEŃSTWA Nr ekspertyzy:... Badania wykonano w: Bio, Ojcostwa. Na zlecenie:... Typ wybranego testu: TIG3-16 Zlecenie z dnia:... Data otrzymania mat.
KARTA MODUŁU/PRZEDMIOTU
 . 2. 3. Nazwa modułu/przedmiotu Kod modułu/przedmiotu Przynależność do grupy przedmiotów 4. Status modułu/przedmiotu 5. Poziom kształcenia 6. 7. Forma studiów Profil kształcenia stacjonarne praktyczny
. 2. 3. Nazwa modułu/przedmiotu Kod modułu/przedmiotu Przynależność do grupy przedmiotów 4. Status modułu/przedmiotu 5. Poziom kształcenia 6. 7. Forma studiów Profil kształcenia stacjonarne praktyczny
PLAN STUDIÓW PODYPLOMOWYCH: DIAGNOSTYKA MOLEKULARNA W ROKU 2019/2020. Nazwa modułu ECTS Semestr I Semestr II. Liczba godzin z.
 Załącznik nr 5 do uchwały nr 79/2018-2019 Senatu Uniwersytetu Przyrodniczego w Lublinie z dnia 24 maja 2019 r. Symbol modułu PLAN STUDIÓW PODYPLOMOWYCH: DIAGNOSTYKA MOLEKULARNA W ROKU 2019/2020 Nazwa modułu
Załącznik nr 5 do uchwały nr 79/2018-2019 Senatu Uniwersytetu Przyrodniczego w Lublinie z dnia 24 maja 2019 r. Symbol modułu PLAN STUDIÓW PODYPLOMOWYCH: DIAGNOSTYKA MOLEKULARNA W ROKU 2019/2020 Nazwa modułu
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
Urszula Poziomek, doradca metodyczny w zakresie biologii Materiał dydaktyczny przygotowany na konferencję z cyklu Na miarę Nobla, 14 stycznia 2010 r.
 Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Zależności pomiędzy przynależnością haplogrupową a haplotypem Y-STR jako potencjalny element kontroli poprawności analiz w medycynie sądowej
 ARCH. MED. SĄD. KRYM., 2006, LVI, 155-164 PRACE ORYGINALNE Marcin Woźniak, Tomasz Grzybowski, Jarosław Starzyński, Marta Papuga, Katarzyna Stopińska, Sylwia Łuczak Zależności pomiędzy przynależnością haplogrupową
ARCH. MED. SĄD. KRYM., 2006, LVI, 155-164 PRACE ORYGINALNE Marcin Woźniak, Tomasz Grzybowski, Jarosław Starzyński, Marta Papuga, Katarzyna Stopińska, Sylwia Łuczak Zależności pomiędzy przynależnością haplogrupową
Badania genetyczne nad populacją jelenia w północno-wschodniej Polsce
 Badania genetyczne nad populacją jelenia w północno-wschodniej Polsce Magdalena Niedziałkowska, Bogumiła Jędrzejewska, Jan Marek Wójcik Instytut Biologii Ssaków PAN w Białowieży Cele badań 1) Poznanie
Badania genetyczne nad populacją jelenia w północno-wschodniej Polsce Magdalena Niedziałkowska, Bogumiła Jędrzejewska, Jan Marek Wójcik Instytut Biologii Ssaków PAN w Białowieży Cele badań 1) Poznanie
Dr hab.n.med. Renata Jacewicz
 GENOM CZŁOWIEKA >99 % 0,05%(100MtDNA) 65% Dr hab.n.med. Renata Jacewicz Kierownik Pracowni Genetyki Medycznej i Sądowej 3% 32% 2013 Pracownia Genetyki Medycznej i Sądowej ZMS Dojrzałe erytrocyty, trzony
GENOM CZŁOWIEKA >99 % 0,05%(100MtDNA) 65% Dr hab.n.med. Renata Jacewicz Kierownik Pracowni Genetyki Medycznej i Sądowej 3% 32% 2013 Pracownia Genetyki Medycznej i Sądowej ZMS Dojrzałe erytrocyty, trzony
ZALEŻNOŚĆ MIĘDZY WYSOKOŚCIĄ I MASĄ CIAŁA RODZICÓW I DZIECI W DWÓCH RÓŻNYCH ŚRODOWISKACH
 S ł u p s k i e P r a c e B i o l o g i c z n e 1 2005 Władimir Bożiłow 1, Małgorzata Roślak 2, Henryk Stolarczyk 2 1 Akademia Medyczna, Bydgoszcz 2 Uniwersytet Łódzki, Łódź ZALEŻNOŚĆ MIĘDZY WYSOKOŚCIĄ
S ł u p s k i e P r a c e B i o l o g i c z n e 1 2005 Władimir Bożiłow 1, Małgorzata Roślak 2, Henryk Stolarczyk 2 1 Akademia Medyczna, Bydgoszcz 2 Uniwersytet Łódzki, Łódź ZALEŻNOŚĆ MIĘDZY WYSOKOŚCIĄ
Filogenetyka molekularna I. Krzysztof Spalik
 Filogenetyka molekularna I Krzysztof Spalik Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498
Filogenetyka molekularna I Krzysztof Spalik Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498
Genomika Porównawcza. Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski
 Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
Zabezpieczanie próbek biologicznych i rejestracja profili w Bazie Danych DNA
 Zabezpieczanie próbek biologicznych i rejestracja profili w Bazie Danych DNA w przypadku: PROWADZENIA CZYNNOŚCI IDENTYFIKACYJNYCH NN ZWŁOK I. PRZEPISY OKREŚLAJĄCE OBOWIĄZKI POLICJANTÓW ORAZ ZASADY POSTĘPOWANIA
Zabezpieczanie próbek biologicznych i rejestracja profili w Bazie Danych DNA w przypadku: PROWADZENIA CZYNNOŚCI IDENTYFIKACYJNYCH NN ZWŁOK I. PRZEPISY OKREŚLAJĄCE OBOWIĄZKI POLICJANTÓW ORAZ ZASADY POSTĘPOWANIA
AP I A 060/79/12. Komunikat prasowy
 PROKURATURA APELACYJNA W GDAŃSKU WYDZIAŁ I ORGANIZACJI PRACY PROKURATUR ul. Wały Jagiellońskie 38 80 853 Gdańsk Gdańsk, dnia 31 grudnia 2012r. AP I A 060/79/12 Komunikat prasowy Wydział V do Spraw Przestępczości
PROKURATURA APELACYJNA W GDAŃSKU WYDZIAŁ I ORGANIZACJI PRACY PROKURATUR ul. Wały Jagiellońskie 38 80 853 Gdańsk Gdańsk, dnia 31 grudnia 2012r. AP I A 060/79/12 Komunikat prasowy Wydział V do Spraw Przestępczości
BADANIA ZRÓŻNICOWANIA RYZYKA WYPADKÓW PRZY PRACY NA PRZYKŁADZIE ANALIZY STATYSTYKI WYPADKÓW DLA BRANŻY GÓRNICTWA I POLSKI
 14 BADANIA ZRÓŻNICOWANIA RYZYKA WYPADKÓW PRZY PRACY NA PRZYKŁADZIE ANALIZY STATYSTYKI WYPADKÓW DLA BRANŻY GÓRNICTWA I POLSKI 14.1 WSTĘP Ogólne wymagania prawne dotyczące przy pracy określają m.in. przepisy
14 BADANIA ZRÓŻNICOWANIA RYZYKA WYPADKÓW PRZY PRACY NA PRZYKŁADZIE ANALIZY STATYSTYKI WYPADKÓW DLA BRANŻY GÓRNICTWA I POLSKI 14.1 WSTĘP Ogólne wymagania prawne dotyczące przy pracy określają m.in. przepisy
SYLABUS DOTYCZY CYKLU KSZTAŁCENIA
 SYLABUS DOTYCZY CYKLU KSZTAŁCENIA 2015-2021 1.1. PODSTAWOWE INFORMACJE O PRZEDMIOCIE/MODULE Nazwa przedmiotu/ modułu Techniki biologii molekularnej Kod przedmiotu/ modułu* Wydział (nazwa jednostki prowadzącej
SYLABUS DOTYCZY CYKLU KSZTAŁCENIA 2015-2021 1.1. PODSTAWOWE INFORMACJE O PRZEDMIOCIE/MODULE Nazwa przedmiotu/ modułu Techniki biologii molekularnej Kod przedmiotu/ modułu* Wydział (nazwa jednostki prowadzącej
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
Biologia medyczna, materiały dla studentów
 Jaka tam ewolucja. Zanim trafię na jednego myślącego, muszę stoczyć bitwę zdziewięcioma orangutanami Carlos Ruis Zafon Wierzbownica drobnokwiatowa Fitosterole, garbniki, flawonoidy Właściwości przeciwzapalne,
Jaka tam ewolucja. Zanim trafię na jednego myślącego, muszę stoczyć bitwę zdziewięcioma orangutanami Carlos Ruis Zafon Wierzbownica drobnokwiatowa Fitosterole, garbniki, flawonoidy Właściwości przeciwzapalne,
Ćwiczenie 12. Diagnostyka molekularna. Poszukiwanie SNPs Odczytywanie danych z sekwencjonowania. Prof. dr hab. Roman Zieliński
 Ćwiczenie 12 Diagnostyka molekularna. Poszukiwanie SNPs Odczytywanie danych z sekwencjonowania Prof. dr hab. Roman Zieliński 1. Diagnostyka molekularna 1.1. Pytania i zagadnienia 1.1.1. Jak definiujemy
Ćwiczenie 12 Diagnostyka molekularna. Poszukiwanie SNPs Odczytywanie danych z sekwencjonowania Prof. dr hab. Roman Zieliński 1. Diagnostyka molekularna 1.1. Pytania i zagadnienia 1.1.1. Jak definiujemy
Dr hab.n.med. Renata Jacewicz
 GENOM CZŁOWIEKA >99,999% 0,0005% 65% Dr hab.n.med. Renata Jacewicz Kierownik Pracowni Genetyki Medycznej i Sądowej 3% 32% 2013 Pracownia Genetyki Medycznej i Sądowej ZMS Dojrzałe erytrocyty, trzony włosów
GENOM CZŁOWIEKA >99,999% 0,0005% 65% Dr hab.n.med. Renata Jacewicz Kierownik Pracowni Genetyki Medycznej i Sądowej 3% 32% 2013 Pracownia Genetyki Medycznej i Sądowej ZMS Dojrzałe erytrocyty, trzony włosów
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
Modelowanie motywów łańcuchami Markowa wyższego rzędu
 Modelowanie motywów łańcuchami Markowa wyższego rzędu Uniwersytet Warszawski Wydział Matematyki, Informatyki i Mechaniki 23 października 2008 roku Plan prezentacji 1 Źródła 2 Motywy i ich znaczenie Łańcuchy
Modelowanie motywów łańcuchami Markowa wyższego rzędu Uniwersytet Warszawski Wydział Matematyki, Informatyki i Mechaniki 23 października 2008 roku Plan prezentacji 1 Źródła 2 Motywy i ich znaczenie Łańcuchy
Ewolucja człowieka. Ostatnie 5 milionów lat
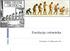 Ewolucja człowieka Ostatnie 5 milionów lat 1 Złożone zagadki } } Odnaleziono wiele skamieniałości naczelnych, różne gatunki w tym samym czasie Trudno ustalić relacje między nimi } Przodkowie, czy boczne
Ewolucja człowieka Ostatnie 5 milionów lat 1 Złożone zagadki } } Odnaleziono wiele skamieniałości naczelnych, różne gatunki w tym samym czasie Trudno ustalić relacje między nimi } Przodkowie, czy boczne
STATYSTYKA MATEMATYCZNA WYKŁAD 1
 STATYSTYKA MATEMATYCZNA WYKŁAD 1 Wykład wstępny Teoria prawdopodobieństwa Magda Mielczarek wykłady, ćwiczenia Copyright 2017, J. Szyda & M. Mielczarek STATYSTYKA MATEMATYCZNA? ASHG 2011 Writing Workshop;
STATYSTYKA MATEMATYCZNA WYKŁAD 1 Wykład wstępny Teoria prawdopodobieństwa Magda Mielczarek wykłady, ćwiczenia Copyright 2017, J. Szyda & M. Mielczarek STATYSTYKA MATEMATYCZNA? ASHG 2011 Writing Workshop;
Ocena rozprawy doktorskiej. Mgr Pauliny Smyk pt.: Wpływ wybranych ksenobiotyków na zmiany parametrów
 Bydgoszcz, 30. 05. 2019 r. prof. dr hab. Marek Bednarczyk Katedra Biotechnologii i Genetyki Zwierząt Uniwersytet Technologiczno-Przyrodniczy im. J.J. Śniadeckich w Bydgoszczy Ocena rozprawy doktorskiej
Bydgoszcz, 30. 05. 2019 r. prof. dr hab. Marek Bednarczyk Katedra Biotechnologii i Genetyki Zwierząt Uniwersytet Technologiczno-Przyrodniczy im. J.J. Śniadeckich w Bydgoszczy Ocena rozprawy doktorskiej
Spis treści. Przedmowa... XI. Wprowadzenie i biologiczne bazy danych. 1 Wprowadzenie... 3. 2 Wprowadzenie do biologicznych baz danych...
 Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Monitoring genetyczny populacji wilka (Canis lupus) jako nowy element monitoringu stanu populacji dużych drapieżników
 Monitoring genetyczny populacji wilka (Canis lupus) jako nowy element monitoringu stanu populacji dużych drapieżników Wojciech Śmietana Co to jest monitoring genetyczny? Monitoring genetyczny to regularnie
Monitoring genetyczny populacji wilka (Canis lupus) jako nowy element monitoringu stanu populacji dużych drapieżników Wojciech Śmietana Co to jest monitoring genetyczny? Monitoring genetyczny to regularnie
OBLICZENIE PRZEPŁYWÓW MAKSYMALNYCH ROCZNYCH O OKREŚLONYM PRAWDOPODOBIEŃSTWIE PRZEWYŻSZENIA. z wykorzystaniem programu obliczeniowego Q maxp
 tel.: +48 662 635 712 Liczba stron: 15 Data: 20.07.2010r OBLICZENIE PRZEPŁYWÓW MAKSYMALNYCH ROCZNYCH O OKREŚLONYM PRAWDOPODOBIEŃSTWIE PRZEWYŻSZENIA z wykorzystaniem programu obliczeniowego Q maxp DŁUGIE
tel.: +48 662 635 712 Liczba stron: 15 Data: 20.07.2010r OBLICZENIE PRZEPŁYWÓW MAKSYMALNYCH ROCZNYCH O OKREŚLONYM PRAWDOPODOBIEŃSTWIE PRZEWYŻSZENIA z wykorzystaniem programu obliczeniowego Q maxp DŁUGIE
Walidacja metod wykrywania, identyfikacji i ilościowego oznaczania GMO. Magdalena Żurawska-Zajfert Laboratorium Kontroli GMO IHAR-PIB
 Walidacja metod wykrywania, identyfikacji i ilościowego oznaczania GMO Magdalena Żurawska-Zajfert Laboratorium Kontroli GMO IHAR-PIB Walidacja Walidacja jest potwierdzeniem przez zbadanie i przedstawienie
Walidacja metod wykrywania, identyfikacji i ilościowego oznaczania GMO Magdalena Żurawska-Zajfert Laboratorium Kontroli GMO IHAR-PIB Walidacja Walidacja jest potwierdzeniem przez zbadanie i przedstawienie
Recenzja rozprawy doktorskiej. mgr Marcina Jana Kamińskiego. pt. Grupa rodzajowa Ectateus (Coleoptera: Tenebrionidae) filogeneza i klasyfikacja.
 Dr hab. Grzegorz Paśnik Instytut Systematyki i Ewolucji Zwierząt Polska Akademia Nauk ul. Sławkowska 17, 31-016 Kraków Recenzja rozprawy doktorskiej mgr Marcina Jana Kamińskiego pt. Grupa rodzajowa Ectateus
Dr hab. Grzegorz Paśnik Instytut Systematyki i Ewolucji Zwierząt Polska Akademia Nauk ul. Sławkowska 17, 31-016 Kraków Recenzja rozprawy doktorskiej mgr Marcina Jana Kamińskiego pt. Grupa rodzajowa Ectateus
dr hab. n. med. Czesław Żaba Kierownik Katedry i Zakładu Medycyny Sądowej Uniwersytetu Medycznego im. Karola Marcinkowskiego w Poznaniu
 dr hab. n. med. Czesław Żaba Kierownik Katedry i Zakładu Medycyny Sądowej Uniwersytetu Medycznego im. Karola Marcinkowskiego w Poznaniu Poznań, dnia 01.09.2016 r. Ocena rozprawy doktorskiej mgr Matyldy
dr hab. n. med. Czesław Żaba Kierownik Katedry i Zakładu Medycyny Sądowej Uniwersytetu Medycznego im. Karola Marcinkowskiego w Poznaniu Poznań, dnia 01.09.2016 r. Ocena rozprawy doktorskiej mgr Matyldy
Wykład 8: Testy istotności
 Wykład 8: Testy istotności Hipotezy Statystyki testowe P-wartości Istotność statystyczna Test dla średniej w populacji Dwustronny test a przedział ufności Używanie i nadużywanie testów Testy istotności
Wykład 8: Testy istotności Hipotezy Statystyki testowe P-wartości Istotność statystyczna Test dla średniej w populacji Dwustronny test a przedział ufności Używanie i nadużywanie testów Testy istotności
Statystyka. Rozkład prawdopodobieństwa Testowanie hipotez. Wykład III ( )
 Statystyka Rozkład prawdopodobieństwa Testowanie hipotez Wykład III (04.01.2016) Rozkład t-studenta Rozkład T jest rozkładem pomocniczym we wnioskowaniu statystycznym; stosuje się go wyznaczenia przedziału
Statystyka Rozkład prawdopodobieństwa Testowanie hipotez Wykład III (04.01.2016) Rozkład t-studenta Rozkład T jest rozkładem pomocniczym we wnioskowaniu statystycznym; stosuje się go wyznaczenia przedziału
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
RÓWNOWAŻNOŚĆ METOD BADAWCZYCH
 RÓWNOWAŻNOŚĆ METOD BADAWCZYCH Piotr Konieczka Katedra Chemii Analitycznej Wydział Chemiczny Politechnika Gdańska Równoważność metod??? 2 Zgodność wyników analitycznych otrzymanych z wykorzystaniem porównywanych
RÓWNOWAŻNOŚĆ METOD BADAWCZYCH Piotr Konieczka Katedra Chemii Analitycznej Wydział Chemiczny Politechnika Gdańska Równoważność metod??? 2 Zgodność wyników analitycznych otrzymanych z wykorzystaniem porównywanych
Analiza korespondencji
 Analiza korespondencji Kiedy stosujemy? 2 W wielu badaniach mamy do czynienia ze zmiennymi jakościowymi (nominalne i porządkowe) typu np.: płeć, wykształcenie, status palenia. Punktem wyjścia do analizy
Analiza korespondencji Kiedy stosujemy? 2 W wielu badaniach mamy do czynienia ze zmiennymi jakościowymi (nominalne i porządkowe) typu np.: płeć, wykształcenie, status palenia. Punktem wyjścia do analizy
ACTA UNIVERSITATIS LODZIENSIS. WIELOLETNIA ZMIENNOŚĆ WYSTĘPOWANIA BURZ W SZCZECINIE, ŁODZI, KRAKOWIE I NA KASPROWYM WIERCHU W LATAm
 ACTA UNIVERSITATIS LODZIENSIS FOLIA GEOGRAPHICA PHYSICA 3, 1998 Zuzanna Bielec WIELOLETNIA ZMIENNOŚĆ WYSTĘPOWANIA BURZ W SZCZECINIE, ŁODZI, KRAKOWIE I NA KASPROWYM WIERCHU W LATAm 1954-1993 LONG-TERM VARIABILITY
ACTA UNIVERSITATIS LODZIENSIS FOLIA GEOGRAPHICA PHYSICA 3, 1998 Zuzanna Bielec WIELOLETNIA ZMIENNOŚĆ WYSTĘPOWANIA BURZ W SZCZECINIE, ŁODZI, KRAKOWIE I NA KASPROWYM WIERCHU W LATAm 1954-1993 LONG-TERM VARIABILITY
METODY CHEMOMETRYCZNE W IDENTYFIKACJI ŹRÓDEŁ POCHODZENIA
 METODY CHEMOMETRYCZNE W IDENTYFIKACJI ŹRÓDEŁ POCHODZENIA AMFETAMINY Waldemar S. Krawczyk Centralne Laboratorium Kryminalistyczne Komendy Głównej Policji, Warszawa (praca obroniona na Wydziale Chemii Uniwersytetu
METODY CHEMOMETRYCZNE W IDENTYFIKACJI ŹRÓDEŁ POCHODZENIA AMFETAMINY Waldemar S. Krawczyk Centralne Laboratorium Kryminalistyczne Komendy Głównej Policji, Warszawa (praca obroniona na Wydziale Chemii Uniwersytetu
Wysoko wiarygodne metody identyfikacji osób
 Biuletyn WAT Vol. LXII, Nr 4, 2013 Wysoko wiarygodne metody identyfikacji osób WIKTOR OLCHOWIK Wojskowa Akademia Techniczna, Wydział Elektroniki, 00-908 Warszawa, ul. gen. S. Kaliskiego 2, wolchowik@wat.edu.pl
Biuletyn WAT Vol. LXII, Nr 4, 2013 Wysoko wiarygodne metody identyfikacji osób WIKTOR OLCHOWIK Wojskowa Akademia Techniczna, Wydział Elektroniki, 00-908 Warszawa, ul. gen. S. Kaliskiego 2, wolchowik@wat.edu.pl
Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna
 Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna Spis treści Przedmowa................................. Podziękowania............................... XIII XIV 1 Metody genetyki molekularnej w badaniach
Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna Spis treści Przedmowa................................. Podziękowania............................... XIII XIV 1 Metody genetyki molekularnej w badaniach
AUTOREFERAT dr n. med. Katarzyna Skonieczna
 AUTOREFERAT dr n. med. Katarzyna Skonieczna Zakład Genetyki Molekularnej i Sądowej Katedra Medycyny Sądowej Wydział Lekarski Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Uniwersytet Mikołaja Kopernika
AUTOREFERAT dr n. med. Katarzyna Skonieczna Zakład Genetyki Molekularnej i Sądowej Katedra Medycyny Sądowej Wydział Lekarski Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Uniwersytet Mikołaja Kopernika
SPRAWOZDANIE Z BADAŃ DNA W KIERUNKU USTALENIA POKREWIEŃSTWA BIOLOGICZNEGO DO CELÓW PRYWATNYCH
 SPRAWOZDANIE Z BADAŃ DNA W KIERUNKU USTALENIA POKREWIEŃSTWA BIOLOGICZNEGO DO CELÓW PRYWATNYCH RAPORT TESTU DNA W KIERUNKU USTALENIA POKREWIEŃSTWA BIOLOGICZNEGO Data wydania wyniku: 15-01-2016 WYNIK TESTU
SPRAWOZDANIE Z BADAŃ DNA W KIERUNKU USTALENIA POKREWIEŃSTWA BIOLOGICZNEGO DO CELÓW PRYWATNYCH RAPORT TESTU DNA W KIERUNKU USTALENIA POKREWIEŃSTWA BIOLOGICZNEGO Data wydania wyniku: 15-01-2016 WYNIK TESTU
Teoria ewolucji. Podstawy wspólne pochodzenie.
 Teoria ewolucji. Podstawy wspólne pochodzenie. Ewolucja biologiczna } Znaczenie ogólne: } proces zmian informacji genetycznej (częstości i rodzaju alleli), } które to zmiany są przekazywane z pokolenia
Teoria ewolucji. Podstawy wspólne pochodzenie. Ewolucja biologiczna } Znaczenie ogólne: } proces zmian informacji genetycznej (częstości i rodzaju alleli), } które to zmiany są przekazywane z pokolenia
Statystyka i opracowanie danych Podstawy wnioskowania statystycznego. Prawo wielkich liczb. Centralne twierdzenie graniczne. Estymacja i estymatory
 Statystyka i opracowanie danych Podstawy wnioskowania statystycznego. Prawo wielkich liczb. Centralne twierdzenie graniczne. Estymacja i estymatory Dr Anna ADRIAN Paw B5, pok 407 adrian@tempus.metal.agh.edu.pl
Statystyka i opracowanie danych Podstawy wnioskowania statystycznego. Prawo wielkich liczb. Centralne twierdzenie graniczne. Estymacja i estymatory Dr Anna ADRIAN Paw B5, pok 407 adrian@tempus.metal.agh.edu.pl
Ćwiczenie 2. Identyfikacja płci z wykorzystaniem genu amelogeniny (AMGXY)
 Ćwiczenie 2. Identyfikacja płci z wykorzystaniem genu amelogeniny (AMGXY) Cel ćwiczenia Amplifikacja fragmentu genu amelogeniny, znajdującego się na chromosomach X i Y, jako celu molekularnego przydatnego
Ćwiczenie 2. Identyfikacja płci z wykorzystaniem genu amelogeniny (AMGXY) Cel ćwiczenia Amplifikacja fragmentu genu amelogeniny, znajdującego się na chromosomach X i Y, jako celu molekularnego przydatnego
Genomika praktyczna. Genomika praktyczna. Zakład Biochemii i Farmakogenomiki. prof. dr hab. Grażyna Nowicka. Rok IV. Semestr 8.
 Genomika praktyczna 1. Metryczka Nazwa Wydziału: Program kształcenia (kierunek studiów, poziom i profil kształcenia, forma studiów, np. Zdrowie publiczne I stopnia profil praktyczny, studia stacjonarne):
Genomika praktyczna 1. Metryczka Nazwa Wydziału: Program kształcenia (kierunek studiów, poziom i profil kształcenia, forma studiów, np. Zdrowie publiczne I stopnia profil praktyczny, studia stacjonarne):
Pełny czas mutacji STR w podręcznej i praktycznej tabeli PCM.
 Pełny czas mutacji STR w podręcznej i praktycznej tabeli PCM. Full Times of Mutations STR in the practical table FTM Obliczanie pełnego czasu mutacji ( PCM ) Calculation of full-time of mutations ( FTM
Pełny czas mutacji STR w podręcznej i praktycznej tabeli PCM. Full Times of Mutations STR in the practical table FTM Obliczanie pełnego czasu mutacji ( PCM ) Calculation of full-time of mutations ( FTM
Horyzonty daktyloskopii
 Horyzonty daktyloskopii Spis treści Rozdział 1 Daktyloskopia wśród nauk sądowych...9 1.1. Polska daktyloskopia wczoraj i dziś wybrane zagadnienia...11 Jarosław Moszczyński 1.1.1. Zakres rejestracji daktyloskopijnej
Horyzonty daktyloskopii Spis treści Rozdział 1 Daktyloskopia wśród nauk sądowych...9 1.1. Polska daktyloskopia wczoraj i dziś wybrane zagadnienia...11 Jarosław Moszczyński 1.1.1. Zakres rejestracji daktyloskopijnej
PODSTAWY BIOINFORMATYKI
 PODSTAWY BIOINFORMATYKI Prowadzący: JOANNA SZYDA ADRIAN DROśDś WSTĘP 1. Katedra Genetyki badania bioinformatyczne 2. Tematyka przedmiotu 3. Charakterystyka wykładów 4. Charakterystyka ćwiczeń 5. Informacje
PODSTAWY BIOINFORMATYKI Prowadzący: JOANNA SZYDA ADRIAN DROśDś WSTĘP 1. Katedra Genetyki badania bioinformatyczne 2. Tematyka przedmiotu 3. Charakterystyka wykładów 4. Charakterystyka ćwiczeń 5. Informacje
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZE W BIOLOGII 1. Wykład wstępny. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. ajczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZE W BIOLOGII 1. Wykład wstępny. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. ajczęściej wykorzystywane testy statystyczne
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
3. Podstawy genetyki S YLABUS MODUŁU (PRZEDMIOTU) I nformacje ogólne. Nazwa modułu. Kod F3/A. Podstawy genetyki. modułu
 S YLABUS MODUŁU (PRZEDMIOTU) 3. Podstawy genetyki I nformacje ogólne Kod F3/A modułu Rodzaj modułu Wydział PUM Kierunek studiów Specjalność Poziom studiów Forma studiów Rok studiów Nazwa modułu Podstawy
S YLABUS MODUŁU (PRZEDMIOTU) 3. Podstawy genetyki I nformacje ogólne Kod F3/A modułu Rodzaj modułu Wydział PUM Kierunek studiów Specjalność Poziom studiów Forma studiów Rok studiów Nazwa modułu Podstawy
Porównywanie i dopasowywanie sekwencji
 Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek narodziła się nowa dyscyplina nauki ewolucja molekularna Ewolucja molekularna
Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek narodziła się nowa dyscyplina nauki ewolucja molekularna Ewolucja molekularna
STATYSTYKA MATEMATYCZNA
 STATYSTYKA MATEMATYCZNA 1. Wykład wstępny. Teoria prawdopodobieństwa i elementy kombinatoryki 2. Zmienne losowe i ich rozkłady 3. opulacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5.
STATYSTYKA MATEMATYCZNA 1. Wykład wstępny. Teoria prawdopodobieństwa i elementy kombinatoryki 2. Zmienne losowe i ich rozkłady 3. opulacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5.
Zad. 4 Należy określić rodzaj testu (jedno czy dwustronny) oraz wartości krytyczne z lub t dla określonych hipotez i ich poziomów istotności:
 Zadania ze statystyki cz. 7. Zad.1 Z populacji wyłoniono próbę wielkości 64 jednostek. Średnia arytmetyczna wartość cechy wyniosła 110, zaś odchylenie standardowe 16. Należy wyznaczyć przedział ufności
Zadania ze statystyki cz. 7. Zad.1 Z populacji wyłoniono próbę wielkości 64 jednostek. Średnia arytmetyczna wartość cechy wyniosła 110, zaś odchylenie standardowe 16. Należy wyznaczyć przedział ufności
Dopasowanie sekwencji (sequence alignment)
 Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Akademia Morska w Szczecinie. Wydział Mechaniczny
 Akademia Morska w Szczecinie Wydział Mechaniczny ROZPRAWA DOKTORSKA mgr inż. Marcin Kołodziejski Analiza metody obsługiwania zarządzanego niezawodnością pędników azymutalnych platformy pływającej Promotor:
Akademia Morska w Szczecinie Wydział Mechaniczny ROZPRAWA DOKTORSKA mgr inż. Marcin Kołodziejski Analiza metody obsługiwania zarządzanego niezawodnością pędników azymutalnych platformy pływającej Promotor:
Analiza częstości mutacji w parach ojciec-syn w wybranych
 ARCH. MED. SĄD. KRYMINOL., 2012, LXII, 147-151 PRACE ORYGINALNE / ORIGINAL PAPERS Joanna Wysocka, Aneta Stasiewicz 1, Krzysztof Rębała, Ewa Kapińska, Lidia Cybulska, Zofia Szczerkowska Analiza częstości
ARCH. MED. SĄD. KRYMINOL., 2012, LXII, 147-151 PRACE ORYGINALNE / ORIGINAL PAPERS Joanna Wysocka, Aneta Stasiewicz 1, Krzysztof Rębała, Ewa Kapińska, Lidia Cybulska, Zofia Szczerkowska Analiza częstości
Badanie doboru naturalnego na poziomie molekularnym
 Badanie doboru naturalnego na poziomie molekularnym Podstawy ewolucji molekulanej Jak ewoluują sekwencje Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na
Badanie doboru naturalnego na poziomie molekularnym Podstawy ewolucji molekulanej Jak ewoluują sekwencje Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na
STATYSTYKA MATEMATYCZNA
 STATYSTYKA MATEMATYCZNA 1. Wykład wstępny. Teoria prawdopodobieństwa i elementy kombinatoryki 2. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5.
STATYSTYKA MATEMATYCZNA 1. Wykład wstępny. Teoria prawdopodobieństwa i elementy kombinatoryki 2. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5.
Kostrzyca dn r. Nadleśnictwo Sława Śląska. Wykorzystanie analizy DNA drewna dębowego w postępowaniu karnym
 Kostrzyca dn. 27.09.2012r. Nadleśnictwo Sława Śląska Wykorzystanie analizy DNA drewna dębowego w postępowaniu karnym PRZYNALEŻNOŚĆ ADMINISTRACYJNA NADLEŚNICTWA Regionalna Dyrekcja Lasów Państwowych w Zielonej
Kostrzyca dn. 27.09.2012r. Nadleśnictwo Sława Śląska Wykorzystanie analizy DNA drewna dębowego w postępowaniu karnym PRZYNALEŻNOŚĆ ADMINISTRACYJNA NADLEŚNICTWA Regionalna Dyrekcja Lasów Państwowych w Zielonej
KARTA PRZEDMIOTU. (pieczęć wydziału)
 (pieczęć wydziału) KARTA PRZEDMIOTU 1. Nazwa przedmiotu: Eksploracja danych w bioinformatyce 2. Kod przedmiotu: EksDaBio 3. Karta przedmiotu ważna od roku akademickiego: 2017/2018 4. Forma kształcenia:
(pieczęć wydziału) KARTA PRZEDMIOTU 1. Nazwa przedmiotu: Eksploracja danych w bioinformatyce 2. Kod przedmiotu: EksDaBio 3. Karta przedmiotu ważna od roku akademickiego: 2017/2018 4. Forma kształcenia:
Wpływ struktury krajobrazu na przestrzenną zmienność genetyczną populacji myszy leśnej Apodemus flavicollis w północno wschodniej Polsce
 Uniwersytet Warszawski Wydział Biologii Sylwia Czarnomska Wpływ struktury krajobrazu na przestrzenną zmienność genetyczną populacji myszy leśnej Apodemus flavicollis w północno wschodniej Polsce Autoreferat
Uniwersytet Warszawski Wydział Biologii Sylwia Czarnomska Wpływ struktury krajobrazu na przestrzenną zmienność genetyczną populacji myszy leśnej Apodemus flavicollis w północno wschodniej Polsce Autoreferat
WSTĘP DO REGRESJI LOGISTYCZNEJ. Dr Wioleta Drobik-Czwarno
 WSTĘP DO REGRESJI LOGISTYCZNEJ Dr Wioleta Drobik-Czwarno REGRESJA LOGISTYCZNA Zmienna zależna jest zmienną dychotomiczną (dwustanową) przyjmuje dwie wartości, najczęściej 0 i 1 Zmienną zależną może być:
WSTĘP DO REGRESJI LOGISTYCZNEJ Dr Wioleta Drobik-Czwarno REGRESJA LOGISTYCZNA Zmienna zależna jest zmienną dychotomiczną (dwustanową) przyjmuje dwie wartości, najczęściej 0 i 1 Zmienną zależną może być:
Białowieża, Prof. dr hab. Jan M. Wójcik Instytut Biologii Ssaków PAN w Białowieży
 Białowieża, 05.04.2017 Prof. dr hab. Jan M. Wójcik Instytut Biologii Ssaków PAN w Białowieży e-mail: jwojcik@ibs.bialowieza.pl Recenzja rozprawy doktorskiej mgr. Macieja Matosiuka pt. Hybridization and
Białowieża, 05.04.2017 Prof. dr hab. Jan M. Wójcik Instytut Biologii Ssaków PAN w Białowieży e-mail: jwojcik@ibs.bialowieza.pl Recenzja rozprawy doktorskiej mgr. Macieja Matosiuka pt. Hybridization and
MODELOWANIE STANÓW CZYNNOŚCIOWYCH W JĘZYKU SIECI BAYESOWSKICH
 Inżynieria Rolnicza 7(105)/2008 MODELOWANIE STANÓW CZYNNOŚCIOWYCH W JĘZYKU SIECI BAYESOWSKICH Katedra Podstaw Techniki, Uniwersytet Przyrodniczy w Lublinie Streszczenie. Zastosowanie sieci bayesowskiej
Inżynieria Rolnicza 7(105)/2008 MODELOWANIE STANÓW CZYNNOŚCIOWYCH W JĘZYKU SIECI BAYESOWSKICH Katedra Podstaw Techniki, Uniwersytet Przyrodniczy w Lublinie Streszczenie. Zastosowanie sieci bayesowskiej
Wykorzystanie testu t dla pojedynczej próby we wnioskowaniu statystycznym
 Wiesława MALSKA Politechnika Rzeszowska, Polska Anna KOZIOROWSKA Uniwersytet Rzeszowski, Polska Wykorzystanie testu t dla pojedynczej próby we wnioskowaniu statystycznym Wstęp Wnioskowanie statystyczne
Wiesława MALSKA Politechnika Rzeszowska, Polska Anna KOZIOROWSKA Uniwersytet Rzeszowski, Polska Wykorzystanie testu t dla pojedynczej próby we wnioskowaniu statystycznym Wstęp Wnioskowanie statystyczne
Analiza mutacji genów EGFR, PIKCA i PTEN w nerwiaku zarodkowym
 Analiza mutacji genów EGFR, PIKCA i PTEN w nerwiaku zarodkowym mgr Magdalena Brzeskwiniewicz Promotor: Prof. dr hab. n. med. Janusz Limon Katedra i Zakład Biologii i Genetyki Gdański Uniwersytet Medyczny
Analiza mutacji genów EGFR, PIKCA i PTEN w nerwiaku zarodkowym mgr Magdalena Brzeskwiniewicz Promotor: Prof. dr hab. n. med. Janusz Limon Katedra i Zakład Biologii i Genetyki Gdański Uniwersytet Medyczny
Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2
 ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
Opis zakładanych efektów kształcenia na studiach podyplomowych WIEDZA
 Opis zakładanych efektów kształcenia na studiach podyplomowych Nazwa studiów: BIOSTATYSTYKA PRAKTYCZNE ASPEKTY STATYSTYKI W BADANIACH MEDYCZNYCH Typ studiów: doskonalące Symbol Efekty kształcenia dla studiów
Opis zakładanych efektów kształcenia na studiach podyplomowych Nazwa studiów: BIOSTATYSTYKA PRAKTYCZNE ASPEKTY STATYSTYKI W BADANIACH MEDYCZNYCH Typ studiów: doskonalące Symbol Efekty kształcenia dla studiów
Składniki jądrowego genomu człowieka
 Składniki jądrowego genomu człowieka Genom człowieka 3 000 Mpz (3x10 9, 100 cm) Geny i sekwencje związane z genami (900 Mpz, 30% g. jądrowego) DNA pozagenowy (2100 Mpz, 70%) DNA kodujący (90 Mpz ~ ok.
Składniki jądrowego genomu człowieka Genom człowieka 3 000 Mpz (3x10 9, 100 cm) Geny i sekwencje związane z genami (900 Mpz, 30% g. jądrowego) DNA pozagenowy (2100 Mpz, 70%) DNA kodujący (90 Mpz ~ ok.
Filogenetyka molekularna I
 2 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498 Filogenetyka molekularna I John C. Avise
2 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498 Filogenetyka molekularna I John C. Avise
Filogenetyka molekularna I. Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji
 Filogenetyka molekularna I Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji 3 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w
Filogenetyka molekularna I Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji 3 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w
STATYSTYKA EKONOMICZNA
 STATYSTYKA EKONOMICZNA Analiza statystyczna w ocenie działalności przedsiębiorstwa Opracowano na podstawie : E. Nowak, Metody statystyczne w analizie działalności przedsiębiorstwa, PWN, Warszawa 2001 Dr
STATYSTYKA EKONOMICZNA Analiza statystyczna w ocenie działalności przedsiębiorstwa Opracowano na podstawie : E. Nowak, Metody statystyczne w analizie działalności przedsiębiorstwa, PWN, Warszawa 2001 Dr
Wykład 9: HUMAN GENOME PROJECT HUMAN GENOME PROJECT
 Wykład 9: Polimorfizm pojedynczego nukleotydu (SNP) odrębność genetyczna, która czyni każdego z nas jednostką unikatową Prof. dr hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej HUMAN GENOME
Wykład 9: Polimorfizm pojedynczego nukleotydu (SNP) odrębność genetyczna, która czyni każdego z nas jednostką unikatową Prof. dr hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej HUMAN GENOME
ACTA UNIVERSITATIS LODZIENSIS KSZTAŁTOWANIE SIĘ WIELKOŚCI OPADÓW NA OBSZARZE WOJEWÓDZTWA MIEJSKIEGO KRAKOWSKIEGO
 ACTA UNIVERSITATIS LODZIENSIS FOLIA GEOGRAPHICA PHYSICA 3, 1998 Elżbieta Cebulak KSZTAŁTOWANIE SIĘ WIELKOŚCI OPADÓW NA OBSZARZE WOJEWÓDZTWA MIEJSKIEGO KRAKOWSKIEGO THE PRECIPITATION ON THE AREA OF CRACOW
ACTA UNIVERSITATIS LODZIENSIS FOLIA GEOGRAPHICA PHYSICA 3, 1998 Elżbieta Cebulak KSZTAŁTOWANIE SIĘ WIELKOŚCI OPADÓW NA OBSZARZE WOJEWÓDZTWA MIEJSKIEGO KRAKOWSKIEGO THE PRECIPITATION ON THE AREA OF CRACOW
Scenariusz lekcji z biologii w szkole ponadgimnazjalnej
 Scenariusz lekcji z biologii w szkole ponadgimnazjalnej Temat lekcji: Planowanie doświadczeń biologicznych jak prawidłowo zaplanować próbę kontrolną? Cele kształcenia IV etap edukacyjny: 1. Wymagania ogólne:
Scenariusz lekcji z biologii w szkole ponadgimnazjalnej Temat lekcji: Planowanie doświadczeń biologicznych jak prawidłowo zaplanować próbę kontrolną? Cele kształcenia IV etap edukacyjny: 1. Wymagania ogólne:
ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH
 1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
