Dopasowanie sekwencji Sequence alignment. Bioinformatyka, wykłady 3 i 4 (16, 23.X.2012)
|
|
|
- Magdalena Witkowska
- 6 lat temu
- Przeglądów:
Transkrypt
1 Dopasowanie sekwencji Sequence alignment Bioinformatyka, wykłady 3 i 4 (16, 23.X.2012) krzysztof_pawlowski@sggw.pl
2 terminologia alignment dopasowanie sekwencji 119 uliniowienie sekwencji 82 uliniowianie sekwencji 125 uliniawianie 6 uliniowanie 3 elajment 2
3 Uliniowianie Z Wikipedii W bioinformatyce, uliniawianie sekwencji to sposób porównywania sekwencji pierwszorzędowej DN, RN bądź białek w celu identyfikacji regionów podobnych, które mogą być wynikiem funkcjonalnych, strukturalnych bądź ewolucyjnych związków pomiędzy sekwencjami. Uliniowione sekwencje rezyduów nukleotydów bądź aminokwasów zwykle są przedstawiane jako wiersze macierzy Pomiędzy znaki wstawiane są przerwy w taki sposób, aby zapewnić jak największą zgodność porównywanych sekwencji....
4 dopasowanie (alignment) Ułożenie dwóch sekwencji biopolimerów (DN, RN lub białka) w celu zidentyfikowania regionów podobieństwa istotnego ze względów ewolucyjnych, strukturalnych lub funkcjonalnych (procedura oraz jej efekt). dwie sekwencje - pairwise alignment wiele sekwencji - multiple sequence alignment gap match mismatch
5 Edgar llan Poe, he Raven
6 Różne sekwencje można dopasować NOWORÓD NOWORD NEUBUR NEUHEL NRUWHI NNRHR
7 Różne sekwencje można dopasować NOWO-RÓD NOWOR-D. NEUBU-R NEUHEL
8 Różne sekwencje można dopasować NOWO-RÓD NOWOR-D N---RUWHI NN-RHR
9 Dopasowanie sekwencji - znaczenie Podobieństwo porównywanych sekwencji (similarity) może świadczyć o: podobnej funkcji sekwencji podobnej strukturze białek wspólnej historii ewolucyjnej sekwencji Podobieństwo porównywanych sekwencji (similarity) może wynikać z: homologii - pochodzeniu sekwencji (homologicznych) od wspólnego przodka; sekwencje mogą, ale nie muszą pełnić te same funkcje konwergencji - podobne motywy, które wyewoluowały w obu sekwencjach (analogicznych) niezależnie; np. chymotrypsyna i subtylizyna - różna struktura 3D, ale podobne centrum aktywne (histydyna, seryna, kwas asparaginowy) {... Problem rozróżnienia odległej homologii a analogii }
10 regiony zachowane = znaczenie funkcjonalne sekwencje trypsyny Mus musculus i stacus astacus S-S * Mouse IVYNEENSVPYQVSLNS-----YHFSLINEQWVVSHYK SRIQV rayfish IVDVLEFPYQLSFQEFLFSFHFSIYNENYIHVYDDYENPSLQI * Mouse RLEHNIEVLENEQFINKIIRHPQYDRKLNNDIMLIKLSSRVINRVSISLP rayfish VELDMSVNESEQIVSKIILHENFDYDLLDNDISLLKLSSLFNNNVPILPQ Mouse PPKLISWNSSDYPDELQLDPVLSQKESYP-KISNMFVFLE rayfish HNVIVW-SENPDVLQKVVPLVSDERDDYDEIFDSMIVPE S-S * Mouse KDSQDSPVVN----QLQVVSWDQKNKPVYKVYNYVKWIKNIN rayfish KDSQDSPLSDSYLIVSWYRPYPVYEVSYHVDWIKNV-- S-S
11 Sequence identity? L1_HUMN vs 1kapp Metalloprotease [Pseudomonas aeruginosa] Z-score % sequence identity L1_HUMN PNVPEDELIQQIKDMVQSLYLFEKRFYFKNVIL 1kapp YSFLKPNDFFNPWKYVSDIYSLKFSFSQQQQKL L1_HUMN IPEWKKDYVRPKLEYKNDVLVESPPNDEPYE 1kapp SLQSWSDVNIHFVDQDQDLFNFSSSVFF L1_HUMN QMNEKERIHLPDFIKKLEYPQRFVHEWH 1kapp LPDVPDLKQSWYLINSSYSNVNP-----QLHEIH L1_HUMN LRWVFDEYNNDE 1kapp --LSHPDYN
12 różnice między sekwencjami świadczą o mutacjach, które zaszły po rozdzieleniu się sekwencji od wspólnego przodka delecja insercja - + -> -> substytucje
13 dopasowanie - zastosowanie Porównywanie sekwencji jest bardzo pomocne w: poszukiwaniu oraz określaniu funkcji i struktury (białek) dla nowych sekwencji określaniu powiązań filogenetycznych między sekwencjami - homologii między sekwencjami oraz w analizach ewolucyjnych
14 dopasowanie - metody dopasowanie par sekwencji: Macierz punktów - dot matrix, dotplot Programowanie dynamiczne (DP) Metody słów (k - tuple methods) - szybkie metody stosowane przy przeszukiwaniu baz danych sekwencji z wykorzystaniem programów FS i BLS dopasowanie wielu sekwencji
15 Dot matrix M S I E L Q W R E V W M S I E Q W R Q V MSIELQWREVW MSIE-QWRQV--
16 Dot matrix porównanie sekwencji samej z sobą
17 Dot matrix porównanie dwóch podobnych sekwencji
18 Dot matrix - metody wstawiania punktów Dla identycznych zasad lub reszt aminokwasowych (symboli) W oparciu o macierz punktacji (PM, BLOSUM), gdy symbole wykazują podobieństwo większe niż pewna wartość punktacji (score) Nadawanie różnych kolorów lub odcieni szarości w zależności od stopnia podobieństwa symboli dy liczba podobnych symboli w danym oknie przekracza pewną wartość
19 Dot matrix - metody wstawiania punktów 5/74/74/712/20 11/20 rodzaj sekwencji sekwencje DN sekwencje białek sekwencje odległe wielkość okna liczba identycznych symboli
20 Dot matrix - zastosowanie identyfikacja regionów podobnych lub identycznych porównywanie sekwencji o strukturze wielodomenowej rozpoznawanie dużych insercji i delecji rozpoznawanie regionów powtórzonych i duplikacji rozpoznawanie rearanżacji rozpoznawanie regionów o słabo zróżnicowanym składzie analiza sekwencji i struktury RN
21 Dot matrix insercja lub delecja
22 Dot matrix duplikacja tandemowa
23 Dot matrix duplikacja tandemowa w tej samej sekwencji
24 Dot matrix inwersja
25 Dot matrix przyłączenie regionu
26 Dot matrix region o licznych powtórzeniach
27 Dot matrix - zastosowanie czynnik krzepnięcia XII (F12) F2 E F1 E K atalytic tkankowy aktywator plazminogenu (PL) F1 E K K atalytic F1 F2 E K atalytic powtórzenia fibronektynowe domena podobna do EF domena kringle aktywność katalityczna proteaz serynowych
28 Dot matrix - zastosowanie czynnik krzepnięcia XII (F12) tkankowy aktywator plazminogenu (PL) F2 E F1 E K atalytic
29 Programowanie dynamiczne Porównuje każdą parę znaków dwóch sekwencji i tworzy dopasowanie Uwzględnia wszystkie możliwe przyrównania uwzględniając: dopasowania (matches), niedopasowania (mismatches), przerwy (gaps). Przerwy są wstawiane, aby uzyskać wzrost liczby dopasowań w innych miejscach. Przyjmuje pewien system punktacji (scoring system) Rozpatruje wszystkie możliwości Stara się uzyskać maksymalną liczbę dopasowań między identycznymi lub podobnymi znakami Znajduje optymalne dopasowanie (może istnieć więcej niż jedno takie dopasowanie) zas obliczeń proporcjonalny do iloczynu długości sekwencji
30 Programowanie dynamiczne Przyjęty system punktacji: dopasowanie (match): +1 niedopasowanie (mismatch): -1 przerwa (gap): -1 gap match mismatch
31 Programowanie dynamiczne Już utworzone dopasowanie, do którego jest dodawana kolejna para znaków, musi być optymalne. Programowanie dynamiczne uwzględnia każdą dodawaną parę znaków i z powrotem przelicza optymalne dopasowanie. sekwencja 1: sekwencja 2: Dotychczasowe dopasowanie musi być optymalne! + = = = = = = +2
32 Programowanie dynamiczne worzenie macierzy punktacji, w której każda komórka reprezentuje punktację dla najlepszego dopasowania kończącego się w danej pozycji ofanie się w macierzy, aby znaleźć optymalne dopasowanie sekwencja 1: sekwencja 2: Przyjęty system punktacji: dopasowanie (match): +1 niedopasowanie (mismatch): -1 przerwa (gap): -1
33 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Znaki sekwencji ułożone wzdłuż dwu-wymiarowej siatki Węzły siatki znajdują się między znakami
34 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy elem jest znalezienie optymalnej ścieżki stąd dotąd
35 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Każda ścieżka odpowiada poszczególnemu dopasowaniu Które dopasowanie jest optymalne?
36 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy punktacja dla ścieżki jest sumą przyrastających punktacji krawędzi. porównane z match = +1
37 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy punktacja dla ścieżki jest sumą przyrastających punktacji krawędzi. porównane z mismatch = -1
38 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy punktacja dla ścieżki jest sumą przyrastających punktacji krawędzi. porównane z przerwą gap = -1 przerwa porównana z
39 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Znaczenie punktu w macierzy: wszystkie znaki do tego miejsca są ze sobą porównane, czego można dokonać na wiele sposobów (ścieżek). Pozycja x oznacza: przyrównanie z x
40 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji
41 0 0-1 = = =
42 0 0-1 = = = = = =
43 0 0-1 = = = = = = = =
44 0 0-1 = = = = = = = = = =
45 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji
46 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji. Wybierana jest najlepsza ścieżka prowadząca do każdego punktu na siatce
47 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji. Wybierana jest najlepsza ścieżka prowadząca do każdego punktu na siatce
48 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji. Wybierana jest najlepsza ścieżka prowadząca do każdego punktu na siatce
49 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji. Wybierana jest najlepsza ścieżka prowadząca do każdego punktu na siatce
50 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji. Wybierana jest najlepsza ścieżka prowadząca do każdego punktu na siatce
51 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Rozszerzanie się ścieżki i przyrastanie punktacji. Wybierana jest najlepsza ścieżka prowadząca do każdego punktu na siatce
52 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy ofanie się do tyłu i wybór optymalnej ścieżki i dopasowania
53 Programowanie dynamiczne dopasowanie jako ścieżka w macierzy Uzyskane dopasowanie -
54 dopasowanie globalne i lokalne dopasowanie globalne (Needleman & Wunsch, 1970) przyrównuje sekwencje na całej długości; wykorzystuje tak dużo znaków, jak to jest tylko możliwe. dopasowanie lokalne (Smith & Waterman, 1981) przyrównuje fragmenty sekwencji, które wykazują największe podobieństwo; poszukuje najlepiej pasujących regionów; znajduje regiony konserwowane. dy obliczana wartość punktacji w macierzy jest mniejsza od zera, to wartość ta jest ustawiana na zero, a dopasowanie ulega zakończeniu do tego miejsca i rozpoczynany jest nowe` dopasowanie od nowego miejsca Optimalne dopasowanie globalne LPSSKQKS-SRIWDN LN-IKSKIMRLD sekwencje o podobnej długości, blisko spokrewnione Optymal ne dopasowanie lolalne K K sekwencje o różnych długościach, posiadające regiony i domeny zachowane, podobne tylko w niektórych obszarach
55 System punktacji Prosty system punktacji: match: mismatch: -1 0 gap: -1-1 Zaawansowany system punktacji (nadawanie różnych wag dla niedopasowań i przerw w zależnosci od ich długości): Macierze podstawień aminokwasów (PM, BLOSUM) Macierze podstawień nukleotydów System punktacji dla przerw: gap penalties, affine gap penalty
56 ypowe systemy punktacji sekwencje DN Match = +1 Mismatch = -3 ap penalty = -5 ap extension penalty = -2 sekwencje białkowe Macierz Blossum62 ap open penalty = -11 ap extension = -1
57 Istotność dopasowania zy punktacja dopasowanie jest znacząco większa od punktacji oczekiwanej dla dopasowania losowych sekwencji o tej samej długości i składzie? worzenie metodą Monte arlo losowych(-ej) sekwencji (o tej samej długości i składzie co rzeczywiste). Przyrównanie losowych(-ej) sekwencji (powtórzenie razy) przy tych samych parametrach. Określenie rozkładu punktacji, średniej i odchylenie standardowego (SD). Wyliczenie Z-score: Z = (score obs score ran )/SD ran 3 > Z brak homologii 3 < Z < 6 istnieje homologia Z > 6 silna homologia Rozkład score-ów nie jest normalny i dlatego nie można przekształcić Z-score na prawdopodobieństwo.
58 Istotność dopasowania Dla dopasowań lokalnych rozkład maksymalnych scorów dopasowania dla sekwencji losowych przyjmuje rozkład wartości ekstremalnych (extreme values distribution)(karlin i ltschul 1990). Log p(s>=s ) S Wykres zależności logarytmu ułamka dopasowań z punktacją S równą lub większą od wartości punktacji S Określenie E-value Oczekiwana liczba przypadkowych dopasowań z punktacją większą niż obserwowana E=Kmn e λs Oczekiwana (wg rozkładu prawdopodobieństwa) liczba dopasowań z punktacją równą przynajmniej S
59 Bit score- znormalizowana punktacja uwzględniająca warunki jego naliczania i przyjęte systemy punktacji (parametry lambda i K) E=mn 2 S' λs ln K S'= ln2
60 Prawdopodobieństwo znalezienia przynajmniej 1 dopasowania o punktacji >=S p=1 e E Jeżeli spodziewamy się znaleźć przynajmniej 3 dopasowania o punktacji >= S, to prawdopodobieństwo tego że znajdziemy co najmniej jedno wynosi 0,95. Programy z grupy BLS posługują się wartością E zamiast bezpośrednim prawdopodobieństwem dla wygody przy analizie wyników
61 Istotność dopasowania alignment significance Łatwiej oszacować istotność statystyczną rudniej ocenić istotność biologiczną
Dopasowanie sekwencji Sequence alignment. Bioinformatyka, wykłady 3 i 4 (19, 26.X.2010)
 Dopasowanie sekwencji Sequence alignment Bioinformatyka, wykłady 3 i 4 (19, 26.X.2010) krzysztof_pawlowski@sggw.pl terminologia alignment 33000 dopasowanie sekwencji 119 uliniowienie sekwencji 82 uliniowianie
Dopasowanie sekwencji Sequence alignment Bioinformatyka, wykłady 3 i 4 (19, 26.X.2010) krzysztof_pawlowski@sggw.pl terminologia alignment 33000 dopasowanie sekwencji 119 uliniowienie sekwencji 82 uliniowianie
PRZYRÓWNANIE SEKWENCJI
 http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
Przyrównanie sekwencji. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Przyrównanie sekwencji Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych),
Przyrównanie sekwencji Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych),
Bioinformatyka. (wykład monograficzny) wykład 5. E. Banachowicz. Zakład Biofizyki Molekularnej IF UAM
 Bioinformatyka (wykład monograficzny) wykład 5. E. Banachowicz Zakład Biofizyki Molekularnej IF UM http://www.amu.edu.pl/~ewas lgorytmy macierze punktowe (DotPlot) programowanie dynamiczne metody heurystyczne
Bioinformatyka (wykład monograficzny) wykład 5. E. Banachowicz Zakład Biofizyki Molekularnej IF UM http://www.amu.edu.pl/~ewas lgorytmy macierze punktowe (DotPlot) programowanie dynamiczne metody heurystyczne
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
Dopasowanie sekwencji (sequence alignment)
 Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Dopasowania par sekwencji DNA
 Dopasowania par sekwencji DNA Tworzenie uliniowień (dopasowań, tzw. alignmentów ) par sekwencji PSA Pairwise Sequence Alignment Dopasowania globalne i lokalne ACTACTAGATTACTTACGGATCAGGTACTTTAGAGGCTTGCAACCA
Dopasowania par sekwencji DNA Tworzenie uliniowień (dopasowań, tzw. alignmentów ) par sekwencji PSA Pairwise Sequence Alignment Dopasowania globalne i lokalne ACTACTAGATTACTTACGGATCAGGTACTTTAGAGGCTTGCAACCA
Politechnika Wrocławska. Dopasowywanie sekwencji Sequence alignment
 Dopasowywanie sekwencji Sequence alignment Drzewo filogenetyczne Kserokopiarka zadanie: skopiować 300 stron. Co może pójść źle? 2x ta sama strona Opuszczona strona Nadmiarowa pusta strona Strona do góry
Dopasowywanie sekwencji Sequence alignment Drzewo filogenetyczne Kserokopiarka zadanie: skopiować 300 stron. Co może pójść źle? 2x ta sama strona Opuszczona strona Nadmiarowa pusta strona Strona do góry
Bioinformatyka. Porównywanie sekwencji
 Bioinformatyka Wykład 5 E. Banachowicz Zakład Biofizyki Molekularnej IF UM 1 http://www.amu.edu.pl/~ewas Porównywanie sekwencji Pierwsze pytanie biologa molekularnego, kiedy odkryje nową sekwencję: zy
Bioinformatyka Wykład 5 E. Banachowicz Zakład Biofizyki Molekularnej IF UM 1 http://www.amu.edu.pl/~ewas Porównywanie sekwencji Pierwsze pytanie biologa molekularnego, kiedy odkryje nową sekwencję: zy
Dopasowanie sekwencji c.d. Sequence alignment. Bioinformatyka, wykład 5 (6.XI.2012) krzysztof_pawlowski@sggw.pl
 Dopasowanie sekwencji c.d. Sequence alignment Bioinformatyka, wykład 5 (6.XI.2012) krzysztof_pawlowski@sggw.pl Dopasowanie sekwencji - znaczenie Podobieństwo porównywanych sekwencji (similarity) może świadczyć
Dopasowanie sekwencji c.d. Sequence alignment Bioinformatyka, wykład 5 (6.XI.2012) krzysztof_pawlowski@sggw.pl Dopasowanie sekwencji - znaczenie Podobieństwo porównywanych sekwencji (similarity) może świadczyć
Dopasowywanie sekwencji (ang. sequence alignment) Metody dopasowywania sekwencji. Homologia a podobieństwo sekwencji. Rodzaje dopasowania
 Wprowadzenie do Informatyki Biomedycznej Wykład 2: Metody dopasowywania sekwencji Wydział Informatyki PB Dopasowywanie sekwencji (ang. sequence alignment) Dopasowywanie (przyrównywanie) sekwencji polega
Wprowadzenie do Informatyki Biomedycznej Wykład 2: Metody dopasowywania sekwencji Wydział Informatyki PB Dopasowywanie sekwencji (ang. sequence alignment) Dopasowywanie (przyrównywanie) sekwencji polega
Wykład 5 Dopasowywanie lokalne
 Wykład 5 Dopasowywanie lokalne Dopasowanie par (sekwencji) Dopasowanie globalne C A T W A L K C A T W A L K C O W A R D C X X O X W X A X R X D X Globalne dopasowanie Schemat punktowania (uproszczony)
Wykład 5 Dopasowywanie lokalne Dopasowanie par (sekwencji) Dopasowanie globalne C A T W A L K C A T W A L K C O W A R D C X X O X W X A X R X D X Globalne dopasowanie Schemat punktowania (uproszczony)
dopasowanie sekwencji Porównywanie sekwencji Etapy dopasowywania sekwencji Homologia, podobieństwo i analogia
 Porównywanie sekwencji Homologia, podobieństwo i analogia dopasowanie sekwencji Dopasowanie/porównywanie Uliniowienie Alignment W bioinformatyce, dopasowanie sekwencji jest sposobem dopasowania struktur
Porównywanie sekwencji Homologia, podobieństwo i analogia dopasowanie sekwencji Dopasowanie/porównywanie Uliniowienie Alignment W bioinformatyce, dopasowanie sekwencji jest sposobem dopasowania struktur
Dopasowanie sekwencji c.d. Sequence alignment. Bioinformatyka, wykład 5 (16.XI.2010) krzysztof_pawlowski@sggw.pl
 Dopasowanie sekwencji c.d. Sequence alignment Bioinformatyka, wykład 5 (16.XI.2010) krzysztof_pawlowski@sggw.pl dopasowanie - metody dopasowanie par sekwencji: Macierz punktów - dot matrix, dotplot Programowanie
Dopasowanie sekwencji c.d. Sequence alignment Bioinformatyka, wykład 5 (16.XI.2010) krzysztof_pawlowski@sggw.pl dopasowanie - metody dopasowanie par sekwencji: Macierz punktów - dot matrix, dotplot Programowanie
Porównywanie i dopasowywanie sekwencji
 Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
Porównywanie sekwencji białkowych
 Bioinformatyka -9 Bioinformatyka Wykład 4. E. Banachowicz Zakład Biofizyki Molekularnej http://www.amu.edu.pl/~ewas Porównywanie sekwencji białkowych Wykład 4, Bioinformatyka -9 Porównywanie sekwencji
Bioinformatyka -9 Bioinformatyka Wykład 4. E. Banachowicz Zakład Biofizyki Molekularnej http://www.amu.edu.pl/~ewas Porównywanie sekwencji białkowych Wykład 4, Bioinformatyka -9 Porównywanie sekwencji
Porównywanie i dopasowywanie sekwencji
 Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek narodziła się nowa dyscyplina nauki ewolucja molekularna Ewolucja molekularna
Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek narodziła się nowa dyscyplina nauki ewolucja molekularna Ewolucja molekularna
Przyrównywanie sekwencji
 Instytut Informatyki i Matematyki Komputerowej UJ, opracowanie: mgr Ewa Matczyńska, dr Jacek Śmietański Przyrównywanie sekwencji 1. Porównywanie sekwencji wprowadzenie Sekwencje porównujemy po to, aby
Instytut Informatyki i Matematyki Komputerowej UJ, opracowanie: mgr Ewa Matczyńska, dr Jacek Śmietański Przyrównywanie sekwencji 1. Porównywanie sekwencji wprowadzenie Sekwencje porównujemy po to, aby
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Często dopasować chcemy nie dwie sekwencje ale kilkanaście lub więcej 2 Istnieją dokładne algorytmy, lecz są one niewydajne
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Często dopasować chcemy nie dwie sekwencje ale kilkanaście lub więcej 2 Istnieją dokładne algorytmy, lecz są one niewydajne
Spis treści. Przedmowa... XI. Wprowadzenie i biologiczne bazy danych. 1 Wprowadzenie... 3. 2 Wprowadzenie do biologicznych baz danych...
 Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Homologia, podobieństwo i analogia
 Porównywanie sekwencji Homologia, podobieństwo i analogia Homologi Ortologi homologiczne geny, których rozdzielenie nastąpiło na skutek specjacji, czyli rozdzielenia gatunków, lub rzadziej horyzontalnego
Porównywanie sekwencji Homologia, podobieństwo i analogia Homologi Ortologi homologiczne geny, których rozdzielenie nastąpiło na skutek specjacji, czyli rozdzielenia gatunków, lub rzadziej horyzontalnego
Dopasowanie par sekwencji
 BIOINFORMTYK edycja 2016 / 2017 wykład 3 Dopasowanie par sekwencji dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Idea i cele dopasowania sekwencji 2. Definicje
BIOINFORMTYK edycja 2016 / 2017 wykład 3 Dopasowanie par sekwencji dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Idea i cele dopasowania sekwencji 2. Definicje
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
Motywy i podobieństwo
 Motywy i podobieństwo Całość funkcja Modularna budowa białek Elementy składowe czyli miejsca wiązania, domeny 1 Motywy Motyw jest opisem określonej części trójwymiarowej struktury zawierającym charakterystyczny
Motywy i podobieństwo Całość funkcja Modularna budowa białek Elementy składowe czyli miejsca wiązania, domeny 1 Motywy Motyw jest opisem określonej części trójwymiarowej struktury zawierającym charakterystyczny
PODSTAWY BIOINFORMATYKI 8 DOPASOWYWANIE SEKWENCJI AMINOKWASÓW
 PODSTAWY BIOINFORMATYKI 8 DOPASOWYWANIE SEKWENCJI AMINOKWASÓW DOPASOWYWANIE SEKWENCJI 1. Miary podobieństwa sekwencji aminokwasów 2. Zastosowanie programów: CLUSTAL OMEGA BLAST Copyright 2013, Joanna Szyda
PODSTAWY BIOINFORMATYKI 8 DOPASOWYWANIE SEKWENCJI AMINOKWASÓW DOPASOWYWANIE SEKWENCJI 1. Miary podobieństwa sekwencji aminokwasów 2. Zastosowanie programów: CLUSTAL OMEGA BLAST Copyright 2013, Joanna Szyda
Wstęp do Biologii Obliczeniowej
 Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Bioinformatyka 2 (BT172) Progresywne metody wyznaczania MSA: T-coffee
 Bioinformatyka 2 (BT172) Wykład 5 Progresywne metody wyznaczania MSA: T-coffee Krzysztof Murzyn 14.XI.2005 PLAN WYKŁADU Ostatnio : definicje, zastosowania MSA, złożoność obliczeniowa algorytmu wyznaczania
Bioinformatyka 2 (BT172) Wykład 5 Progresywne metody wyznaczania MSA: T-coffee Krzysztof Murzyn 14.XI.2005 PLAN WYKŁADU Ostatnio : definicje, zastosowania MSA, złożoność obliczeniowa algorytmu wyznaczania
prof. dr hab. inż. Marta Kasprzak Instytut Informatyki, Politechnika Poznańska Dopasowanie sekwencji
 Bioinformatyka wykład 5: dopasowanie sekwencji prof. dr hab. inż. Marta Kasprzak Instytut Informatyk Politechnika Poznańska Dopasowanie sekwencji Badanie podobieństwa sekwencji stanowi podstawę wielu gałęzi
Bioinformatyka wykład 5: dopasowanie sekwencji prof. dr hab. inż. Marta Kasprzak Instytut Informatyk Politechnika Poznańska Dopasowanie sekwencji Badanie podobieństwa sekwencji stanowi podstawę wielu gałęzi
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Wyszukiwanie sekwencji Jak wyszukad z baz danych bioinformatycznych sekwencje podobne do sekwencji zadanej (ang. query
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Wyszukiwanie sekwencji Jak wyszukad z baz danych bioinformatycznych sekwencje podobne do sekwencji zadanej (ang. query
Statystyczna analiza danych
 Statystyczna analiza danych ukryte modele Markowa, zastosowania Anna Gambin Instytut Informatyki Uniwersytet Warszawski plan na dziś Ukryte modele Markowa w praktyce modelowania rodzin białek multiuliniowienia
Statystyczna analiza danych ukryte modele Markowa, zastosowania Anna Gambin Instytut Informatyki Uniwersytet Warszawski plan na dziś Ukryte modele Markowa w praktyce modelowania rodzin białek multiuliniowienia
Generator testów Bioinformatyka wer / 0 Strona: 1
 Przedmiot: Nazwa przedmiotu Nazwa testu: Bioinformatyka wer. 1.0.6 Nr testu 0 Klasa: V zaoczne WNB UZ Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Analiza porównawcza białek zwykle zaczyna się na badaniach
Przedmiot: Nazwa przedmiotu Nazwa testu: Bioinformatyka wer. 1.0.6 Nr testu 0 Klasa: V zaoczne WNB UZ Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Analiza porównawcza białek zwykle zaczyna się na badaniach
Porównywanie sekwencji białek i kwasów nukleinowych
 Porównywanie sekwencji białek i kwasów nukleinowych Krzysztof Lewiński 1. Podobieństwo i jego miara Wprawdzie podobieństwo jest pojęciem często używanym w życiu codziennym ale nie oznacza to, że możemy
Porównywanie sekwencji białek i kwasów nukleinowych Krzysztof Lewiński 1. Podobieństwo i jego miara Wprawdzie podobieństwo jest pojęciem często używanym w życiu codziennym ale nie oznacza to, że możemy
BIOINFORMATYKA. edycja 2016 / wykład 11 RNA. dr Jacek Śmietański
 BIOINFORMATYKA edycja 2016 / 2017 wykład 11 RNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Rola i rodzaje RNA 2. Oddziaływania wewnątrzcząsteczkowe i struktury
BIOINFORMATYKA edycja 2016 / 2017 wykład 11 RNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Rola i rodzaje RNA 2. Oddziaływania wewnątrzcząsteczkowe i struktury
Genomika Porównawcza. Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski
 Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
3 Przeszukiwanie baz danych
 Spis treści 3 Przeszukiwanie baz danych 1 3.1 Heurystyczne algorytmy...................... 1 3.1.1 FASTA........................... 1 3.1.2 BLAST........................... 3 3.2 Macierze substytucyjne.......................
Spis treści 3 Przeszukiwanie baz danych 1 3.1 Heurystyczne algorytmy...................... 1 3.1.1 FASTA........................... 1 3.1.2 BLAST........................... 3 3.2 Macierze substytucyjne.......................
Algorytmy kombinatoryczne w bioinformatyce
 lgorytmy kombinatoryczne w bioinformatyce wykład 4: dopasowanie sekwencj poszukiwanie motywów prof. dr hab. inż. Marta Kasprzak Instytut Informatyk Politechnika Poznańska Dopasowanie sekwencji Badanie
lgorytmy kombinatoryczne w bioinformatyce wykład 4: dopasowanie sekwencj poszukiwanie motywów prof. dr hab. inż. Marta Kasprzak Instytut Informatyk Politechnika Poznańska Dopasowanie sekwencji Badanie
Bioinformatyka II Modelowanie struktury białek
 Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania? Dlaczego? Struktura krystaliczną czy NMR (to samo białko, ta sama rozdzielczość)? Strukturę
Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania? Dlaczego? Struktura krystaliczną czy NMR (to samo białko, ta sama rozdzielczość)? Strukturę
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Filogenetyka molekularna wykorzystuje informację zawartą w sekwencjach aminokwasów lub nukleotydów do kontrukcji drzew
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Filogenetyka molekularna wykorzystuje informację zawartą w sekwencjach aminokwasów lub nukleotydów do kontrukcji drzew
Algorytmy kombinatoryczne w bioinformatyce
 lgorytmy kombinatoryczne w bioinformatyce wykład 4: dopasowanie sekwencj poszukiwanie motywów prof. dr hab. inż. Marta Kasprzak Instytut Informatyk Politechnika Poznańska Dopasowanie sekwencji Badanie
lgorytmy kombinatoryczne w bioinformatyce wykład 4: dopasowanie sekwencj poszukiwanie motywów prof. dr hab. inż. Marta Kasprzak Instytut Informatyk Politechnika Poznańska Dopasowanie sekwencji Badanie
Bioinformatyka. Ocena wiarygodności dopasowania sekwencji.
 Bioinformatyka Ocena wiarygodności dopasowania sekwencji www.michalbereta.pl Załóżmy, że mamy dwie sekwencje, które chcemy dopasować i dodatkowo ocenić wiarygodność tego dopasowania. Interesujące nas pytanie
Bioinformatyka Ocena wiarygodności dopasowania sekwencji www.michalbereta.pl Załóżmy, że mamy dwie sekwencje, które chcemy dopasować i dodatkowo ocenić wiarygodność tego dopasowania. Interesujące nas pytanie
Generator testów 1.3.1 Bioinformatyka_zdalne wer. 1.0.13 / 0 Strona: 1
 Przedmiot: Bioinformatyka Nazwa testu: Bioinformatyka_zdalne wer. 1.0.13 Nr testu 0 Klasa: WNB UZ Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Model Markowa substytucji aminokwasów w mutagenezie białek zakłada...
Przedmiot: Bioinformatyka Nazwa testu: Bioinformatyka_zdalne wer. 1.0.13 Nr testu 0 Klasa: WNB UZ Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Model Markowa substytucji aminokwasów w mutagenezie białek zakłada...
D: Dopasowanie sekwencji. Programowanie dynamiczne
 D: Dopasowanie sekwencji. Programowanie dynamiczne Problem: jak porównywać sekwencje DNA? Czy te sekwencje są podobne? Jeśli są podobne, to jak mierzyć to podobieństwo? Odpowiedzi są kluczowe dla konstrukcji
D: Dopasowanie sekwencji. Programowanie dynamiczne Problem: jak porównywać sekwencje DNA? Czy te sekwencje są podobne? Jeśli są podobne, to jak mierzyć to podobieństwo? Odpowiedzi są kluczowe dla konstrukcji
Wykład 10 2008-04-30. Bioinformatyka. Wykład 9. E. Banachowicz. Zakład Biofizyki Molekularnej IF UAM
 Bioinformatyka Wykład 9 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Konsekwencje zestawieo wielu sekwencji - rodziny białkowe, domeny, motywy i wzorce 2 Bioinformatyka,
Bioinformatyka Wykład 9 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Konsekwencje zestawieo wielu sekwencji - rodziny białkowe, domeny, motywy i wzorce 2 Bioinformatyka,
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
Podstawy bioinformatyki dla biotechnologów
 dla biotechnologów Wykład 3 alignment Wykład 2 Porównywanie sekwencji Homologia, podobieństwo i analogia Wykład 2; slajd 2 Duplikacja, specjacja Wykład 2; slajd 3 Homologi Ortologi homologiczne geny, których
dla biotechnologów Wykład 3 alignment Wykład 2 Porównywanie sekwencji Homologia, podobieństwo i analogia Wykład 2; slajd 2 Duplikacja, specjacja Wykład 2; slajd 3 Homologi Ortologi homologiczne geny, których
Spis treści 8 Ewolucja molekularna... 87. 9 Ewolucyjne podstawy porównywania sekwencji... 87. 9.1 Identyfikacja sekwencji i jej funkcji...
 Spis treści 8 Ewolucja molekularna... 87 9 Ewolucyjne podstawy porównywania sekwencji... 87 9.1 Identyfikacja sekwencji i jej funkcji... 87 9.2 Homologia... 88 9.3 Modele ewolucji sekwencji białkowej...
Spis treści 8 Ewolucja molekularna... 87 9 Ewolucyjne podstawy porównywania sekwencji... 87 9.1 Identyfikacja sekwencji i jej funkcji... 87 9.2 Homologia... 88 9.3 Modele ewolucji sekwencji białkowej...
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych???
 Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
1. Eliminuje się ze zbioru potencjalnych zmiennych te zmienne dla których korelacja ze zmienną objaśnianą jest mniejsza od krytycznej:
 Metoda analizy macierzy współczynników korelacji Idea metody sprowadza się do wyboru takich zmiennych objaśniających, które są silnie skorelowane ze zmienną objaśnianą i równocześnie słabo skorelowane
Metoda analizy macierzy współczynników korelacji Idea metody sprowadza się do wyboru takich zmiennych objaśniających, które są silnie skorelowane ze zmienną objaśnianą i równocześnie słabo skorelowane
Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2
 ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
Bioinformatyka wykład 8, 27.XI.2012
 Bioinformatyka wykład 8, 27.XI.2012 białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2013-01-21 1 Plan wykładu regiony nieuporządkowane sposoby przedstawienia struktur białkowych powierzchnia
Bioinformatyka wykład 8, 27.XI.2012 białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2013-01-21 1 Plan wykładu regiony nieuporządkowane sposoby przedstawienia struktur białkowych powierzchnia
Zadanie 1 Zakładając liniową relację między wydatkami na obuwie a dochodem oszacować MNK parametry modelu: y t. X 1 t. Tabela 1.
 tel. 44 683 1 55 tel. kom. 64 566 811 e-mail: biuro@wszechwiedza.pl Zadanie 1 Zakładając liniową relację między wydatkami na obuwie a dochodem oszacować MNK parametry modelu: gdzie: y t X t y t = 1 X 1
tel. 44 683 1 55 tel. kom. 64 566 811 e-mail: biuro@wszechwiedza.pl Zadanie 1 Zakładając liniową relację między wydatkami na obuwie a dochodem oszacować MNK parametry modelu: gdzie: y t X t y t = 1 X 1
SMOP - wykład. Rozkład normalny zasady przenoszenia błędów. Ewa Pawelec
 SMOP - wykład Rozkład normalny zasady przenoszenia błędów Ewa Pawelec 1 iepewność dla rozkładu norm. Zamiast dodawania całych zakresów uwzględniamy prawdopodobieństwo trafienia dwóch wartości: P x 1, x
SMOP - wykład Rozkład normalny zasady przenoszenia błędów Ewa Pawelec 1 iepewność dla rozkładu norm. Zamiast dodawania całych zakresów uwzględniamy prawdopodobieństwo trafienia dwóch wartości: P x 1, x
Bioinformatyka II Modelowanie struktury białek
 Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania dla każdego z podanych przypadków? Dlaczego? Struktura krystaliczną czy NMR (to samo białko,
Bioinformatyka II Modelowanie struktury białek 1. Który spośród wymienionych szablonów wybierzesz do modelowania dla każdego z podanych przypadków? Dlaczego? Struktura krystaliczną czy NMR (to samo białko,
Wstęp do programowania
 Wstęp do programowania Programowanie dynamiczne Paweł Daniluk Wydział Fizyki Jesień 2013 P. Daniluk(Wydział Fizyki) WP w. X Jesień 2013 1 / 21 Dziel i zwyciężaj przypomnienie 1 Podział problemu na 2 lub
Wstęp do programowania Programowanie dynamiczne Paweł Daniluk Wydział Fizyki Jesień 2013 P. Daniluk(Wydział Fizyki) WP w. X Jesień 2013 1 / 21 Dziel i zwyciężaj przypomnienie 1 Podział problemu na 2 lub
Bioinformatyka. Podsumowanie algorytmów dynamicznych
 Bioinformatyka Wykład 5 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Podsumowanie algorytmów dynamicznych Algorytmy porównywania sekwencji oparte na programowaniu dynamicznym
Bioinformatyka Wykład 5 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Podsumowanie algorytmów dynamicznych Algorytmy porównywania sekwencji oparte na programowaniu dynamicznym
Modelowanie motywów łańcuchami Markowa wyższego rzędu
 Modelowanie motywów łańcuchami Markowa wyższego rzędu Uniwersytet Warszawski Wydział Matematyki, Informatyki i Mechaniki 23 października 2008 roku Plan prezentacji 1 Źródła 2 Motywy i ich znaczenie Łańcuchy
Modelowanie motywów łańcuchami Markowa wyższego rzędu Uniwersytet Warszawski Wydział Matematyki, Informatyki i Mechaniki 23 października 2008 roku Plan prezentacji 1 Źródła 2 Motywy i ich znaczenie Łańcuchy
Grafy i sieci wybrane zagadnienia wykład 3: modele służące porównywaniu sieci
 Grafy i sieci wybrane zagadnienia wykład 3: modele służące porównywaniu sieci prof. dr hab. inż. Marta Kasprzak Instytut Informatyki, Politechnika Poznańska Plan wykładu 1. Sieci jako modele interakcji
Grafy i sieci wybrane zagadnienia wykład 3: modele służące porównywaniu sieci prof. dr hab. inż. Marta Kasprzak Instytut Informatyki, Politechnika Poznańska Plan wykładu 1. Sieci jako modele interakcji
Działanie algorytmu oparte jest na minimalizacji funkcji celu jako suma funkcji kosztu ( ) oraz funkcji heurystycznej ( ).
 Algorytm A* Opracowanie: Joanna Raczyńska 1.Wstęp Algorytm A* jest heurystycznym algorytmem służącym do znajdowania najkrótszej ścieżki w grafie. Jest to algorytm zupełny i optymalny, co oznacza, że zawsze
Algorytm A* Opracowanie: Joanna Raczyńska 1.Wstęp Algorytm A* jest heurystycznym algorytmem służącym do znajdowania najkrótszej ścieżki w grafie. Jest to algorytm zupełny i optymalny, co oznacza, że zawsze
Wstęp do programowania
 Wstęp do programowania Algorytmy zachłanne, programowanie dynamiczne Paweł Daniluk Wydział Fizyki Jesień 2014 P. Daniluk(Wydział Fizyki) WP w. IX Jesień 2014 1 / 26 Algorytmy zachłanne Strategia polegająca
Wstęp do programowania Algorytmy zachłanne, programowanie dynamiczne Paweł Daniluk Wydział Fizyki Jesień 2014 P. Daniluk(Wydział Fizyki) WP w. IX Jesień 2014 1 / 26 Algorytmy zachłanne Strategia polegająca
WIZUALIZACJA ALGORYTMÓW OPTYMALNEGO DOPASOWANIA SEKWENCJI NUKLEOTYDÓW I AMINOKWASÓW
 STUDIA INFORMATICA 2011 Volume 32 Number 2A (96) Adam SKOWRON, Dariusz MROZEK Politechnika Śląska, Instytut Informatyki WIZUALIZACJA ALGORYTMÓW OPTYMALNEGO DOPASOWANIA SEKWENCJI NUKLEOTYDÓW I AMINOKWASÓW
STUDIA INFORMATICA 2011 Volume 32 Number 2A (96) Adam SKOWRON, Dariusz MROZEK Politechnika Śląska, Instytut Informatyki WIZUALIZACJA ALGORYTMÓW OPTYMALNEGO DOPASOWANIA SEKWENCJI NUKLEOTYDÓW I AMINOKWASÓW
KORELACJE I REGRESJA LINIOWA
 KORELACJE I REGRESJA LINIOWA Korelacje i regresja liniowa Analiza korelacji: Badanie, czy pomiędzy dwoma zmiennymi istnieje zależność Obie analizy się wzajemnie przeplatają Analiza regresji: Opisanie modelem
KORELACJE I REGRESJA LINIOWA Korelacje i regresja liniowa Analiza korelacji: Badanie, czy pomiędzy dwoma zmiennymi istnieje zależność Obie analizy się wzajemnie przeplatają Analiza regresji: Opisanie modelem
Rozdział 7 ZARZĄDZANIE PROJEKTAMI
 Wprowadzenie do badań operacyjnych z komputerem Opisy programów, ćwiczenia komputerowe i zadania. T. Trzaskalik (red.) Rozdział 7 ZARZĄDZANIE PROJEKTAMI 7.2. Ćwiczenia komputerowe Ćwiczenie 7.1 Wykorzystując
Wprowadzenie do badań operacyjnych z komputerem Opisy programów, ćwiczenia komputerowe i zadania. T. Trzaskalik (red.) Rozdział 7 ZARZĄDZANIE PROJEKTAMI 7.2. Ćwiczenia komputerowe Ćwiczenie 7.1 Wykorzystując
W kierunku równoległej implementacji pakietu T-Coffee
 W kierunku równoległej implementacji pakietu T-Coffee Adrian Rospondek 1 1 Wydział Inżynierii Mechanicznej i Informatyki Kierunek Informatyka, Rok V a.rospondek@poczta.fm Streszczenie Artykuł ten prezentuje
W kierunku równoległej implementacji pakietu T-Coffee Adrian Rospondek 1 1 Wydział Inżynierii Mechanicznej i Informatyki Kierunek Informatyka, Rok V a.rospondek@poczta.fm Streszczenie Artykuł ten prezentuje
Generator testów bioinformatyka wer / Strona: 1
 Przedmiot: wyklad monograficzny Nazwa testu: bioinformatyka wer. 1.0.6 Nr testu 10469906 Klasa: 5 IBOS Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Aminokwas jest to związek organiczny zawierający A) grupę
Przedmiot: wyklad monograficzny Nazwa testu: bioinformatyka wer. 1.0.6 Nr testu 10469906 Klasa: 5 IBOS Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Aminokwas jest to związek organiczny zawierający A) grupę
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych???
 Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Wykład Bioinformatyka 2012-09-24. Bioinformatyka. Wykład 7. E. Banachowicz. Zakład Biofizyki Molekularnej IF UAM. Ewolucyjne podstawy Bioinformatyki
 Bioinformatyka Wykład 7 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Plan Bioinformatyka Ewolucyjne podstawy Bioinformatyki Filogenetyka Bioinformatyczne narzędzia
Bioinformatyka Wykład 7 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Plan Bioinformatyka Ewolucyjne podstawy Bioinformatyki Filogenetyka Bioinformatyczne narzędzia
STATYSTYKA MATEMATYCZNA WYKŁAD 4. Testowanie hipotez Estymacja parametrów
 STATYSTYKA MATEMATYCZNA WYKŁAD 4 Testowanie hipotez Estymacja parametrów WSTĘP 1. Testowanie hipotez Błędy związane z testowaniem hipotez Etapy testowana hipotez Testowanie wielokrotne 2. Estymacja parametrów
STATYSTYKA MATEMATYCZNA WYKŁAD 4 Testowanie hipotez Estymacja parametrów WSTĘP 1. Testowanie hipotez Błędy związane z testowaniem hipotez Etapy testowana hipotez Testowanie wielokrotne 2. Estymacja parametrów
Bioinformatyka wykład 10
 Bioinformatyka wykład 10 21.XII.2010 białkowa bioinformatyka strukturalna, c.d. krzysztof_pawlowski@sggw.pl 2011-01-17 1 Regiony nieuporządkowane disordered regions trudna definicja trudne do przewidzenia
Bioinformatyka wykład 10 21.XII.2010 białkowa bioinformatyka strukturalna, c.d. krzysztof_pawlowski@sggw.pl 2011-01-17 1 Regiony nieuporządkowane disordered regions trudna definicja trudne do przewidzenia
MultiSETTER: web server for multiple RNA structure comparison. Sandra Sobierajska Uniwersytet Jagielloński
 MultiSETTER: web server for multiple RNA structure comparison Sandra Sobierajska Uniwersytet Jagielloński Wprowadzenie Budowa RNA: - struktura pierwszorzędowa sekwencja nukleotydów w łańcuchu: A, U, G,
MultiSETTER: web server for multiple RNA structure comparison Sandra Sobierajska Uniwersytet Jagielloński Wprowadzenie Budowa RNA: - struktura pierwszorzędowa sekwencja nukleotydów w łańcuchu: A, U, G,
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
Wykład 4. Plan: 1. Aproksymacja rozkładu dwumianowego rozkładem normalnym. 2. Rozkłady próbkowe. 3. Centralne twierdzenie graniczne
 Wykład 4 Plan: 1. Aproksymacja rozkładu dwumianowego rozkładem normalnym 2. Rozkłady próbkowe 3. Centralne twierdzenie graniczne Przybliżenie rozkładu dwumianowego rozkładem normalnym Niech Y ma rozkład
Wykład 4 Plan: 1. Aproksymacja rozkładu dwumianowego rozkładem normalnym 2. Rozkłady próbkowe 3. Centralne twierdzenie graniczne Przybliżenie rozkładu dwumianowego rozkładem normalnym Niech Y ma rozkład
Konstruowanie drzew filogenetycznych. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
; B = Wykonaj poniższe obliczenia: Mnożenia, transpozycje etc wykonuję programem i przepisuję wyniki. Mam nadzieję, że umiesz mnożyć macierze...
 Tekst na niebiesko jest komentarzem lub treścią zadania. Zadanie. Dane są macierze: A D 0 ; E 0 0 0 ; B 0 5 ; C Wykonaj poniższe obliczenia: 0 4 5 Mnożenia, transpozycje etc wykonuję programem i przepisuję
Tekst na niebiesko jest komentarzem lub treścią zadania. Zadanie. Dane są macierze: A D 0 ; E 0 0 0 ; B 0 5 ; C Wykonaj poniższe obliczenia: 0 4 5 Mnożenia, transpozycje etc wykonuję programem i przepisuję
STATYSTYKA I DOŚWIADCZALNICTWO Wykład 4
 STATYSTYKA I DOŚWIADCZALNICTWO Wykład 4 Inne układy doświadczalne 1) Układ losowanych bloków Stosujemy, gdy podejrzewamy, że może występować systematyczna zmienność między powtórzeniami np. - zmienność
STATYSTYKA I DOŚWIADCZALNICTWO Wykład 4 Inne układy doświadczalne 1) Układ losowanych bloków Stosujemy, gdy podejrzewamy, że może występować systematyczna zmienność między powtórzeniami np. - zmienność
ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH
 1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
Bioinformatyka wykład 3.I.2008
 Bioinformatyka wykład 3.I.2008 Białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2008-01-03 1 Plan wykładu analiza i porównywanie struktur białek. doświadczalne metody badania struktur
Bioinformatyka wykład 3.I.2008 Białkowa bioinformatyka strukturalna c.d. krzysztof_pawlowski@sggw.pl 2008-01-03 1 Plan wykładu analiza i porównywanie struktur białek. doświadczalne metody badania struktur
Zmienność ewolucyjna. Ewolucja molekularna
 Zmienność ewolucyjna Ewolucja molekularna Mechanizmy ewolucji Generujące zmienność mutacje rearanżacje genomu horyzontalny transfer genów! Działające na warianty wytworzone przez zmienność dobór naturalny
Zmienność ewolucyjna Ewolucja molekularna Mechanizmy ewolucji Generujące zmienność mutacje rearanżacje genomu horyzontalny transfer genów! Działające na warianty wytworzone przez zmienność dobór naturalny
STATYSTYKA I DOŚWIADCZALNICTWO Wykład 6
 STATYSTYKA I DOŚWIADCZALNICTWO Wykład 6 Metody sprawdzania założeń w analizie wariancji: -Sprawdzanie równości (jednorodności) wariancji testy: - Cochrana - Hartleya - Bartletta -Sprawdzanie zgodności
STATYSTYKA I DOŚWIADCZALNICTWO Wykład 6 Metody sprawdzania założeń w analizie wariancji: -Sprawdzanie równości (jednorodności) wariancji testy: - Cochrana - Hartleya - Bartletta -Sprawdzanie zgodności
Analizy filogenetyczne
 BIOINFORMATYKA edycja 2016 / 2017 wykład 6 Analizy filogenetyczne dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Cele i zastosowania 2. Podstawy ewolucyjne
BIOINFORMATYKA edycja 2016 / 2017 wykład 6 Analizy filogenetyczne dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Cele i zastosowania 2. Podstawy ewolucyjne
Mechanizmy zmienności ewolucyjnej. Podstawy ewolucji molekularnej.
 Mechanizmy zmienności ewolucyjnej Podstawy ewolucji molekularnej. Mechanizmy ewolucji } Generujące zmienność } mutacje } rearanżacje genomu } horyzontalny transfer genów } Działające na warianty wytworzone
Mechanizmy zmienności ewolucyjnej Podstawy ewolucji molekularnej. Mechanizmy ewolucji } Generujące zmienność } mutacje } rearanżacje genomu } horyzontalny transfer genów } Działające na warianty wytworzone
Wstęp do Informatyki zadania ze złożoności obliczeniowej z rozwiązaniami
 Wstęp do Informatyki zadania ze złożoności obliczeniowej z rozwiązaniami Przykład 1. Napisz program, który dla podanej liczby n wypisze jej rozkład na czynniki pierwsze. Oblicz asymptotyczną złożoność
Wstęp do Informatyki zadania ze złożoności obliczeniowej z rozwiązaniami Przykład 1. Napisz program, który dla podanej liczby n wypisze jej rozkład na czynniki pierwsze. Oblicz asymptotyczną złożoność
SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO
 SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO. Rzeczywistość (istniejąca lub projektowana).. Model fizyczny. 3. Model matematyczny (optymalizacyjny): a. Zmienne projektowania
SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO. Rzeczywistość (istniejąca lub projektowana).. Model fizyczny. 3. Model matematyczny (optymalizacyjny): a. Zmienne projektowania
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl Zasady zaliczenia przedmiotu Kolokwia (3 4 ) Ocena aktywności i przygotowania Obecność Literatura, materiały Bioinformatyka i ewolucja
Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl Zasady zaliczenia przedmiotu Kolokwia (3 4 ) Ocena aktywności i przygotowania Obecność Literatura, materiały Bioinformatyka i ewolucja
Ćwiczenie 5/6. Informacja genetyczna i geny u różnych grup organizmów. Porównywanie sekwencji nukleotydowych w bazie NCBI z wykorzystaniem BLAST.
 Ćwiczenie 5/6 Informacja genetyczna i geny u różnych grup organizmów. Porównywanie sekwencji nukleotydowych w bazie NCBI z wykorzystaniem BLAST. Prof. dr hab. Roman Zieliński 1. Informacja genetyczna u
Ćwiczenie 5/6 Informacja genetyczna i geny u różnych grup organizmów. Porównywanie sekwencji nukleotydowych w bazie NCBI z wykorzystaniem BLAST. Prof. dr hab. Roman Zieliński 1. Informacja genetyczna u
Korzystanie z podstawowych rozkładów prawdopodobieństwa (tablice i arkusze kalkulacyjne)
 Korzystanie z podstawowych rozkładów prawdopodobieństwa (tablice i arkusze kalkulacyjne) Przygotował: Dr inż. Wojciech Artichowicz Katedra Hydrotechniki PG Zima 2014/15 1 TABLICE ROZKŁADÓW... 3 ROZKŁAD
Korzystanie z podstawowych rozkładów prawdopodobieństwa (tablice i arkusze kalkulacyjne) Przygotował: Dr inż. Wojciech Artichowicz Katedra Hydrotechniki PG Zima 2014/15 1 TABLICE ROZKŁADÓW... 3 ROZKŁAD
Zespół kanoniczny N,V, T. acc o n =min {1, exp [ U n U o ] }
![Zespół kanoniczny N,V, T. acc o n =min {1, exp [ U n U o ] } Zespół kanoniczny N,V, T. acc o n =min {1, exp [ U n U o ] }](/thumbs/64/51033976.jpg) Zespół kanoniczny Zespół kanoniczny N,V, T acc o n =min {1, exp [ U n U o ] } Zespół izobaryczno-izotermiczny Zespół izobaryczno-izotermiczny N P T acc o n =min {1, exp [ U n U o ] } acc o n =min {1, exp[
Zespół kanoniczny Zespół kanoniczny N,V, T acc o n =min {1, exp [ U n U o ] } Zespół izobaryczno-izotermiczny Zespół izobaryczno-izotermiczny N P T acc o n =min {1, exp [ U n U o ] } acc o n =min {1, exp[
Analiza korespondencji
 Analiza korespondencji Kiedy stosujemy? 2 W wielu badaniach mamy do czynienia ze zmiennymi jakościowymi (nominalne i porządkowe) typu np.: płeć, wykształcenie, status palenia. Punktem wyjścia do analizy
Analiza korespondencji Kiedy stosujemy? 2 W wielu badaniach mamy do czynienia ze zmiennymi jakościowymi (nominalne i porządkowe) typu np.: płeć, wykształcenie, status palenia. Punktem wyjścia do analizy
REGRESJA I KORELACJA MODEL REGRESJI LINIOWEJ MODEL REGRESJI WIELORAKIEJ. Analiza regresji i korelacji
 Statystyka i opracowanie danych Ćwiczenia 5 Izabela Olejarczyk - Wożeńska AGH, WIMiIP, KISIM REGRESJA I KORELACJA MODEL REGRESJI LINIOWEJ MODEL REGRESJI WIELORAKIEJ MODEL REGRESJI LINIOWEJ Analiza regresji
Statystyka i opracowanie danych Ćwiczenia 5 Izabela Olejarczyk - Wożeńska AGH, WIMiIP, KISIM REGRESJA I KORELACJA MODEL REGRESJI LINIOWEJ MODEL REGRESJI WIELORAKIEJ MODEL REGRESJI LINIOWEJ Analiza regresji
RMSD - Ocena jakości wybranych molekularnych struktur przestrzennych
 RMSD - Ocena jakości wybranych molekularnych struktur przestrzennych Joanna Wiśniewska Promotor: dr inż. P. Łukasiak Spis treści 1. Zakres pracy magisterskiej 2. Struktura białka 3. Struktura kwasów nukleionowych
RMSD - Ocena jakości wybranych molekularnych struktur przestrzennych Joanna Wiśniewska Promotor: dr inż. P. Łukasiak Spis treści 1. Zakres pracy magisterskiej 2. Struktura białka 3. Struktura kwasów nukleionowych
WYKŁAD 8 ANALIZA REGRESJI
 WYKŁAD 8 ANALIZA REGRESJI Regresja 1. Metoda najmniejszych kwadratów-regresja prostoliniowa 2. Regresja krzywoliniowa 3. Estymacja liniowej funkcji regresji 4. Testy istotności współczynnika regresji liniowej
WYKŁAD 8 ANALIZA REGRESJI Regresja 1. Metoda najmniejszych kwadratów-regresja prostoliniowa 2. Regresja krzywoliniowa 3. Estymacja liniowej funkcji regresji 4. Testy istotności współczynnika regresji liniowej
( x) Równanie regresji liniowej ma postać. By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : Gdzie:
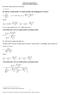 ma postać y = ax + b Równanie regresji liniowej By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : xy b = a = b lub x Gdzie: xy = też a = x = ( b ) i to dane empiryczne, a ilość
ma postać y = ax + b Równanie regresji liniowej By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : xy b = a = b lub x Gdzie: xy = też a = x = ( b ) i to dane empiryczne, a ilość
weryfikacja hipotez dotyczących parametrów populacji (średnia, wariancja)
 PODSTAWY STATYSTYKI. Teoria prawdopodobieństwa i elementy kombinatoryki. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5. Testy parametryczne (na
PODSTAWY STATYSTYKI. Teoria prawdopodobieństwa i elementy kombinatoryki. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5. Testy parametryczne (na
Urszula Poziomek, doradca metodyczny w zakresie biologii Materiał dydaktyczny przygotowany na konferencję z cyklu Na miarę Nobla, 14 stycznia 2010 r.
 Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
parametrów strukturalnych modelu = Y zmienna objaśniana, X 1,X 2,,X k zmienne objaśniające, k zmiennych objaśniających,
 诲 瞴瞶 瞶 ƭ0 ƭ 瞰 parametrów strukturalnych modelu Y zmienna objaśniana, = + + + + + X 1,X 2,,X k zmienne objaśniające, k zmiennych objaśniających, α 0, α 1, α 2,,α k parametry strukturalne modelu, k+1 parametrów
诲 瞴瞶 瞶 ƭ0 ƭ 瞰 parametrów strukturalnych modelu Y zmienna objaśniana, = + + + + + X 1,X 2,,X k zmienne objaśniające, k zmiennych objaśniających, α 0, α 1, α 2,,α k parametry strukturalne modelu, k+1 parametrów
K wartość kapitału zaangażowanego w proces produkcji, w tys. jp.
 Sprawdzian 2. Zadanie 1. Za pomocą KMNK oszacowano następującą funkcję produkcji: Gdzie: P wartość produkcji, w tys. jp (jednostek pieniężnych) K wartość kapitału zaangażowanego w proces produkcji, w tys.
Sprawdzian 2. Zadanie 1. Za pomocą KMNK oszacowano następującą funkcję produkcji: Gdzie: P wartość produkcji, w tys. jp (jednostek pieniężnych) K wartość kapitału zaangażowanego w proces produkcji, w tys.
Ważne rozkłady i twierdzenia c.d.
 Ważne rozkłady i twierdzenia c.d. Funkcja charakterystyczna rozkładu Wielowymiarowy rozkład normalny Elipsa kowariacji Sploty rozkładów Rozkłady jednostajne Sploty z rozkładem normalnym Pobieranie próby
Ważne rozkłady i twierdzenia c.d. Funkcja charakterystyczna rozkładu Wielowymiarowy rozkład normalny Elipsa kowariacji Sploty rozkładów Rozkłady jednostajne Sploty z rozkładem normalnym Pobieranie próby
STATYSTYKA MATEMATYCZNA
 STATYSTYKA MATEMATYCZNA 1. Wykład wstępny. Teoria prawdopodobieństwa i elementy kombinatoryki 2. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5.
STATYSTYKA MATEMATYCZNA 1. Wykład wstępny. Teoria prawdopodobieństwa i elementy kombinatoryki 2. Zmienne losowe i ich rozkłady 3. Populacje i próby danych, estymacja parametrów 4. Testowanie hipotez 5.
0 + 0 = 0, = 1, = 1, = 0.
 5 Kody liniowe Jak już wiemy, w celu przesłania zakodowanego tekstu dzielimy go na bloki i do każdego z bloków dodajemy tak zwane bity sprawdzające. Bity te są w ścisłej zależności z bitami informacyjnymi,
5 Kody liniowe Jak już wiemy, w celu przesłania zakodowanego tekstu dzielimy go na bloki i do każdego z bloków dodajemy tak zwane bity sprawdzające. Bity te są w ścisłej zależności z bitami informacyjnymi,
Kodowanie i kompresja Streszczenie Studia Licencjackie Wykład 11,
 1 Kwantyzacja skalarna Kodowanie i kompresja Streszczenie Studia Licencjackie Wykład 11, 10.05.005 Kwantyzacja polega na reprezentowaniu dużego zbioru wartości (być może nieskończonego) za pomocą wartości
1 Kwantyzacja skalarna Kodowanie i kompresja Streszczenie Studia Licencjackie Wykład 11, 10.05.005 Kwantyzacja polega na reprezentowaniu dużego zbioru wartości (być może nieskończonego) za pomocą wartości
