Różnorodność osobników gatunku
|
|
|
- Kazimiera Wawrzyniak
- 7 lat temu
- Przeglądów:
Transkrypt
1 ALEKSANDRA ŚWIERCZ
2 Różnorodność osobników gatunku Single Nucleotide Polymorphism (SNP) Różnica na jednej pozycji, małe delecje, insercje (INDELs) SNP pojawia się ~1/1000 pozycji Można je znaleźć porównując odczyty z jednego osobnika do genomu referencyjnego Structural variations to duże różnice w genomach. Mogą to być duże: Delecje brak fragmentu genomu Insercje wstawienie fragmentu genomu Inwersje odwrócenie fragmentu genomu Translokacje zmiana położenia fragmentu genomu (może być również na innym chromosomie) Duplikacje powtórzenia fragmentów genomu A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2
3 SNP Niedopasowania SNP Screen z mapowania IGV Błąd sekwencjonowania SNP homozygota SNP heterozygota A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 3
4 Który z osobników jest rodzicem, a który dzieckiem? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 4
5 R.Nielsen, JS.Paul, A.Albrechtsen, YS.Song Genotype and SNP calling from next-generation sequencing data Nature Reviews Genetics 12, (2011) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 5
6 dbsnp oraz HapMap dbsnp obecnie 139 wersja bazy. Pojawiły się nowe organizmy wraz z listą różnic (dotychczas zbadaną) między osobnikami HapMap międzynarodowy projekt, który ma na celu wykrycie i skatalogowanie podobieństw i różnic pomiędzy organizmami ludzkimi. Ośrodki biorące udział w projekcie pochodzą z Japonii, Wielkiej Brytanii, Kanady, Chin, Nigerii oraz Stanów Zjednoczonych. Projekt HapMap jest ogólnodostępny, i ma na celu pomoc środowisku biomedycznemu w znalezieniu genów powodujących choroby i odpowiedzi na leki terapeutyczne. W bazie HapMap analizowano DNA z 270 osobników populacji Afrykańskiej, Azjatyckiej i Europejskiej. Badano zarówno osobników pojedynczych, jak i trio, czyli rodziców wraz z ich dorosłym potomkiem A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 6
7 Różnice strukturalne SV Monya Baker Structural variation: the genome's hidden architecture Nature Methods 9, (2012) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 7
8 Sposoby na wykrywanie SV RD Read Depth badanie głębokości pokrycia RP Read Pairs sprawdzenie mapowania odczytów sparowanych, czy mapują się z taką samą odległością (wg. rozkładu), czy mapują się w odpowiednią stronę, czy mapują się oba odczyty z pary SR Split Reads szukanie odczytów, które nie mapują się w całości do genomu referencyjnego, lecz jego fragmenty mapują się w odległych miejscach odczyty te świadczą o nietypowym (innym niż w gen. ref.) połączeniu między fragmentami genomu AS AsseMbly de novo asemblacja de novo (bez mapowania) odczytów, następnie porównanie, czy zasemblowane kontigi pokrywają się z genomem referencyjnym A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 8
9 Jakie SV można odkryć dzięki różnym podejściom? Monya Baker Structural variation: the genome's hidden architecture Nature Methods 9, (2012) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 9
10 R.E. Mills et al., Mapping copy number variation by population-scale genome sequencing, Nature 470, 59 65, 2011 A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 10
11 Read Depth A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 11
12 Kropka zielona to średnie pokrycie odczytami dla okna 1kbp zdrowej tkanki pacjenta Kropka czerwona to średnie pokrycie odczytami dla okna 1kbp chorej tkanki pacjenta Takie same falowanie kropek zielonych i czerwonych oznacza, że nie ma różnic między zdrową i chorą tkanką (tego samego) pacjenta, tylko są różnice między genomem pacjenta, a genomem referencyjnym Średnie pokrycie dla zdrowej tkanki jest ok. 52, natomiast dla chorej tkanki ok. 40 Zmiana w zachowaniu między kropkami zielonymi i czerwonymi oznacza zmianę liczby kopii danego fragmentu Dzięki analizie zmiany głębokości pokrycia można znaleźć jedynie różnice w liczbie kopii poszczególnych fragmentów, nie wiemy jednak nic na temat położenia fragmentów w genomie. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 12
13 Głębokość pokrycia wykrywanie duplikacji Badany genom Genom referencyjny A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 13
14 Wykrywanie SV za pomocą odczytów sparowanych Długość fragmentu Badany genom Genom referencyjny Odległość mapowania Brak różnic w strukturze genomu badanego i referencyjnego, gdyż: Długość fragmentu jest taka sama jak odległość mapowania na genomie referencyjnym Odczyty są zmapowane na genomie referencyjnym zgodnie z oryginalnym fragmentem A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 14
15 Insercja insert Badany genom Długość fragmentu Genom referencyjny Odległość mapowania Insercja w genomie badanym, gdyż: Odległość mapowania w genomie referencyjnym jest mniejsza niż długość fragmentu długość insertu = długość fragmentu - odległość mapowania (± rozrzut długości) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 15
16 Insercja przypadek czy na pewno? insert Długość fragmentu Badany genom Genom referencyjny Odległość mapowania Spójność przy mapowaniu innych odczytów w tym miejscu A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 16
17 Insercja przypadek czy na pewno? insert Długość fragmentów Badany genom Genom referencyjny Odległość mapowania Długość fragmentów nie jest równa, może się różnić ± 10% (zależy od przygotowania biblioteki) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 17
18 Insercja przypadek czy na pewno? insert? Długość fragmentu Badany genom Genom referencyjny Odległość mapowania Zbyt długi fragment wziął udział w sekwencjonowaniu, a reszta odczytów nie potwierdza insercji. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 18
19 Inwersja Badany genom Długość fragmentu Genom referencyjny Odległość mapowania = m m długość fragmentu < długość inwersji Fragment genomu uległ inwersji, gdyż: Odczyty zmapowane są na genomie referencyjnym odwrotnie (discordant) Długość fragmentu oraz odległość mapowania są różne (to nie jest konieczne!) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 19
20 Inwersja jaka jest długość fragmentu? Długość fragmentu Badany genom Odległość mapowania = m Genom referencyjny m długość fragmentu < długość inwersji < m+ długość fragmentu A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 20
21 Inwersja spójność mapowania x a Badany genom x' a x b x' b Genom referencyjny Odległość mapowania A Odległość mapowania B Odległość mapowania A = odległość mapowania B A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 21
22 Inwersja Badany genom Genom referencyjny A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 22
23 Delecja Badany genom Długość fragmentu Genom referencyjny Odległość mapowania Fragment genomu uległ delecji w genomie badanym, gdyż: Długość fragmentu jest krótsza niż odległość mapowania na genomie referencyjnym A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 23
24 Translokacja, duplikacja? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 24
25 Split reads Odczyty, które nie mapują się w całości do genomu referencyjnego, lecz jego fragmenty zmapowane w odległych miejscach świadczą o rearanżacjach chromosomowych Sekwencjonowanie => odczyty sparowane Mapowanie do genomu referencyjnego Odczyty zmapowane do genomu Odczyty niezmapowane mogą leżeć na łączeniu wariantów strukturalnych A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 25
26 Split reads Ht-seq_2012_module3.pdf Canadian Bioinformatics Workshop A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 26
27 A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 27
28 A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 28
29 Jak duże są różnice SV? Ile SV jest pomiędzy dwoma osobnikami? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 29
30 A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 30
31 A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 31
32 Różnice między ludźmi Ludzie różnią się: kilkoma tysiącami delecji Kilkuset duplikacjami Kilkuset inwersjami Kilkuset insercjami transpozonów Kilkuset przesunięciami genów W wynikach różnych metod do wykrywania SV mamy: Wiele błędów pozytywnych Wiele błędów negatywnych Trudności w wykryciu SV, które nachodzą na sekwencje repetytywne A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 32
33 False positives Walidacja innymi metodami, w celu likwidacji błędów false positives A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 33
34 Wpływ wariantów strukturalnych na dawkę genów Feuk, L. et al. Structural variation in the human genome. Nature Review Genetics 7, 92 (2006) Did you know that a large number of your genes exist in variable numbers of copies? While they can overlap with disease-related genes, these variants exist in healthy individuals too. Większość zmian liczby kopii można znaleźć w zdrowych osobnikach. Podejrzewa się że te zmiany powodują choroby poprzez szereg mechanizmów pokazanych na rysunku. Po pierwsze różna liczba kopii może spowodować różną dawkę genu poprzez delecje lub insercje, które może spowodować że odmienny gen ulegnie ekspresji potencjalnie powodując chorobę. Dawka genu opisuje liczbę kopii genu w komórce, co się przekłada na zwiększoną lub zmniejszoną ekspresję tego genu. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 34
35 Delecje mogą spowodować zmniejszoną dawkę genu, poprzez usunięcie jednego allelu lub poprzez delecję allelu (dominującego) ujawni się recesywna wersja genu. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 35
36 Jeśli warianty strukturalne nakładają się na geny, to może zostać zredukowana lub w ogóle zablokowana ekspresja genu poprzez inwersję, translokację czy delecję. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 36
37 Warianty SV mogą także mieć wpływ na elementy regulatorowe, jeśli zostanie on usunięty może zostać zwiększona lub zmniejszona ekspresja genu. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 37
38 Czasami kombinacja dwóch lub większej liczby wariantów może spowodować złożoną chorobę, podczas gdy pojedyncze zmiany nie powodują żadnego efektu. Dodatkowo złożone choroby mogą się pojawić jeśli różna liczba kopii jest połączona z innymi genetycznymi lub środowiskowymi czynnikami. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 38
39 Podsumowanie Różne podejścia do sekwencjonowania wysokoprzepustowego: Wady i zalety metod Długość odczytów Jakość sekwencji na końcówkach sewkencji Odczyty sparowane, pojedyncze Specyficzne rodzaje błędów Mapowanie do genomu referencyjnego: Algorytmy dopasowania lokalnego, globalnego i semiglobalnego Macierze kropkowe (dotmatrix) Tworzenie indeksu BWT Haszowanie A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 39
40 Podsumowanie 2 Asemblacja de novo Trudności w asemblacji Powtórzenia zaburzają obliczenia Overlap layout consensus Grafy de Bruijna(błędne ścieżki w grafach) Wady i zalety obu podejść RNA-sequencing: Algorytmy mapowania sekwencji RNA (różne podejścia: asemblacja de novo, mapowanie do transkryptomu, mapowanie do genomu) Przeszkody w mapowaniu RNA do genomu Trudność w rozpoznawaniu nowych transkryptów Różnicowa ekspresja genów i alternatywny splicing(warianty splicingowe) A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 40
Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2
 ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
ALEKSANDRA ŚWIERCZ Co to jest transkryptom? A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH
Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing. Wykład 6 Część 1 NGS - wstęp Dr Wioleta Drobik-Czwarno
 Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing Wykład 6 Część 1 NGS - wstęp Dr Wioleta Drobik-Czwarno Macierze tkankowe TMA ang. Tissue microarray Technika opisana w 1987 roku (Wan i
Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing Wykład 6 Część 1 NGS - wstęp Dr Wioleta Drobik-Czwarno Macierze tkankowe TMA ang. Tissue microarray Technika opisana w 1987 roku (Wan i
Sekwencjonowanie, przewidywanie genów
 Instytut Informatyki i Matematyki Komputerowej UJ, opracowanie: mgr Ewa Matczyńska, dr Jacek Śmietański Sekwencjonowanie, przewidywanie genów 1. Technologie sekwencjonowania Genomem nazywamy sekwencję
Instytut Informatyki i Matematyki Komputerowej UJ, opracowanie: mgr Ewa Matczyńska, dr Jacek Śmietański Sekwencjonowanie, przewidywanie genów 1. Technologie sekwencjonowania Genomem nazywamy sekwencję
PODSTAWY BIOINFORMATYKI 11 BAZA DANYCH HAPMAP
 PODSTAWY BIOINFORMATYKI 11 BAZA DANYCH HAPMAP WSTĘP 1. SNP 2. haplotyp 3. równowaga sprzężeń 4. zawartość bazy HapMap 5. przykłady zastosowań Copyright 2013, Joanna Szyda HAPMAP BAZA DANYCH HAPMAP - haplotypy
PODSTAWY BIOINFORMATYKI 11 BAZA DANYCH HAPMAP WSTĘP 1. SNP 2. haplotyp 3. równowaga sprzężeń 4. zawartość bazy HapMap 5. przykłady zastosowań Copyright 2013, Joanna Szyda HAPMAP BAZA DANYCH HAPMAP - haplotypy
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI
 ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI JOANNA SZYDA MAGDALENA FRĄSZCZAK MAGDA MIELCZAREK WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI JOANNA SZYDA MAGDALENA FRĄSZCZAK MAGDA MIELCZAREK WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI
 ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI Joanna Szyda Magdalena Frąszczak Magda Mielczarek WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI Joanna Szyda Magdalena Frąszczak Magda Mielczarek WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
"Zapisane w genach, czyli Python a tajemnice naszego genomu."
 "Zapisane w genach, czyli Python a tajemnice naszego genomu." Dr Kaja Milanowska Instytut Biologii Molekularnej i Biotechnologii UAM VitaInSilica sp. z o.o. Warszawa, 9 lutego 2015 Dane biomedyczne 1)
"Zapisane w genach, czyli Python a tajemnice naszego genomu." Dr Kaja Milanowska Instytut Biologii Molekularnej i Biotechnologii UAM VitaInSilica sp. z o.o. Warszawa, 9 lutego 2015 Dane biomedyczne 1)
Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing
 Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing Wykład 7 Etapy analizy NGS Dr Wioleta Drobik-Czwarno Etapy analizy NGS Kontrola jakości surowych danych (format fastq) Jakość odczytów,
Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing Wykład 7 Etapy analizy NGS Dr Wioleta Drobik-Czwarno Etapy analizy NGS Kontrola jakości surowych danych (format fastq) Jakość odczytów,
Reswkwencjonowanie vs asemblacja de novo
 ALEKSANDRA ŚWIERCZ Reswkwencjonowanie vs asemblacja de novo Resekwencjonowanie to odtworzenie badanej sekwencji poprzez mapowanie odczytów do genomu/transkryptomu referencyjnego (tego samego gatunku lub
ALEKSANDRA ŚWIERCZ Reswkwencjonowanie vs asemblacja de novo Resekwencjonowanie to odtworzenie badanej sekwencji poprzez mapowanie odczytów do genomu/transkryptomu referencyjnego (tego samego gatunku lub
Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing
 Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing Wykład 7 Etapy analizy NGS Dr Wioleta Drobik-Czwarno Etapy analizy NGS Kontrola jakości surowych danych (format fastq) Jakość odczytów,
Sekwencjonowanie Nowej Generacji ang. Next Generation Sequencing Wykład 7 Etapy analizy NGS Dr Wioleta Drobik-Czwarno Etapy analizy NGS Kontrola jakości surowych danych (format fastq) Jakość odczytów,
PODSTAWY BIOINFORMATYKI WYKŁAD 4 ANALIZA DANYCH NGS
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 ANALIZA DANYCH NGS SEKWENCJONOWANIE GENOMÓW NEXT GENERATION METODA NOWEJ GENERACJI Sekwencjonowanie bardzo krótkich fragmentów 50-700 bp DNA unieruchomione na płytce Szybkie
PODSTAWY BIOINFORMATYKI WYKŁAD 4 ANALIZA DANYCH NGS SEKWENCJONOWANIE GENOMÓW NEXT GENERATION METODA NOWEJ GENERACJI Sekwencjonowanie bardzo krótkich fragmentów 50-700 bp DNA unieruchomione na płytce Szybkie
Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu
 Ćwiczenie 4 Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu Wstęp CYP2D6 kodowany przez gen występujący w co najmniej w 78 allelicznych formach związanych ze zmniejszoną
Ćwiczenie 4 Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu Wstęp CYP2D6 kodowany przez gen występujący w co najmniej w 78 allelicznych formach związanych ze zmniejszoną
Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu
 Ćwiczenie 4 Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu Wstęp CYP2D6 kodowany przez gen występujący w co najmniej w 78 allelicznych formach związanych ze zmniejszoną
Ćwiczenie 4 Oznaczenie polimorfizmu genetycznego cytochromu CYP2D6: wykrywanie liczby kopii genu Wstęp CYP2D6 kodowany przez gen występujący w co najmniej w 78 allelicznych formach związanych ze zmniejszoną
Przetarg nieograniczony na zakup specjalistycznej aparatury laboratoryjnej Znak sprawy: DZ-2501/6/17
 Część nr 2: SEKWENATOR NASTĘPNEJ GENERACJI Z ZESTAWEM DEDYKOWANYCH ODCZYNNIKÓW Określenie przedmiotu zamówienia zgodnie ze Wspólnym Słownikiem Zamówień (CPV): 38500000-0 aparatura kontrolna i badawcza
Część nr 2: SEKWENATOR NASTĘPNEJ GENERACJI Z ZESTAWEM DEDYKOWANYCH ODCZYNNIKÓW Określenie przedmiotu zamówienia zgodnie ze Wspólnym Słownikiem Zamówień (CPV): 38500000-0 aparatura kontrolna i badawcza
Spis treści. Przedmowa... XI. Wprowadzenie i biologiczne bazy danych. 1 Wprowadzenie... 3. 2 Wprowadzenie do biologicznych baz danych...
 Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
BUDOWA I FUNKCJA GENOMU LUDZKIEGO
 BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
Niepełnosprawność intelektualna
 Niepełnosprawność intelektualna stan badań a możliwości diagnostyki molekularnej Agnieszka Charzewska Zakład Genetyki Medycznej Instytut Matki i Dziecka Niepełnosprawność intelektualna (NI, ID) zaburzenie
Niepełnosprawność intelektualna stan badań a możliwości diagnostyki molekularnej Agnieszka Charzewska Zakład Genetyki Medycznej Instytut Matki i Dziecka Niepełnosprawność intelektualna (NI, ID) zaburzenie
Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej
 Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
ZARZĄDZANIE POPULACJAMI ZWIERZĄT
 ZARZĄDZANIE POPULACJAMI ZWIERZĄT Ćwiczenia 1 mgr Magda Kaczmarek-Okrój magda_kaczmarek_okroj@sggw.pl 1 ZAGADNIENIA struktura genetyczna populacji obliczanie frekwencji genotypów obliczanie frekwencji alleli
ZARZĄDZANIE POPULACJAMI ZWIERZĄT Ćwiczenia 1 mgr Magda Kaczmarek-Okrój magda_kaczmarek_okroj@sggw.pl 1 ZAGADNIENIA struktura genetyczna populacji obliczanie frekwencji genotypów obliczanie frekwencji alleli
Przydatność technologii Sekwencjonowania Nowej Generacji (NGS) w kolekcjach Banków Genów Joanna Noceń Kinga Smolińska Marta Puchta Kierownik tematu:
 Przydatność technologii Sekwencjonowania Nowej Generacji (NGS) w kolekcjach Banków Genów Joanna Noceń Kinga Smolińska Marta Puchta Kierownik tematu: prof. dr hab. Jerzy H. Czembor SEKWENCJONOWANIE I generacji
Przydatność technologii Sekwencjonowania Nowej Generacji (NGS) w kolekcjach Banków Genów Joanna Noceń Kinga Smolińska Marta Puchta Kierownik tematu: prof. dr hab. Jerzy H. Czembor SEKWENCJONOWANIE I generacji
Ekspresja genów. A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2
 ALEKSANDRA ŚWIERCZ Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Różnice między eksperymentem mikromacierzowym a RNA-seq Przy użyciu mikromacierzy
ALEKSANDRA ŚWIERCZ Ekspresja genów http://genome.wellcome.ac.uk/doc_wtd020757.html A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Różnice między eksperymentem mikromacierzowym a RNA-seq Przy użyciu mikromacierzy
Jaki koń jest nie każdy widzi - genomika populacji polskich ras koni
 Jaki koń jest nie każdy widzi - genomika populacji polskich ras koni Gurgul A., Jasielczuk I., Semik-Gurgul E., Pawlina-Tyszko K., Szmatoła T., Bugno-Poniewierska M. Instytut Zootechniki PIB Zakład Biologii
Jaki koń jest nie każdy widzi - genomika populacji polskich ras koni Gurgul A., Jasielczuk I., Semik-Gurgul E., Pawlina-Tyszko K., Szmatoła T., Bugno-Poniewierska M. Instytut Zootechniki PIB Zakład Biologii
DHPLC. Denaturing high performance liquid chromatography. Wiktoria Stańczyk Zofia Kołeczko
 DHPLC Denaturing high performance liquid chromatography Wiktoria Stańczyk Zofia Kołeczko Mini-słowniczek SNP (Single Nucleotide Polymorphism) - zmienność sekwencji DNA; HET - analiza heterodupleksów; HPLC
DHPLC Denaturing high performance liquid chromatography Wiktoria Stańczyk Zofia Kołeczko Mini-słowniczek SNP (Single Nucleotide Polymorphism) - zmienność sekwencji DNA; HET - analiza heterodupleksów; HPLC
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii
 Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Na czym skończyliśmy BLACK BOX. Sekwencjonowanie polega na odczytaniu sekwencji liter DNA/RNA badanego fragmentu genomu
 ALEKSANDRA ŚWIERCZ Na czym skończyliśmy BLACK BOX AAATGCCTGCCCTGAAGGCCTGCGTA GTTTTGGGAGAAGACCCACGGATA AAGGTGTAGCCCCGTAGC GGGGGGTATTATTTATTTTATACCCAC.. ACAGGAUCGUUGGAUGGTGGGA. Sekwencjonowanie polega na
ALEKSANDRA ŚWIERCZ Na czym skończyliśmy BLACK BOX AAATGCCTGCCCTGAAGGCCTGCGTA GTTTTGGGAGAAGACCCACGGATA AAGGTGTAGCCCCGTAGC GGGGGGTATTATTTATTTTATACCCAC.. ACAGGAUCGUUGGAUGGTGGGA. Sekwencjonowanie polega na
Ekologia molekularna. wykład 10
 Ekologia molekularna wykład 10 Zasięg gatunku wykład 10/2 Środowisko Człowiek rozumny posiada bardzo szeroki zasięg występowania, nie dorównuje mu w tym względzie żaden inny ssak. Zamieszkuje on wszystkie
Ekologia molekularna wykład 10 Zasięg gatunku wykład 10/2 Środowisko Człowiek rozumny posiada bardzo szeroki zasięg występowania, nie dorównuje mu w tym względzie żaden inny ssak. Zamieszkuje on wszystkie
Składniki jądrowego genomu człowieka
 Składniki jądrowego genomu człowieka Genom człowieka 3 000 Mpz (3x10 9, 100 cm) Geny i sekwencje związane z genami (900 Mpz, 30% g. jądrowego) DNA pozagenowy (2100 Mpz, 70%) DNA kodujący (90 Mpz ~ ok.
Składniki jądrowego genomu człowieka Genom człowieka 3 000 Mpz (3x10 9, 100 cm) Geny i sekwencje związane z genami (900 Mpz, 30% g. jądrowego) DNA pozagenowy (2100 Mpz, 70%) DNA kodujący (90 Mpz ~ ok.
Analizy wielkoskalowe w badaniach chromatyny
 Analizy wielkoskalowe w badaniach chromatyny Analizy wielkoskalowe wykorzystujące mikromacierze DNA Genotypowanie: zróżnicowane wewnątrz genów RNA Komórka eukariotyczna Ekspresja genów: Które geny? Poziom
Analizy wielkoskalowe w badaniach chromatyny Analizy wielkoskalowe wykorzystujące mikromacierze DNA Genotypowanie: zróżnicowane wewnątrz genów RNA Komórka eukariotyczna Ekspresja genów: Które geny? Poziom
PODSTAWY BIOINFORMATYKI 12 MIKROMACIERZE
 PODSTAWY BIOINFORMATYKI 12 MIKROMACIERZE WSTĘP 1. Mikromacierze ekspresyjne tworzenie macierzy przykłady zastosowań 2. Mikromacierze SNP tworzenie macierzy przykłady zastosowań MIKROMACIERZE EKSPRESYJNE
PODSTAWY BIOINFORMATYKI 12 MIKROMACIERZE WSTĘP 1. Mikromacierze ekspresyjne tworzenie macierzy przykłady zastosowań 2. Mikromacierze SNP tworzenie macierzy przykłady zastosowań MIKROMACIERZE EKSPRESYJNE
GENOMIKA. MAPOWANIE GENOMÓW MAPY GENOMICZNE
 GENOMIKA. MAPOWANIE GENOMÓW MAPY GENOMICZNE Bioinformatyka, wykład 3 (21.X.2008) krzysztof_pawlowski@sggw.waw.pl tydzień temu Gen??? Biologiczne bazy danych historia Biologiczne bazy danych najważniejsze
GENOMIKA. MAPOWANIE GENOMÓW MAPY GENOMICZNE Bioinformatyka, wykład 3 (21.X.2008) krzysztof_pawlowski@sggw.waw.pl tydzień temu Gen??? Biologiczne bazy danych historia Biologiczne bazy danych najważniejsze
Politechnika Wrocławska. Dopasowywanie sekwencji Sequence alignment
 Dopasowywanie sekwencji Sequence alignment Drzewo filogenetyczne Kserokopiarka zadanie: skopiować 300 stron. Co może pójść źle? 2x ta sama strona Opuszczona strona Nadmiarowa pusta strona Strona do góry
Dopasowywanie sekwencji Sequence alignment Drzewo filogenetyczne Kserokopiarka zadanie: skopiować 300 stron. Co może pójść źle? 2x ta sama strona Opuszczona strona Nadmiarowa pusta strona Strona do góry
CHARAKTERYSTYKA PRZEDMIOTU Pracownia Informatyczna 1 PRACOWNIA INFORMATYCZNA 2018/2019 MAGDA MIELCZAREK 1
 CHARAKTERYSTYKA PRZEDMIOTU Pracownia Informatyczna 1 PRACOWNIA INFORMATYCZNA 2018/2019 MAGDA MIELCZAREK 1 PRACOWNIA INFORMATYCZNA PROWADZĄCY: Dr Magda Mielczarek (biolog) Katedra Genetyki, pokój nr 21
CHARAKTERYSTYKA PRZEDMIOTU Pracownia Informatyczna 1 PRACOWNIA INFORMATYCZNA 2018/2019 MAGDA MIELCZAREK 1 PRACOWNIA INFORMATYCZNA PROWADZĄCY: Dr Magda Mielczarek (biolog) Katedra Genetyki, pokój nr 21
Podłoże molekularne NF1 i RASopatii. Możliwości diagnostyczne.
 Podłoże molekularne NF1 i RASopatii. Możliwości diagnostyczne. MONIKA G O S Z AKŁAD G ENETYKI MEDYCZ NEJ, I N STYTUT MATKI I DZIECKA SYMPOZJUM A LBA - JULIA WARSZAWA, 2-3.12.2017 Czym jest gen? Definicja:
Podłoże molekularne NF1 i RASopatii. Możliwości diagnostyczne. MONIKA G O S Z AKŁAD G ENETYKI MEDYCZ NEJ, I N STYTUT MATKI I DZIECKA SYMPOZJUM A LBA - JULIA WARSZAWA, 2-3.12.2017 Czym jest gen? Definicja:
Wstęp do Biologii Obliczeniowej
 Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Metody badania polimorfizmu/mutacji DNA. Aleksandra Sałagacka Pracownia Diagnostyki Molekularnej i Farmakogenomiki Uniwersytet Medyczny w Łodzi
 Metody badania polimorfizmu/mutacji DNA Aleksandra Sałagacka Pracownia Diagnostyki Molekularnej i Farmakogenomiki Uniwersytet Medyczny w Łodzi Mutacja Mutacja (łac. mutatio zmiana) - zmiana materialnego
Metody badania polimorfizmu/mutacji DNA Aleksandra Sałagacka Pracownia Diagnostyki Molekularnej i Farmakogenomiki Uniwersytet Medyczny w Łodzi Mutacja Mutacja (łac. mutatio zmiana) - zmiana materialnego
Konkurs szkolny Mistrz genetyki etap II
 onkurs szkolny istrz genetyki etap II 1.W D pewnego pierwotniaka tymina stanowi 28 % wszystkich zasad azotowych. blicz i zapisz, jaka jest zawartość procentowa każdej z pozostałych zasad w D tego pierwotniaka.
onkurs szkolny istrz genetyki etap II 1.W D pewnego pierwotniaka tymina stanowi 28 % wszystkich zasad azotowych. blicz i zapisz, jaka jest zawartość procentowa każdej z pozostałych zasad w D tego pierwotniaka.
PODSTAWY BIOINFORMATYKI 3 SEKWENCJONOWANIE GENOMÓW I
 PODSTAWY BIOINFORMATYKI 3 SEKWENCJONOWANIE GENOMÓW I PROJEKTY POZNANIA INNYCH GENOMÓW 1 400 2009 2010 1 200 2011 2012 1 000 800 600 400 200 986 1153 1285 914 954 717 759 757 251 254 889 0 23 ukończone
PODSTAWY BIOINFORMATYKI 3 SEKWENCJONOWANIE GENOMÓW I PROJEKTY POZNANIA INNYCH GENOMÓW 1 400 2009 2010 1 200 2011 2012 1 000 800 600 400 200 986 1153 1285 914 954 717 759 757 251 254 889 0 23 ukończone
Imię i nazwisko...kl...
 Gimnazjum nr 4 im. Ojca Świętego Jana Pawła II we Wrocławiu SPRAWDZIAN GENETYKA GR. A Imię i nazwisko...kl.... 1. Nauka o regułach i mechanizmach dziedziczenia to: (0-1pkt) a) cytologia b) biochemia c)
Gimnazjum nr 4 im. Ojca Świętego Jana Pawła II we Wrocławiu SPRAWDZIAN GENETYKA GR. A Imię i nazwisko...kl.... 1. Nauka o regułach i mechanizmach dziedziczenia to: (0-1pkt) a) cytologia b) biochemia c)
BIOLOGICZNE BAZY DANYCH (2) GENOMY I ICH ADNOTACJE. Podstawy Bioinformatyki wykład 4
 BIOLOGICZNE BAZY DANYCH (2) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 4 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI 2017/2018 MAGDA MIELCZAREK 2 GENOMY I ICH ADNOTACJE
BIOLOGICZNE BAZY DANYCH (2) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 4 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI 2017/2018 MAGDA MIELCZAREK 2 GENOMY I ICH ADNOTACJE
PODSTAWY BIOINFORMATYKI 6 BAZA DANYCH NCBI - II
 PODSTAWY BIOINFORMATYKI 6 BAZA DANYCH NCBI - II BAZA DANYCH NCBI 1. NCBI 2. Dane gromadzone przez NCBI 3. Przegląd baz danych NCBI: Publikacje naukowe Projekty analizy genomów OMIM: fenotypy człowieka
PODSTAWY BIOINFORMATYKI 6 BAZA DANYCH NCBI - II BAZA DANYCH NCBI 1. NCBI 2. Dane gromadzone przez NCBI 3. Przegląd baz danych NCBI: Publikacje naukowe Projekty analizy genomów OMIM: fenotypy człowieka
GENETYKA POPULACJI. Ćwiczenia 1 Biologia I MGR /
 GENETYKA POPULACJI Ćwiczenia 1 Biologia I MGR 1 ZAGADNIENIA struktura genetyczna populacji obliczanie frekwencji genotypów obliczanie frekwencji alleli przewidywanie struktury następnego pokolenia przy
GENETYKA POPULACJI Ćwiczenia 1 Biologia I MGR 1 ZAGADNIENIA struktura genetyczna populacji obliczanie frekwencji genotypów obliczanie frekwencji alleli przewidywanie struktury następnego pokolenia przy
Analiza zmienności czasowej danych mikromacierzowych
 Systemy Inteligencji Obliczeniowej Analiza zmienności czasowej danych mikromacierzowych Kornel Chromiński Instytut Informatyki Uniwersytet Śląski Plan prezentacji Dane mikromacierzowe Cel badań Prezentacja
Systemy Inteligencji Obliczeniowej Analiza zmienności czasowej danych mikromacierzowych Kornel Chromiński Instytut Informatyki Uniwersytet Śląski Plan prezentacji Dane mikromacierzowe Cel badań Prezentacja
BIOINFORMATYKA. edycja 2016 / wykład 11 RNA. dr Jacek Śmietański
 BIOINFORMATYKA edycja 2016 / 2017 wykład 11 RNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Rola i rodzaje RNA 2. Oddziaływania wewnątrzcząsteczkowe i struktury
BIOINFORMATYKA edycja 2016 / 2017 wykład 11 RNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Rola i rodzaje RNA 2. Oddziaływania wewnątrzcząsteczkowe i struktury
1. System analizy danych NGS z paneli genów
 1. System analizy danych NGS z paneli genów (programistyczny) Sekwenator to instrument odczytujący sekwencję DNA w kilku-kilkudziesieciu probkach na raz. Instrument zapisuje na dysku dane w skompresowanych
1. System analizy danych NGS z paneli genów (programistyczny) Sekwenator to instrument odczytujący sekwencję DNA w kilku-kilkudziesieciu probkach na raz. Instrument zapisuje na dysku dane w skompresowanych
1. Analiza asocjacyjna. Cechy ciągłe. Cechy binarne. Analiza sprzężeń. Runs of homozygosity. Signatures of selection
 BIOINFORMATYKA 1. Wykład wstępny 2. Bazy danych: projektowanie i struktura 3. Równowaga Hardyego-Weinberga, wsp. rekombinacji 4. Analiza asocjacyjna 5. Analiza asocjacyjna 6. Sekwencjonowanie nowej generacji
BIOINFORMATYKA 1. Wykład wstępny 2. Bazy danych: projektowanie i struktura 3. Równowaga Hardyego-Weinberga, wsp. rekombinacji 4. Analiza asocjacyjna 5. Analiza asocjacyjna 6. Sekwencjonowanie nowej generacji
wykład dla studentów II roku biotechnologii Andrzej Wierzbicki
 Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Budowa rybosomu Translacja
Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Budowa rybosomu Translacja
BIOLOGICZNE BAZY DANYCH (1) GENOMY I ICH ADNOTACJE
 BIOLOGICZNE BAZY DANYCH (1) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 2 PODSTAWY BIOINFORMATYKI 2018/2019 MAGDA MIELCZAREK 1 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI
BIOLOGICZNE BAZY DANYCH (1) GENOMY I ICH ADNOTACJE Podstawy Bioinformatyki wykład 2 PODSTAWY BIOINFORMATYKI 2018/2019 MAGDA MIELCZAREK 1 GENOMY I ICH ADNOTACJE NCBI Ensembl UCSC PODSTAWY BIOINFORMATYKI
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii
 Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Transgeneza - genetycznie zmodyfikowane oraganizmy 2. Medycyna i ochrona zdrowia 3. Genomika poznawanie genomów Przełom XX i
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Transgeneza - genetycznie zmodyfikowane oraganizmy 2. Medycyna i ochrona zdrowia 3. Genomika poznawanie genomów Przełom XX i
Dopasowanie sekwencji (sequence alignment)
 Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
POPULARNE POLECENIA SKRYPTY. Pracownia Informatyczna 2
 SKRYPTY Pracownia Informatyczna 2 PRACOWNIA INFORMATYCZNA 2017/2018 MAGDA MIELCZAREK PRACOWNIA INFORMATYCZNA 2017/2018 MAGDA MIELCZAREK 2 cal wyświetlenie kalendarza Składnia: cal 2017, cal Polecenie cal
SKRYPTY Pracownia Informatyczna 2 PRACOWNIA INFORMATYCZNA 2017/2018 MAGDA MIELCZAREK PRACOWNIA INFORMATYCZNA 2017/2018 MAGDA MIELCZAREK 2 cal wyświetlenie kalendarza Składnia: cal 2017, cal Polecenie cal
Bliskie Spotkanie z Biologią. Genetyka populacji
 Bliskie Spotkanie z Biologią Genetyka populacji Plan wykładu 1) Częstości alleli i genotypów w populacji 2) Prawo Hardy ego-weinberga 3) Dryf genetyczny 4) Efekt założyciela i efekt wąskiego gardła 5)
Bliskie Spotkanie z Biologią Genetyka populacji Plan wykładu 1) Częstości alleli i genotypów w populacji 2) Prawo Hardy ego-weinberga 3) Dryf genetyczny 4) Efekt założyciela i efekt wąskiego gardła 5)
Spokrewnienie prawdopodobieństwo, że dwa losowe geny od dwóch osobników są genami IBD. IBD = identical by descent, geny identycznego pochodzenia
 prawdopodobieństwo, że dwa losowe geny od dwóch osobników są genami ID. Relationship Relatedness Kinship Fraternity ID = identical by descent, geny identycznego pochodzenia jest miarą względną. Przyjmuje
prawdopodobieństwo, że dwa losowe geny od dwóch osobników są genami ID. Relationship Relatedness Kinship Fraternity ID = identical by descent, geny identycznego pochodzenia jest miarą względną. Przyjmuje
Czy można zmniejszyć ryzyko występowania defektów genetycznych w populacji polskich koni arabskich?
 Czy można zmniejszyć ryzyko występowania defektów genetycznych w populacji polskich koni arabskich? Monika Bugno-Poniewierska, Monika Stefaniuk-Szmukier, Agata Piestrzyńska-Kajtoch, Agnieszka Fornal, Katarzyna
Czy można zmniejszyć ryzyko występowania defektów genetycznych w populacji polskich koni arabskich? Monika Bugno-Poniewierska, Monika Stefaniuk-Szmukier, Agata Piestrzyńska-Kajtoch, Agnieszka Fornal, Katarzyna
Wykład 9: HUMAN GENOME PROJECT HUMAN GENOME PROJECT
 Wykład 9: Polimorfizm pojedynczego nukleotydu (SNP) odrębność genetyczna, która czyni każdego z nas jednostką unikatową Prof. dr hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej HUMAN GENOME
Wykład 9: Polimorfizm pojedynczego nukleotydu (SNP) odrębność genetyczna, która czyni każdego z nas jednostką unikatową Prof. dr hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej HUMAN GENOME
Sekwencjonowanie RNA po kolei
 ALEKSANDRA ŚWIERCZ Sekwencjonowanie RNA po kolei RNA-seq Module, 2013, www.bioinformatics.ca A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Różnice między eksperymentem mikromacierzowym a RNA-seq Przy
ALEKSANDRA ŚWIERCZ Sekwencjonowanie RNA po kolei RNA-seq Module, 2013, www.bioinformatics.ca A. Świercz ANALIZA DANYCH WYSOKOPRZEPUSTOWYCH 2 Różnice między eksperymentem mikromacierzowym a RNA-seq Przy
Strefa pokrycia radiowego wokół stacji bazowych. Zasięg stacji bazowych Zazębianie się komórek
 Problem zapożyczania kanałów z wykorzystaniem narzędzi optymalizacji Wprowadzenie Rozwiązanie problemu przydziału częstotliwości prowadzi do stanu, w którym każdej stacji bazowej przydzielono żądaną liczbę
Problem zapożyczania kanałów z wykorzystaniem narzędzi optymalizacji Wprowadzenie Rozwiązanie problemu przydziału częstotliwości prowadzi do stanu, w którym każdej stacji bazowej przydzielono żądaną liczbę
Rozkład materiału z biologii dla klasy III AD. 7 godz / tyg rok szkolny 2016/17
 Rozkład materiału z biologii dla klasy III AD zakres rozszerzony LO 7 godz / tyg rok szkolny 2016/17 Biologia na czasie 2 zakres rozszerzony nr dopuszczenia 564/2/2012 Biologia na czasie 3 zakres rozszerzony
Rozkład materiału z biologii dla klasy III AD zakres rozszerzony LO 7 godz / tyg rok szkolny 2016/17 Biologia na czasie 2 zakres rozszerzony nr dopuszczenia 564/2/2012 Biologia na czasie 3 zakres rozszerzony
PRZYRÓWNANIE SEKWENCJI
 http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
Choroba syropu klonowego
 Choroba syropu klonowego Geny i zespoły genetyczne Gen Choroba/objawy Sposób dziedziczenia Znane warianty chorobotwórcze BCKDHA Maple syrup urine disease AR 40 BCKDHB Maple syrup urine disease AR 64 DBT
Choroba syropu klonowego Geny i zespoły genetyczne Gen Choroba/objawy Sposób dziedziczenia Znane warianty chorobotwórcze BCKDHA Maple syrup urine disease AR 40 BCKDHB Maple syrup urine disease AR 64 DBT
Dane mikromacierzowe. Mateusz Markowicz Marta Stańska
 Dane mikromacierzowe Mateusz Markowicz Marta Stańska Mikromacierz Mikromacierz DNA (ang. DNA microarray) to szklana lub plastikowa płytka (o maksymalnych wymiarach 2,5 cm x 7,5 cm) z naniesionymi w regularnych
Dane mikromacierzowe Mateusz Markowicz Marta Stańska Mikromacierz Mikromacierz DNA (ang. DNA microarray) to szklana lub plastikowa płytka (o maksymalnych wymiarach 2,5 cm x 7,5 cm) z naniesionymi w regularnych
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZE W BIOLOGII 1. Wykład wstępny. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. ajczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZE W BIOLOGII 1. Wykład wstępny. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. ajczęściej wykorzystywane testy statystyczne
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych???
 Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Temat 6: Genetyczne uwarunkowania płci. Cechy sprzężone z płcią.
 Temat 6: Genetyczne uwarunkowania płci. Cechy sprzężone z płcią. 1. Kariotyp człowieka. 2. Determinacja płci u człowieka. 3. Warunkowanie płci u innych organizmów. 4. Cechy związane z płcią. 5. Cechy sprzężone
Temat 6: Genetyczne uwarunkowania płci. Cechy sprzężone z płcią. 1. Kariotyp człowieka. 2. Determinacja płci u człowieka. 3. Warunkowanie płci u innych organizmów. 4. Cechy związane z płcią. 5. Cechy sprzężone
Przyrównanie sekwencji. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Przyrównanie sekwencji Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych),
Przyrównanie sekwencji Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych),
Mitochondrialna Ewa;
 Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Wielotorbielowatość wątroby
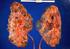 Wielotorbielowatość wątroby Wielotorbielowatość wątroby cechuje występowanie w obrębie wątroby obszernych torbieli, czyli przestrzeni wypełnionych płynem. Istnieją trzy główne postacie choroby - w najczęstszym
Wielotorbielowatość wątroby Wielotorbielowatość wątroby cechuje występowanie w obrębie wątroby obszernych torbieli, czyli przestrzeni wypełnionych płynem. Istnieją trzy główne postacie choroby - w najczęstszym
Choroby genetyczne na tle zmian w genomie człowieka rodzaje, fenotyp, diagnostyka genetyczna
 Przedmiot: Genetyka kliniczna V Rok, Wydział Lekarski I Katedra i Zakład Genetyki Medycznej UM w Poznaniu Choroby genetyczne na tle zmian w genomie człowieka rodzaje, fenotyp, diagnostyka genetyczna Opracowanie:
Przedmiot: Genetyka kliniczna V Rok, Wydział Lekarski I Katedra i Zakład Genetyki Medycznej UM w Poznaniu Choroby genetyczne na tle zmian w genomie człowieka rodzaje, fenotyp, diagnostyka genetyczna Opracowanie:
Informacje dla pacjentów i rodzin
 16 Zmiany chromosomowe Wrzesień 2007 Ta praca została wsparta przez EuroGentest, EU-FP6 supported NoE numer kontraktu 512148 Tłumaczone z języka angielskiego przez Ewę Piotrowską i Annę Kloskę, Uniwersytet
16 Zmiany chromosomowe Wrzesień 2007 Ta praca została wsparta przez EuroGentest, EU-FP6 supported NoE numer kontraktu 512148 Tłumaczone z języka angielskiego przez Ewę Piotrowską i Annę Kloskę, Uniwersytet
Mutacje jako źródło różnorodności wewnątrzgatunkowej
 Mutacje jako źródło różnorodności wewnątrzgatunkowej Zajęcia terenowe: Zajęcia w klasie: Poziom nauczania oraz odniesienie do podstawy programowej: Liceum IV etap edukacyjny zakres rozszerzony: Różnorodność
Mutacje jako źródło różnorodności wewnątrzgatunkowej Zajęcia terenowe: Zajęcia w klasie: Poziom nauczania oraz odniesienie do podstawy programowej: Liceum IV etap edukacyjny zakres rozszerzony: Różnorodność
Ekologia molekularna. wykład 11
 Ekologia molekularna wykład 11 Sekwencjonowanie nowej generacji NGS = next generation sequencing = high throughput sequencing = massive pararell sequencing =... Różne techniki i platformy Illumina (MiSeq,
Ekologia molekularna wykład 11 Sekwencjonowanie nowej generacji NGS = next generation sequencing = high throughput sequencing = massive pararell sequencing =... Różne techniki i platformy Illumina (MiSeq,
Bioinformatyka. Rodzaje Mutacji
 Bioinformatyka (wykład monograficzny) wykład 3. E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Rodzaje Mutacji zmienność sekwencji (sequence variation) mutacje polimorfizm
Bioinformatyka (wykład monograficzny) wykład 3. E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas Rodzaje Mutacji zmienność sekwencji (sequence variation) mutacje polimorfizm
Perspektywy zastosowania badań genomicznych w hodowli zwierząt
 Wiadomości Zootechniczne, R. XLIX (2011), 4: 103 108 Perspektywy zastosowania badań genomicznych w hodowli zwierząt W prowadzenie Wobec stałego wzrostu zaludnienia, rolnictwo odgrywa kluczową rolę w globalnym
Wiadomości Zootechniczne, R. XLIX (2011), 4: 103 108 Perspektywy zastosowania badań genomicznych w hodowli zwierząt W prowadzenie Wobec stałego wzrostu zaludnienia, rolnictwo odgrywa kluczową rolę w globalnym
Przewlekła choroba ziarniniakowa
 Przewlekła choroba ziarniniakowa Przewlekła choroba ziarniniakowa związana jest z defektami aktywności granulocytów obojętnochłonnych. Choroba typowo objawia się nawracającymi, ciężkimi infekcjami bakteryjnymi
Przewlekła choroba ziarniniakowa Przewlekła choroba ziarniniakowa związana jest z defektami aktywności granulocytów obojętnochłonnych. Choroba typowo objawia się nawracającymi, ciężkimi infekcjami bakteryjnymi
Dobór naturalny. Ewolucjonizm i eugenika
 Dobór naturalny Ewolucjonizm i eugenika Silna i słaba selekcja - symulacje W cieniu eugeniki Początki - XIX w. (Francis Galton) XX w. - eugenika totalitarna Poprawa jakości gatunku ludzkiego poprzez kierowanie
Dobór naturalny Ewolucjonizm i eugenika Silna i słaba selekcja - symulacje W cieniu eugeniki Początki - XIX w. (Francis Galton) XX w. - eugenika totalitarna Poprawa jakości gatunku ludzkiego poprzez kierowanie
prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie
 prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie Sekwencyjność występowania zaburzeń molekularnych w niedrobnokomórkowym raku płuca
prof. Joanna Chorostowska-Wynimko Zakład Genetyki i Immunologii Klinicznej Instytut Gruźlicy i Chorób Płuc w Warszawie Sekwencyjność występowania zaburzeń molekularnych w niedrobnokomórkowym raku płuca
Rak tarczycy - prognostyka
 Rak tarczycy - prognostyka Wariant rs966423-tt w genie DIRC3 jest czynnikiem rokowniczo niekorzystnym, związanym ze zwiększonym ryzykiem zgonu w przebiegu raka zróżnicowanego tarczycy (Świerniak et al.
Rak tarczycy - prognostyka Wariant rs966423-tt w genie DIRC3 jest czynnikiem rokowniczo niekorzystnym, związanym ze zwiększonym ryzykiem zgonu w przebiegu raka zróżnicowanego tarczycy (Świerniak et al.
Sekwencje akinezji płodu
 Sekwencje akinezji płodu Geny i zespoły genetyczne Gen Choroba/objawy Sposób dziedziczenia Znane warianty chorobotwórcze CHRNA1 Myasthenic syndrome, congenital AD/AR 19 CHRND Myasthenic syndrome AD/AR
Sekwencje akinezji płodu Geny i zespoły genetyczne Gen Choroba/objawy Sposób dziedziczenia Znane warianty chorobotwórcze CHRNA1 Myasthenic syndrome, congenital AD/AR 19 CHRND Myasthenic syndrome AD/AR
Choroba Leśniowskiego i Crohna
 Choroba Leśniowskiego i Crohna Choroba Leśniowskiego i Crohna należy do nieswoistych chorób zapalnych jelita, może dotyczyć każdego odcinka przewodu pokarmowego. Rokrocznie zapada na nią 1 na 10 000 osób;
Choroba Leśniowskiego i Crohna Choroba Leśniowskiego i Crohna należy do nieswoistych chorób zapalnych jelita, może dotyczyć każdego odcinka przewodu pokarmowego. Rokrocznie zapada na nią 1 na 10 000 osób;
Kwasica metylomalonowa
 Kwasica metylomalonowa Kwasica metylomalonowa to choroba u podstawy której leżą zaburzenia metabolizmu kwasu metylomalonowego i kobalaminy (witamina B12). Choroba ma bardzo różnorodną postać, od form o
Kwasica metylomalonowa Kwasica metylomalonowa to choroba u podstawy której leżą zaburzenia metabolizmu kwasu metylomalonowego i kobalaminy (witamina B12). Choroba ma bardzo różnorodną postać, od form o
Sekwencjonowanie DNA
 BIOINFORMATYKA edycja 2016 / 2017 wykład 7 Sekwencjonowanie DNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Techniki sekwencjonowania 2. Problemy bioinformatyczne
BIOINFORMATYKA edycja 2016 / 2017 wykład 7 Sekwencjonowanie DNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Techniki sekwencjonowania 2. Problemy bioinformatyczne
Hemochromatoza. Gen Choroba/objawy Sposób dziedziczenia. HAMP Hemochromatoza, Choroba Alzheimera, postać późna AR 2
 Hemochromatoza Hemochromatoza jest związana z nadmiernym wchłanianiem żelaza w jelitach i zwiększonym gromadzeniem tego pierwiastka w tkankach. Choroba jest najczęściej dziedziczona w sposób autosomalny
Hemochromatoza Hemochromatoza jest związana z nadmiernym wchłanianiem żelaza w jelitach i zwiększonym gromadzeniem tego pierwiastka w tkankach. Choroba jest najczęściej dziedziczona w sposób autosomalny
Hemochromatoza. Gen Choroba/objawy Sposób dziedziczenia. HAMP Hemochromatosis AR 5. HFE Hemochromatosis, choroba Alzheimera, postać późna AR/Digenic 7
 Hemochromatoza Hemochromatoza jest związana z nadmiernym wchłanianiem żelaza w jelitach i zwiększonym gromadzeniem tego pierwiastka w tkankach. Choroba jest najczęściej dziedziczona w sposób autosomalny
Hemochromatoza Hemochromatoza jest związana z nadmiernym wchłanianiem żelaza w jelitach i zwiększonym gromadzeniem tego pierwiastka w tkankach. Choroba jest najczęściej dziedziczona w sposób autosomalny
Zaburzenia metabolizmu kreatyny
 Zaburzenia metabolizmu kreatyny Kreatyna jest związkiem, który u człowieka występuje głównie w mięśniach i jest wykorzystywany do magazynowania i uwalniania energii, wykorzystywanej w wielu procesach komórkowych.
Zaburzenia metabolizmu kreatyny Kreatyna jest związkiem, który u człowieka występuje głównie w mięśniach i jest wykorzystywany do magazynowania i uwalniania energii, wykorzystywanej w wielu procesach komórkowych.
Wrodzony przerost nadnerczy
 Wrodzony przerost nadnerczy Geny i zespoły genetyczne Gen Choroba/objawy Sposób dziedziczenia Znane warianty chorobotwórcze CYP11B1 Wrodzony przerost nadnerczy AD/AR 22 CYP17A1 Wrodzony przerost nadnerczy
Wrodzony przerost nadnerczy Geny i zespoły genetyczne Gen Choroba/objawy Sposób dziedziczenia Znane warianty chorobotwórcze CYP11B1 Wrodzony przerost nadnerczy AD/AR 22 CYP17A1 Wrodzony przerost nadnerczy
Testowanie hipotez statystycznych
 9 października 2008 ...czyli definicje na rozgrzewkę n-elementowa próba losowa - wektor n zmiennych losowych (X 1,..., X n ); intuicyjnie: wynik n eksperymentów realizacja próby (X 1,..., X n ) w ω Ω :
9 października 2008 ...czyli definicje na rozgrzewkę n-elementowa próba losowa - wektor n zmiennych losowych (X 1,..., X n ); intuicyjnie: wynik n eksperymentów realizacja próby (X 1,..., X n ) w ω Ω :
Zespół Walkera-Warburga
 Zespół Walkera-Warburga Zespół Walkera-Warburga to jedna z najcięższych postaci dystrofii mięśniowych. Objawy choroby są widoczne już w pierwszych miesiącach życia, kiedy stwierdza się obniżone napięcie
Zespół Walkera-Warburga Zespół Walkera-Warburga to jedna z najcięższych postaci dystrofii mięśniowych. Objawy choroby są widoczne już w pierwszych miesiącach życia, kiedy stwierdza się obniżone napięcie
Podstawy genetyki człowieka. Cechy wieloczynnikowe
 Podstawy genetyki człowieka Cechy wieloczynnikowe Dziedziczenie Mendlowskie - jeden gen = jedna cecha np. allele jednego genu decydują o barwie kwiatów groszku Bardziej złożone - interakcje kilku genów
Podstawy genetyki człowieka Cechy wieloczynnikowe Dziedziczenie Mendlowskie - jeden gen = jedna cecha np. allele jednego genu decydują o barwie kwiatów groszku Bardziej złożone - interakcje kilku genów
Zespół Robinowa. Gen Choroba/objawy Sposób dziedziczenia. DVL1 Zespół Robinowa AD 17. ROR2 Zespół Robinow, Brachydaktylia AD/AR 17
 Zespół Robinowa Wyróżnia się dwie formy zespołu Robinowa zależnie od sposobu dziedziczenia choroby. Forma o dziedziczeniu autosomalnym recesywnym przebiega z większymi zaburzeniami niż postać dziedziczona
Zespół Robinowa Wyróżnia się dwie formy zespołu Robinowa zależnie od sposobu dziedziczenia choroby. Forma o dziedziczeniu autosomalnym recesywnym przebiega z większymi zaburzeniami niż postać dziedziczona
a) Zapisz genotyp tego mężczyzny... oraz zaznacz poniżej (A, B, C lub D), jaki procent gamet tego mężczyzny będzie miało genotyp ax b.
 W tomie 2 zbioru zadań z biologii z powodu nieprawidłowego wprowadzenia komendy przenoszenia spójników i przyimków do następnej linii wystąpiła zamiana samotnych dużych liter (A, I, W, U) na małe litery.
W tomie 2 zbioru zadań z biologii z powodu nieprawidłowego wprowadzenia komendy przenoszenia spójników i przyimków do następnej linii wystąpiła zamiana samotnych dużych liter (A, I, W, U) na małe litery.
ZARZĄDZANIE POPULACJAMI ZWIERZĄT 1. RÓWNOWAGA GENETYCZNA POPULACJI. Prowadzący: dr Wioleta Drobik Katedra Genetyki i Ogólnej Hodowli Zwierząt
 ZARZĄDZANIE POPULACJAMI ZWIERZĄT 1. RÓWNOWAGA GENETYCZNA POPULACJI Fot. W. Wołkow Prowadzący: dr Wioleta Drobik Katedra Genetyki i Ogólnej Hodowli Zwierząt POPULACJA Zbiór organizmów żywych, które łączy
ZARZĄDZANIE POPULACJAMI ZWIERZĄT 1. RÓWNOWAGA GENETYCZNA POPULACJI Fot. W. Wołkow Prowadzący: dr Wioleta Drobik Katedra Genetyki i Ogólnej Hodowli Zwierząt POPULACJA Zbiór organizmów żywych, które łączy
Profilowanie somatyczne BRCA1, BRCA2
 Profilowanie somatyczne BRCA1, BRCA2 Nowotwór złośliwy rozwija się w momencie, gdy w DNA komórek nagromadzone zostaną liczne mutacje. Od rodzaju tych mutacji zależy dobór właściwego schematu leczenia,
Profilowanie somatyczne BRCA1, BRCA2 Nowotwór złośliwy rozwija się w momencie, gdy w DNA komórek nagromadzone zostaną liczne mutacje. Od rodzaju tych mutacji zależy dobór właściwego schematu leczenia,
Acrodermatitis enteropathica
 Acrodermatitis enteropathica Acrodermatitis enteropathica (zespół Brandta) to choroba związana z uszkodzeniem białka odpowiedzialnego za wchłanianie cynku. Objawy choroby ujawniają się w pierwszych miesiącach
Acrodermatitis enteropathica Acrodermatitis enteropathica (zespół Brandta) to choroba związana z uszkodzeniem białka odpowiedzialnego za wchłanianie cynku. Objawy choroby ujawniają się w pierwszych miesiącach
STATYSTYKA MATEMATYCZNA
 STATYSTYKA MATEMATYCZA 1. Wykład wstępny. Zmienne losowe i teoria prawdopodobieństwa 3. Populacje i próby danych 4. Testowanie hipotez i estymacja parametrów 5. ajczęściej wykorzystywane testy statystyczne
STATYSTYKA MATEMATYCZA 1. Wykład wstępny. Zmienne losowe i teoria prawdopodobieństwa 3. Populacje i próby danych 4. Testowanie hipotez i estymacja parametrów 5. ajczęściej wykorzystywane testy statystyczne
Plan wykładu: Budowa chromatyny - nukleosomy. Wpływ nukleosomów na replikację i transkrypcję
 Nukleosomy 1 Plan wykładu: Budowa chromatyny - nukleosomy Wpływ nukleosomów na replikację i transkrypcję Metody pozwalające na wyznaczanie miejsc wiązania nukleosomów Charakterystyka obsadzenia nukleosomów
Nukleosomy 1 Plan wykładu: Budowa chromatyny - nukleosomy Wpływ nukleosomów na replikację i transkrypcję Metody pozwalające na wyznaczanie miejsc wiązania nukleosomów Charakterystyka obsadzenia nukleosomów
Adres strony internetowej, na której Zamawiający udostępnia Specyfikację Istotnych Warunków Zamówienia:
 Adres strony internetowej, na której Zamawiający udostępnia Specyfikację Istotnych Warunków Zamówienia: www.inhort.pl/przetargi_2013_lista.html Skierniewice: Sukcesywne usługi sekwencjonowania DNA i transkryptomów
Adres strony internetowej, na której Zamawiający udostępnia Specyfikację Istotnych Warunków Zamówienia: www.inhort.pl/przetargi_2013_lista.html Skierniewice: Sukcesywne usługi sekwencjonowania DNA i transkryptomów
Zespół hemolityczno-mocznicowy
 Zespół hemolityczno-mocznicowy Badanie obejmuje: analizę sekwencji genów ADAMTS13; C3; CD46; CFB; CFH; CFI; THBD; CFHR1; CFHR2; CFHR3; CFHR4; CFHR5 metodą NGS analizę poziomu przeciwciał przeciw czynnikowi
Zespół hemolityczno-mocznicowy Badanie obejmuje: analizę sekwencji genów ADAMTS13; C3; CD46; CFB; CFH; CFI; THBD; CFHR1; CFHR2; CFHR3; CFHR4; CFHR5 metodą NGS analizę poziomu przeciwciał przeciw czynnikowi
Mutacje. delecja insercja strukturalne
 Mutacje Genowe (Punktowe) Chromosomowe substytucje: delecja insercja strukturalne liczbowe (genomowe) tranzycja delecja deficjencja transwersja duplikacja translokacja inwersja Mutacje chromosomowe strukturalne
Mutacje Genowe (Punktowe) Chromosomowe substytucje: delecja insercja strukturalne liczbowe (genomowe) tranzycja delecja deficjencja transwersja duplikacja translokacja inwersja Mutacje chromosomowe strukturalne
Zespół Alporta. Gen Choroba/objawy Sposób dziedziczenia. COL4A3 Zespół Alporta AD/AR 100. COL4A4 Zespół Alporta AD/AR 84. COL4A5 Zespół Alporta XL 583
 Zespół Alporta Zespół Alporta jest przykładem zespołu wad wrodzonych ze współistniejącym niedosłuchem. Charakteryzuje się występowaniem nefropatii (choroby nerek), powodowanej zaburzeniami powstawania
Zespół Alporta Zespół Alporta jest przykładem zespołu wad wrodzonych ze współistniejącym niedosłuchem. Charakteryzuje się występowaniem nefropatii (choroby nerek), powodowanej zaburzeniami powstawania
