Małe RNA i epigenetyczna regulacja ekspresji genów
|
|
|
- Grzegorz Wojciechowski
- 6 lat temu
- Przeglądów:
Transkrypt
1 Małe RNA i epigenetyczna regulacja ekspresji genów Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas) srna nt wyciszanie ekspresji genów: (gene silencing, RNA silencing) post-transkrypcyjne (post-transcriptional gene silencing, PTGS) degradacja mrna, inhibicja translacji transkrypcyjne (transcriptional gene silencing, TGS) epigenetyczne modyfikacje chromatyny AAAAA RNA Pol modyfikacja histonów, metylacja DNA 2 1
2 PTGS: post-transkrypcyjne wyciszanie genów Klasy małych RNA: mirna (microrna) rośliny, zwierzęta, wirusy, Protista 20 25nt Drosha (u zwierząt) + Dicer Transkrypcja przez Pol II/Pol III Regulacja stabilności mrna (cięcie mrna), inhibicja translacji mirtrony pochodzące z intronów prekursorów mrna genów kodujących białka; występują u zwierząt; niezależne od Drosha sirna (small interferring RNA) większość działa in cis, za wyjątkiem tasirna exo-sirna (pochodzenia egzogennego) rośliny, grzyby, zwierzęta, Protista 21-24nt Dicer Transgeniczny, wirusowy lub inny egzogenny RNA Post-transkrypcyjna regulacja ekspresji genów, obrona przeciwwirusowa endo-sirna (pochodzenia endogennego) rośliny, grzyby, zwierzęta, Protista ~21nt Dicer Transkrypcja dwukierunkowa lub zbieżna, wiązanie mrna z transkryptami pseudogenów o przeciwnej orientacji Post-transkrypcyjna i transkrypcyjna regulacja ekspresji genów, regulacja aktywności transpozonów tasirna (trans-acting sirna) rośliny 21nt DCL4 TAS RNA cięte przez mirna Regulacja posttranskrypcyjna natsirna (natural antisense transcripts-derived sirna) rośliny 24nt 21nt DCL2 DCL1 Transkrypcja dwukierunkowa indukowana stresem Regulacja genów odpowiedzi na stres 3 TGS: transkrypcyjne wyciszanie genów Klasy małych RNA: sirna (small interferring RNA) endo-sirna (pochodzenia endogennego) hc-sirna (heterochromatic sirna) rośliny, grzyby, zwierzęta, Protista rośliny, S. pombe ~21nt Dicer Transkrypcja dwukierunkowa lub zbieżna, wiązanie mrna z transkryptami pseudogenów o przeciwnej orientacji Post-transkrypcyjna i transkrypcyjna regulacja ekspresji genów, regulacja aktywności transpozonów 24-26nt DCL3 Transpozony, powtórzenia Modyfikacja chromatyny pirna (Piwi-interacting RNA) Drosophila, C. elegans, ssaki, Danio rerio 24 30nt niezależne od Dicer Długie pierwotne transkrypty (?) Regulacja aktywności transpozonów, inne nieznane funkcje 4 2
3 Białka Dicer i Argonaute (AGO): główne elementy szlaku wyciszania RNA Dwuniciowy RNA (double-stranded RNA, dsrna) jest cięty przez Dicer na krótkie dwuniciowe cząsteczki RNA, które są wiązane przez jedno z białek rodziny ARGONAUTE. AGO wyciszenie 5 Dicer i Dicer-like Podczas biogenezy sirna i mirna, białka Dicer lub Dicer-like (DCL) tną długie dsrna lub RNA o strukturze spinki (hairpin) na fragmenty ~ 21 25nt. RIII PAZ RIII Struktura białka Dicer umożliwia równomierne rozcinanie RNA na fragmenty o takiej samej długości. MacRae, I.J., Zhou, K., Li, F., Repic, A., Brooks, A.N., Cande, W.., Adams, P.D., and Doudna, J.A. (2006) Science 6 3
4 Dicer i Dicer-like RIII PAZ konserwowana domena wiążąca dsrna (dsrbd- dsrna-binding domain) oddziałuje z końcem 3 dsrna PAZ RIII 2 domeny typu RNazy III aktywność endonukleolityczna 5 P / 3 OH MacRae, I.J., Zhou, K., Li, F., Repic, A., Brooks, A.N., Cande, W.., Adams, P.D., and Doudna, J.A. (2006) Science 7 Argonaute Białka ARGONAUTE wiążą srna i mrna PAZ PAZ oddziałuje z końcem 3 srna MID (middle) oddziałuje z nukleotydem na końcu 5 srna PIWI MID PIWI struktura podobna do RNazyH w niektórych białkach Ago: cięcie RNA zwiazanego z srna (slicer activity) Nazwa ARGONAUTE pochodzi od mutanta argonaute1 A. thaliana; który swoim kształtem przypomina ośmiornicę żeglarka (Argonauta). A. thaliana ago1 Argonauta argo 8 Bohmert, K., Camus, I., Bellini, C., Bouchez, D., Caboche, M., and Benning, C. (1998) EMBO J. Song, J.-J., Smith, S.K., Hannon, G.J., and Joshua-Tor, L. (2004) Science 4
5 Dicer i Argonaute u różnych organizmów Plantae Fungi Metazoa Argonaute-PIWI-like Species Argonaute PIWI Dicer-like RdRP Arabidopsis thaliana 10 (AGO1-10) - 4 (DCL 1-4) 6 Oryza sativa Saccharomyces cerevisiae Schizosaccharomyces pombe Neurospora crassa Aspergillus nidulans Caenorhabditis elegans (Dicer + Drosha) 4 Drosophila melanogaster (2 Dicers + Drosha) - Danio rerio (Dicer + Drosha) - Homo sapiens (Dicer + Drosha) - 9 U roślin w biogenezie mirna i sirna uczestniczą różne białka DCL AtDCL1 mirna AtDCL2-4 sirna Rośliny mają 4 (i więcej) białek DCL: większa niż u innych organizmów liczba białek DCL umożliwia roślinom bardziej precyzyjną i skuteczną obronę przed patogenami. Margis, R., Fusaro, A.F., Smith, N.A., Curtin, S.J., Watson, J.M., Finnegan, E.J., and Waterhouse, P.M. (2006) FEBS Lett. 10 5
6 U roślin w biogenezie mirna i sirna uczestniczą różne białka DCL AtDCL1 mirna Arabidopsis thaliana: DCL1 21nt mirna AGO1/7/10 DCL2 22nt sirna DCL3 24nt sirna AGO4/6 DCL4 21nt sirna (tasirna) AGO1 AtDCL2-4 sirna 11 Bologna N.G., Voinnet O. (2014) Annu. Rev. Plant Biol. Exo-siRNA: wyciszanie transgenów Transgeny wprowadzane sztucznie są często wyciszane przez sirna Wyciszenie może być wywołane: bardzo wysokim poziomem ekspresji transgenu dwuniciowym RNA pochodzącym z ekspresji transgenu nieprawidłowymi RNA pochodzącymi z ekspresji transgenu Transgeny są wyciszane post-transkrypcyjnie i transkrypcyjnie sirna-posttranskrypcyjne lub transkrypcyjne wyciszanie ekspresji genów RISC (RNA-induced silencing complex) AGO AGO RNA Pol 12 6
7 Post-transkrypcyjne wyciszanie genów u roślin transgene-induced post-transcriptional silencing Eksperymenty modyfikacji koloru kwiatów petunii (Petunia hybrida) - odkrycie mechanizmu wyciszania genów jądro komórkowe komórka roślinna DNA Agrobacterium tumefaciens na powierzchni komórki roślinnej Martha Hawes, University of Arizona 13 Manipulacja ekspresją genu syntazy chalkonowej: zmiana pigmentacji kwiatów petunii CHS Dzikie kwiaty petunii zawierają antocyjaniny nadające purpurową barwę syntaza chalkonowa (CHS) enzym szlaku biosyntezy antocyjanin antocyjaniny 14 Aksamit-Stachurska et al. BMC Biotechnology 2008 Foto: Richard Jorgensen 7
8 Oczekiwanie synteza RNA o prawidłowej orientacji pogłębi barwę kwiatów... gen PRO ORF mrna translacja mrna transgen konstrukt o orientacji sens PRO ORF mrna dodatkowa translacja mrna mrna 15 a synteza RNA o odwróconej orientacji zablokuje syntezę barwnika gen PRO ORF mrna translacja mrna transgen konstrukt o orientacji sens PRO ORF mrna dodatkowa translacja mrna mrna transgen konstrukt o orientacji antysens PRO ORF RNA antysensowny tworzenie dupleksów RNA sens-antysens inhibicja translacji 16 8
9 Nieoczekiwanie, wprowadzenie zarówno konstruktów sens jak i antysens prowadzi do inhibicji syntezy barwnika rośliny zawierające transgen CHS CaMV 35S pro : CHS lub CaMV 35S pro : sens antysens CHS 17 Foto: Richard Jorgensen W modyfikowanych roślinach nie dochodzi do syntezy ani endogennej ani transgenicznej CHS purpurowe kwiaty białe kwiaty Zjawisko wyciszania zarówno sztucznie wprowadzonego genu, jak i jego endogennego odpowiednika nazywamy kosupresją transgeniczny RNA endogenny RNA RNase protection 18 Napoli, C., Lemieux, C., and Jorgensen, R. (1990) Plant Cell 9
10 Kosupresja jest wynikiem produkcji sirna typ dziki PRO gen ORF mrna translacja mrna roślina transgeniczna kosupresja PRO ORF konstrukt sens PRO ORF gen mrna mrna produkcja sirna De Paoli, E., Dorantes-Acosta, A., Zhai, J., Accerbi, M., Jeong, D.-H., Park, S., Meyers, B.C., Jorgensen, R.A., and Green, P.J. (2009). RNA 19 VIGS - viral induced gene silencing wirusowa polimeraza RNA zależna od RNA wirusowy ssrna Większość roślinnych wirusów to wirusy RNA, których replikacja odbywa się poprzez dsrna wirusowy dsrna dsrna jest cięty przez DCL4 powstają sirna które wiążą się z AGO1 wyciszenie replikacji wirusa AGO1 AGO
11 VIGS - viral induced gene silencing Obrona przeciwwirusowa u roślin: głównie na poziomie wyciszania RNA TRV: 21-nt (DCL4), 24-nt sirna (DCL3) TCV (Turnip Crinkle Virus): 22-nt sirna (DCL2) degradacja wirusowego RNA: 21-nt i 24-nt sirna WT Arabidopsis inokulowany TRV Podwójny mutant dcl2-dcl4 inokulowany TRV Waterhouse, P.M. and Fusaro, A.F. (2006) Science. Wyciszanie wirusa TRV Tobacco Rattle Virus (wirus nekrotycznej kędzierzawki tytoniu) w roślinach A. thaliana dzikiego typu zapobiega objawom choroby. Mutanty dcl są niezdolne do zahamowania infekcji. Mutanty biogenezy sirna są mniej odporne na choroby wirusowe 21 Deleris, A., Gallego-Bartolome, J., Bao, J., Kasschau, K., Carrington, J.C., and Voinnet, O. (2006) Science Infekcja wirusowa powoduje akumulację sirna Małe RNA komplementarne do wirusowego RNA są obecne zarówno w liściach inokulowanych, jak i w młodszych dystalnych liściach (ale nie w liściach kontrolnych) liść liść inokulowany dystalny dni po inokulacji liść inokulowany Northern blot Hamilton, A.J., and Baulcombe, D.C. (1999) Science 22 11
12 Sygnał wyciszający może rozprzestrzeniać się w całej roślinie poprzez floem inokulowany liść wyciszenie egzogenne sirna 21 i 24nt (PTGS) wyciszanie transgenu GFP endogenne 24nt sirna (TGS poprzez remodelowanie chromatyny, pochodzą z transpozonów i innych zmetylowanych rejonów genomu) mirna (PTGS) Sarkies P., Miska E.A. (2014) Nat. Rev. MCB 23 VIGS - podsumowanie Wyciszanie ekspresji genów za pośrednictwem sirna jest ważnym mechanizmem obrony roślin przed patogenami wirusami i bakteriami sirna hamują replikację wirusową u roślin (również u zwierząt - Drosophila melanogaster) sirna działają ogólnoustrojowo u roślin mogą się przemieszczać poprzez wiązki przewodzące Większość wirusów wytwarza białka supresorowe, hamujące szlak wyciszania 24 12
13 Exo-siRNA u zwierząt: dsrna najsilniejszy inicjator wyciszania modyfikacje genetyczne nicienia C. elegans: Wprowadzanie do organizmów nicieni Caenorhabditis elegans RNA o prawidłowej lub odwróconej orientacji, oraz dwuniciowych RNA, odpowiadających sekwencji genu unc-22, kodującego miofilamentowe białko komórek mięśniowych. Wyciszenie unc-22 powoduje fenotyp kurczenia ciała (twitching) RNA sens bez zmian fenotypowych RNA antysens bez zmian fenotypowych dsrna fenotyp twitching 25 Fire, A. et al., (1998) Nature Exo-siRNA u zwierząt: U ssaków i C. elegans tylko jedno białko Dicer, odpowiada za biogenezę zarówno sirna, jak i mirna Drosophila melanogaster: Dcr1 mirna Dcr2 sirna, wiązane przez AGO2 obrona przeciwwirusowa egzogenny dsrna wirusowy dsrna cięcie mrna obrona przeciwwirusowa 26 Okamura K. & Lai E.C. (2008) Mol Cell Biol 13
14 Wyciszenie może się rozprzestrzeniać dzięki aktywności RdRP (RNAdependent RNA polymerase) i biogenezie 2-rzędowych sirna. Efekt wyciszenia jest wzmacniany przez amplifikację sirna 1 sirna 1 sirna primary Argonaute 2 sirna 2 sirna Ghildiyal & Zamore (2009) Nat Rev Genet 27 Endo-siRNA: 28 Ghildiyal & Zamore (2009) Nat Rev Genet 14
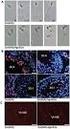
15 Większość endogennych sirna powstaje z transpozonów i powtórzeń sekwencji DNA Chromosom Centromer małe RNA transpozony retrotranspozony Wysoko-przepustowe sekwencjonowanie małych RNA Większość sirna w komórce pochodzi z sekwencji transpozonowych i powtórzonych. U Arabidopsis thaliana duże zagęszczenie tego typu sekwencji występuje w rejonach centromerowych chromosomów. Kasschau, K.D., Fahlgren, N., Chapman, E.J., Sullivan, C.M., Cumbie, J.S., Givan, S.A., and Carrington, J.C. (2007) PLoS Biol 29 Endo-siRNA u roślin: cis-acting sirna (casirna, hc-sirna) trans-acting sirna (tasirna) natural antisense sirna (natsirna) 30 Ghildiyal & Zamore (2009) Nat Rev Genet 15
16 tasirna: roślinne endogenne sirna gen TAS tasirna trans-acting sirna specyficzne dla roślin kodowane przez geny TAS obróbka pierwotnego transkryptu jest inicjowana przez mirna RNA Pol II transkrypcja locus TAS przez polimerazę RNA II AGO wiązanie przez mirna i cięcie rekrutacja RDR6 RDR6 synteza drugiej nici RNA przez RDR6 (RNA-dependent RNA polymerase) 31 biogeneza tasirna c.d. dsrna jest cięty przez DCL4 na serię krótszych dsrna, uwalniając wiele cząsteczek tasirna z jednego genu TAS
17 Z jednego genu TAS powstaje wiele cząsteczek tasirna miejsce cięcia pierwotnego transkryptu przy udziale mirna kluczowe dla zapewnienia specyficzności tasirna; DCL4 zaczyna ciąć prekursor dokładnie w tym miejscu i tnie w odstępach 21nt tasirna mogą powstawać z obu nici RNA DCL4 przesuwa się wzdłuż dsrna, odmierzając i tnąc 33 Allen, E., Xie, Z., Gustafson, A M., and Carrington, J.C. (2005) Cell Zaburzenia biogenezy tasirna zaburzenia rozwoju mutacja rdr6-15: brak tasirna zip-1 (AGO7): brak TAS3 tasirna Arabidopsis 4 rodziny genów TAS TAS1 i TAS2 tasirna PPR TAS3 tasirna czynniki transkrypcyjne ARF TAS4 tasirnas czynniki transkrypcyjne MYB Northern Mutacje rdr6 i zip, podobnie jak dcl4 i tas3, przyspieszają zmiany rozwojowe przyspieszone przejście z fazy juwenilnej do dorosłej: wydłużone liście, wywinięte pod spód brzegi blaszek liściowych Fahlgren, N., Montgomery, T.A., Howell, M.D., Allen, E., Dvorak, S.K., Alexander, A.L., and Carrington, J.C. (2006) Curr. Biol
18 natsirna: roślinne endogenne sirna natsirna natural antisense-transcript derived sirna cis-acting pochodzą z zachodzących na siebie transkryptów genów związanych ze stresem zachodzące na siebie sekwencje sąsiadujących genów wyciszenie komplementarne względem siebie transkrypty NAT (natural antisense) tworzą dsrna AGO AGO Wg Katiyar-Agarwal, S., Morgan, R., Dahlbeck, D., Borsani, O., Villegas Jr. A., Zhu, J.-K., Staskawicz, B.J., and Jin, H. (2006) Proc. Natl. Acad. Sci. USA 35 natsirna: roślinne endogenne sirna syntetyzowane w odpowiedzi na stres np. wysokie zasolenie gleby biogeneza: DCL2 i/lub DCL1, RDR6 (polimeraza RNA zależna od RNA), SGS3 (białko wiążące RNA) i polimeraza RNA IV 24 nt natsirna cięcie jednej z cząsteczek mrna, druga służy jako matryca do syntezy 2-rzędowych 21nt natsirna przez RDR6 i DCL1 A. thaliana: lsirna (long sirna) 30-40nt również powstają z transkryptów NAT inne białka uczestniczą w biogenezie 36 Ghildiyal & Zamore (2009) Nat Rev Genet 18
19 Transkrypcyjne wyciszanie genów (TGS): hc-sirna małe RNA mogą hamować transkrypcję określonych genów poprzez kowalencyjne modyfikacje DNA lub białek histonowych białka histonowe transkrypcja Ten rodzaj wyciszenia jest często związany ze stale nieaktywnym transkrypcyjnie DNA, włączając rejony centromerowe i transpozony, ale również zachodzi w genach. DNA wyciszenie 37 Mechanizmy epigenetyczne kontroli ekspresji genów metylacja DNA potranslacyjne modyfikacje histonów przebudowa chromatyny zależna od ATP warianty histonów ncrna histony chromatyna DNA Dulac C. (2010) Nature 38 19
20 Co oznacza epigenetyka? Epigenetyczna regulacja ekspresji genów = zmiana ekspresji genu, która zachodzi bez zmiany sekwencji DNA Dosłownie, słowo epigenetyka oznacza powyżej genetyki Transkrypcja Jest to informacja pozagenowa, nie dotycząca samej sekwencji, ale kowalencyjnych modyfikacji DNA i zmian struktury chromatyny wyciszanie epigenetyczne komórki (organizmy) identyczne genetycznie Różne epigenetyczne modyfikacje prowadzą do różnych wzorów ekspresji genów różne fenotypy W praktyce, epigenetyka opisuje zjawiska, dzięki którym w identycznych genetycznie komórkach lub organizmach dochodzi do różnego sposobu ekspresji genów czego efektem są różnice fenotypowe. 39 sirna mogą wyciszać DNA za pośrednictwem enzymów metylujących cytozyny lub modyfikujących białka histonowe O N NH2 N ~ cytozyna O N NH2 N ~ CH3 5-metylocytozyna DNA może być kowalencyjnie modyfikowany w reakcji metylacji cytozyny W mechanizm transkrypcyjnego wyciszania DNA przez sirna zaangażowane są dwie specyficzne dla roślin polimerazy RNA: Pol IV i Pol V metylotransferaza DNA modyfikacja histonów metylacja DNA 40 20
21 Roślinne polimerazy RNA RNA DNA polimeraza RNA enzym występowanie funkcja Polimeraza RNA I wszystkie Eucaryota synteza rrna Polimeraza RNA II wszystkie Eucaryota synteza mrna, microrna Polimeraza RNA III wszystkie Eucaryota synteza trna, 5S rrna Polimeraza RNA IV rośliny biogeneza sirna Polimeraza RNA V rośliny rekrutacja białek AGO do DNA 41 Polimerazy RNA IV i V uczestniczą w transkrypcyjnym wyciszaniu genów Model mechanizmu metylacji DNA zależnej od RNA (RdDM- RNA-directed DNA methylation) chromatin remodelling GW/WG chromatin remodelling DDR elongation factor dsrna-binding protein Polimeraza RNA IV uczestniczy w biogenezie sirna. Niekodujące transkrypty polimerazy RNA V nakierowują maszynerię wyciszającą do odpowiednich sekwencji DNA
22 Metylacja DNA NH2 N N O ~ cytozyna NH2 CH3 N N O ~ 5-metylocytozyna DNA może być kowalencyjnie modyfikowany w reakcji metylacji cytozyny: wyst. u ssaków i roslin, ale nie u niższych zwierzat czy drożdży 5-metylocytozyna TTCGCCGACTAA Rola metylacji DNA: imprinting, inaktywacja chromosomu X, rozwój embrionalny, represja sekwencji powtórzonych i transpozonów 43 Metylotransferazy DNA (DNMT) metylotransferaza DNA 1 de novo: metylotransferaza DNA 3A metylotransferaza DNA 3B MET1 (METHYLTRANSFERASE1) 5'-CG-3 i 5 -CNG-3 wyciszanie transpozonów, powtórzeń DNA, piętnowanie niektórych genów CMT3 (CHROMOMETHYLASE3) 5'-CHG-3' (H= A, C lub T) specyficzna dla roślin, może być rekrutowana przez metylotransferazę histonową SUVH4 (KYP), a więc odpowiadać na modyfikację białek histonowych DRM1/ DRM2 (DOMAINS REARRANGED 1/2) 5'-CHH-3' metylacja de novo DRM2 metylacja powtórzeń DNA wyciszanych przez sirna 44 22
23 Głównym celem metylacji cytozyn jest wyciszenie transpozonów i powtórzeń DNA Nie wszystkie transpozony są wyciszane na drodze mechanizmu RNAi GENY A. thaliana TRANSPOZONY demetylaza H3K9 KYP (SUVH4) H3K9 metylotransferaza DDM1 ATPaza z rodziny SNF2; przebudowa chromatyny Do zmetylowanego DNA przyłączają się białka z domeną MBD (methyl-cpg binding domain), do których z kolei wiąże się kompleks deacetylazy histonowej, co prowadzi do kondensacji chromatyny i represji ekspresji genów; mogą się również przyłączać metylotransferazy histonowe 45 Texeira & Colot (2009) EMBO J. Modyfikacje białek histonowych wpływają na zmiany struktury chromatyny DNA oktamer histonowy NUKLEOSOM Końce N białek histonowych (tzw. ogony histonowe) wystają poza nukleosom, są dostępne dla enzymów modyfikujących 46 23
24 Modyfikacje białek histonowych wpływają na zmiany struktury chromatyny DNA oktamer histonowy Modyfikacje histonów: (kod histonowy) acetylacja (Ac) metylacja (Me) fosforylacja (P) ubikwitynacja (Ub) sumoilacja (Su) W zalezności od miejsca w/w modyfikacji mogą one przyczyniać się do aktywacji lub inaktywacji transkrypcji 47 Known post-translational modifications and the amino acid residues they modify 48 Latham J. A., Dent S. Y. R. (2007) Nat. Struct. Mol. Biol. 24
25 Modyfikacje białek histonowych wpływają na zmiany struktury chromatyny S/T kinazy fosfatazy -P S/T fosforylacja K acetylotransferazy histonowe (HAT) deacetylazy histonowe (HDAC) -COCH3 K acetylacja K/R metylotransferazy demetylazy -CH3 K/R metylacja 49 Acetylacja lizyny białek histonowych seria acetylo-lizyn (bardziej obojętne niż dodatnio naładowane lizyny) na ogonach histonów osłabia interakcje elektrostatyczne miedzy histonami a DNA, co pozwala na rozluźnienie struktury chromatyny inne funkcje: regulacja naprawy DNA poprzez przebudowę chromatyny; acetylowane lizyny są wtedy miejscami przyłączenia dla białek przebudowujących chromatynę (poprzez bromodomenę) + NH3 lizyna (K) N CH3 O acetylowana lizyna (KAc) za acetylację odpowiedzialne sa acetylotransferazy (HAT), a donorem grupy acetylowej jest acetylo-coa acetylacja jest odwracalna; deacetylację przeprowadzaja deacetylazy (HDAC) K acetylotransferazy histonowe (HAT) deacetylazy histonowe (HDAC) -COCH3 K 50 25
26 Metylacja białek histonowych metylacja lizyny powoduje zwiekszenie hydrofobowego i kationowego charakteru tej reszty aminokwasowej; w zależnosci od enzymu lizyna może byc mono, di lub trimetylowana. metylowana lizyna Mono (Kme1) Di (Kme2) + NH2 CH3 + NH CH3 CH3 za metylację odpowiedzialne są metylotransferazy (HMT) zawierające domenę SET, a donorem grupy metylowej jest S-adenozylometionina (SAM) lub S- adenozylohomocysteina (AdoHcy); metylacja H3K79 katalizowana jest przez Dot1 spoza rodziny Set Tri (Kme3) + N CH3 CH3 CH3 usuniecie grup metylowych: wymiana nukleosomów, modyfikacje chemiczne zmetylowanych reszt lub enzymatyczna demetylacja K/R metylotransferazy demetylazy -CH3 K/R 51 Fosforylacja białek histonowych Fosforylacje seryn H3S10 i H3S28 niezbedne są do kondensacji chromosomów i prawidłowej mitozy; także pozytywna rola w aktywacji transkrypcji: hamuje metylację H3K9 i promuje acetylację lizyn położonych w jej sąsiedztwie Zidentyfikowano ufosforylowane seryny na wszystkich histonach (fosforylacja wariantu H2A niezbedna jest w aktywacji naprawy DNA i regulacji cyklu komórkowego po uszkodzeniu DNA) S/T kinazy fosfatazy -P S/T 52 26
27 Przykład modyfikacje H3 Me Me P Ac Me Ac Ac Me Me P H3 A R T K Q T A R K S T G G K A P R K Q L A T K A A R K S Koniec N histonu H3 jest często modyfikowany (w jednym lub kilku miejscach), co przyczynia się do aktywacji lub inhibicji transkrypcji. Modyfikacje białek histonowych zmieniają strukturę chromatyny H3 Me P Ac K4 S10 K14 H3 Me K9 Me P K27 S28 53 Inne modyfikacje białek histonowych występują w sekwencjach genów kodujących białka, inne w sekwencjach transpozonowych Analiza typu ChIP-chip gen mrna H3K4me metylacja H3K4 występuje w genach aktywnie transkrybowanych H3K9me Me-C transpozon metylacja H3K9 jest związana z metylowanym DNA (Me-C) i transpozonami czerwony = silna korelacja zielony = słaba korelacja Lippman, Z., Gendrel, A.-V., Black, M., Vaughn, M.W., Dedhia, N., McCombie, W.R., Lavine, K., Mittal, V., May, B., Kasschau, K.D., Carrington, J.C.,Doerge, R.W., Colot, V., Martienssen, R. (2004) Nature 54 27
28 H3K27me3: w genach kodujących białka Analiza typu ChIP-chip niebieski = geny czerwony = powtórzenia DNA H3K27me3 u A. thaliana występuje w rejonach bogatych w sekwencje kodujące zielony = H3K27me3 fioletowy = metylocytozyna 55 Zhang, X., Clarenz, O., Cokus, S., Bernatavichute, Y.V., Pellegrini, M., Goodrich, J., Jacobsen, S.E. (2007) PLoS Biol. Mechanizm działania modyfikacji potranslacyjnych białek histonowych Działanie bezpośrednie: zmiany w oddziaływaniach histon-dna i histon-histon Działanie pośrednie: rekrutacja białek rozpoznających określone modyfikacje histonów Kozaurides
29 Przebudowa chromatyny zależna od ATP (chromatin remodelling) metylacja DNA potranslacyjne modyfikacje histonów przebudowa chromatyny zależna od ATP warianty histonów ncrna histony chromatyna DNA Aktywność kompleksów przebudowujących chromatynę zależy od ATP, w wyniku ich działania zmienia się sposób oddziaływania histon-dna. Kompleksy remodelujące zaangażowane są zarówno w aktywację, jak i represję transkrypcji. Dulac C. (2010) Nature 57 Przebudowa chromatyny: przesunięcie oktameru histonowego usunięcie oktameru histonowego odsłonięcie DNA rozwinięcie nici DNA zamiana dimeru H2A-H2B na H2A.Z-H2B (Htz1 u S. cerevisiae) usunięcie dimerów H2A-H2B zmiana składu oktameru histonowego Aktywność kompleksów przebudowujących chromatynę zależy od ATP, w wyniku ich działania zmienia się sposób oddziaływania histon-dna. Kompleksy remodelujące zaangażowane są zarówno w aktywację, jak i represję transkrypcji. 58 Clapier C. R., Cairns B. R Annu Rev Biochem 29
30 Wyróżniamy cztery rodziny kompleksów odpowiedzialnych za przebudowę chromatyny: SWI2: zawieraja bromodomenę, wszystkie rodzaje przebudowy chromatyny ISWI: przesuwanie nukleosomów CHD: zawieraja chromodomene, regulacja transkrypcji INO80/SWR: wymiana histonów 59 Clapier C. R., Cairns B. R Annu Rev Biochem Kompleksy przebudowujące chromatynę składają się z wielu białkowych podjednostek Drożdżowy SWI/SNF: 11 białek potrzebny do ekspresji genów decydujących o typie koniugacyjnym drożdży (switching), oraz ekspresji genów regulujących metabolizm sacharozy (sucrose non-fermenting) 60 30
31 DDM1 (decrease in DNA methylation1) : ATPaza z rodziny SWI/SNF u A. thaliana DDM1 jest specyficznie zaangażowana w metylację transpozonów GENY A. thaliana TRANSPOZONY demetylacja H3K9 KYP (SUVH4) H3K9 metylotransferaza DDM1 ATPaza z rodziny SNF2; remodeling chromatyny DDM1 jest kluczowym czynnikiem łączącym przebudowę chromatyny i wprowadzanie/utrzymywanie metylacji DNA 61 Texeira & Colot (2009) EMBO J. Warianty histonów metylacja DNA potranslacyjne modyfikacje histonów przebudowa chromatyny zależna od ATP warianty histonów ncrna histony chromatyna DNA Dulac C. (2010) Nature Za wymianę różnych wariantów histonów w oktamerze histonowym odpowiadają kompleksy przebudowujące chromatynę z rodziny INO80/SWR 62 31
32 Warianty histonów: X-inactivation Talbert & Henikoff (2010) Nat Rev Mol Cell Biol 63 Histon H2A.Z: aktywacja transkrypcji gen nieaktywny gen aktywny kompleks SWR1/ SRCAP Obecność odmiany histonu H2 - H2A.Z sprzyja transkrypcji. Kompleks SWR1/SRCAP zamienia histon H2A na H2A.Z 64 32
33 Histon CENH3: centromery H3 CENH3 nukleosomy w centromerach zawierają wariant histonu H3: CENH3 (CENP-A u zwierząt) centromery zawierające CENH3 są otoczone obszarem bogatym w metylacje H3K9me2 (heterochromatyna przycentromerowa) Jiang, J., Birchler, J.A., Parrott, W.A., and Dawe, R.K. (2003) Trends Plant Sci. Elsevier. Zhang, W., Lee, H.-R., Koo, D.-H., and Jiang, J. (2008) Plant Cell 65 Epigenetyczna regulacja ekspresji genów przez ncrna metylacja DNA potranslacyjne modyfikacje histonów przebudowa chromatyny zależna od ATP warianty histonów ncrna histony chromatyna DNA RNA jest jedynym jak dotąd poznanym czynnikiem inicjującym dziedziczenie epigenetyczne i odróżniającym sekwencje, które mają zostać wyciszone lub aktywowane Dulac C. (2010) Nature 66 33
34 Epigenetyczna regulacja ekspresji genów przez lncrna u ssaków niektóre lncrna (transkrypty polimerazy RNA II) rekrutują kompleksy wyciszające transkrypcję do odpowiednich rejonów genomu, zarówno in cis, jak in trans PcG- Polycomb group proteins G9a- metylotransferazy histonowe /H3K9/H3K27 XIST inaktywacja chromosomu X u ssaków (dosage compensation: wyrównanie poziomu ekspresji genów chromosomów X) 67 Chen & Carmichael (2010) Wiley Interdisciplinary Reviews: RNA Inaktywacja chromosomu X: wyciszanie epigenetyczne Mus musculus: u samic od wczesnego etapu rozwoju zarodkowego na aktywnym chromosomie X ekspresja Tsix, na nieaktywnym ekspresja Xist John E. Froberg, Lin Yang, Jeannie T. Lee (2013) Journal of Molecular Biology 17kb (u myszy; ludzki 19kb) ncrna XIST opłaszcza chromosom X tzw. ciałko Barra: skondensowana forma chromosomu X, głównie w postaci heterochromatyny 68 34
35 Inaktywacja chromosomu X: wyciszanie epigenetyczne RepA: 1,6kb ncrna zawierający sekwencje rejonu 5 transkryptu XIST bezpośrednio wiąże kompleks PRC2 John E. Froberg, Lin Yang, Jeannie T. Lee (2013) Journal of Molecular Biology 69 Inaktywacja chromosomu X: wyciszanie epigenetyczne XIST ncrna uruchamia zmiany epigenetyczne, które zapewniają pamięć komórkową stanu nieaktywnego: zamiana histonu H2A na makroh2a metylacja histonu H3: H3K9 H3K27 deacetylacja histonu H4 (?) metylacja DNA /już po inaktywacji chromosomu Ferrari F., Alekseyenko A.A., Park P.J., Kuroda M.I. (2013) Nat. Struct. Mol. Biol
36 Epigenetyczna regulacja ekspresji genów przez ncrna kompensacja dawki chromosomów płciowych (dosage compensation) u Drosophila melanogaster rox rox1/rox2 ncrna inicjują modyfikacje histonów u samców Drosophila zwiększenie aktywności chromosomu X acetylacja histonów demetylacja H3K9 Ferrari F., Alekseyenko A.A., Park P.J., Kuroda M.I. (2013) Nat. Struct. Mol. Biol. 71 sirna - podsumowanie Szlak sirna wycisza egzogenny, obcy DNA, transpozony oraz sekwencje powtórzone sirna powstają dzięki aktywności białek typu Dicer, które tną dsrna sirna są wiązane przez białka z rodziny AGO i tworzą kompleksy wyciszające (RISC) Kompleksy wyciszające mogą działać potranskrypcyjnie, poprzez cięcie mrna lub inhibicję translacji Kompleksy wyciszające mogą zmieniać strukturę chromatyny poprzez metylację DNA lub modyfikację białek histonowych 72 36
37 microrna - mirna uważa się, że mirna wyewoluowały z sirna powstają i dojrzewają w podobny (do pewnego stopnia) sposób mirna są kodowane przez specyficzne geny MIR, ale wpływają na ekspresję innych genów są cząsteczkami regulatorowymi działającymi in trans mirna regulują procesy rozwojowe i fizjologiczne 73 microrna - mirna microrna działaja poprzez cięcie mrna lub inhibicję translacji gen MIR Inhibicja translacji AGO AAAn RNA Pol II cięcie mrna AGO AGO AAAn mrna AAAn RNA Pol II 74 37
38 microrna - mirna aktywna translacja inhibicja inicjacji translacji degradacja mrna inhibicja translacji 75 Stefani G., Slack F. J., (2008) Mol Cell Biol microrna - mirna aktywna translacja degradacja mrna 76 Stefani G., Slack F. J., (2008) Mol Cell Biol 38
39 Destabilization of target mrna is the predominant reason for reduced protein output. Step 1. Initial effect of mirnas: inhibition of translation at the initiation step without mrna decay. Step 2. mrna deadenylation by PAN2 PAN3 and CCR4 NOT complexes recruited by mirisc as a consequence of translation inhibition that makes poly(a) tail more accessible. Step3. Stimulated deadenylation potentiates the effect on translational inhibition and leads to decay of target mrnas through the recruitment of the decapping machinery. 77 Geny MIR: transkrypcja długich cząsteczek pri-mirna z których powstają mirna mirna są kodowane przez geny MIR pierwotne transkrypty mirna (pri-mirna) tworzą drugorzędowe, dwuniciowe struktury, które są rozpoznawane i cięte przez białka Dicer (u roślin DCL1) nić mirna* jest degradowana MIR gene 5' pri-mirna 3' 5' mirna 3' mirna* mirna mrna target 78 39
40 Biogeneza mirna u roślin transkrypcja DNA pri-mirna HYL1: oddziaływanie z DCL1 cięcie mirna mirna* HEN1: metylotransferaza, metylacja mirna/mirna* HST (HASTY): eksport mirna/mirna* do cytoplazmy, homolog eksportyny 5 metylacja eksport tworzenie kompleksu RISC jądro komórkowe cytoplazma mrna cięcie mrna 79 Mallory A., Vaucheret H. (2006) Nature Genet. Biogeneza mirna u zwierząt DNA transkrypcja pri-mirna Drosha+DGCR8 (Pasha): cięcie pri-mirna pre-mirna jądro komórkowe cięcie pre-mirna EXP5 (eksportyna 5): eksport pre-mirna do cytoplazmy cytoplazma eksport pre-mirna Dicer: cięcie pre-mirna mirna/mirna* cięcie mirna/mirna* tworzenie kompleksu RISC 80 Wg: Ding X.C., Weiler J., Grosshans H. (2009) Trends in Biotechnology 40
41 Biogeneza mirna u roślin i zwierząt eksport z jądra komórkowego do cytoplazmy pri-mirna pri-mirna MIRTRONY 81 Carthew R. W., Sontheimer E. J., (2009) Cell Mirtrony występują u D. melanogaster, C. elegans, ssaków lariat powstaja z intronów wyciętych z pre-mrna podczas składania mrna (splicing) niezależne od Drosha rozcięcie struktury lariatu (debranching) prowadzi do powstania pre-mirna pre-mirna biogeneza mirna 82 Kim V. N., Han J., Siomi M.C. (2009) Mol Cell Biol 41
42 mirna - podsumowanie Uważa się, że mirna regulują większość procesów biologicznych zarówno u roślin jak i zwierząt, kontrolując ekspresję specyficznych genów działając in trans mirna regulują procesy rozwojowe: np. u A. thaliana mir156 i mir172 regulują procesy kwitnienia; u C. elegans wyciszenie lin-14 przez lin-4 jest potrzebne do prawidłowego rozwoju mirna działają głównie poprzez obniżenie poziomu mrna, ale również poprzez inhibicję translacji 83 Małe RNA niezależne od Dicer: pirna ~25-30 nt, 2 -O-metylowane końce 3 występują u zwierząt, zidentyfikowane w liniach komórek rozrodczych D. melanogaster wiążą się z białkami PIWI: Piwi, Aubergine, Ago3 D. melanogaster MILI, MIWI, MIWI2 mysie HILI, HIWI1, HIWI2 ludzkie C. elegans 21U RNA 21 nt wiąże PRG-1 (Piwi-related gene 1) metylacja końca 3 wyciszanie transpozonów i powtórzeń DNA, u ssaków metylacja DNA sekwencji transpozonowych 84 Ghildiyal & Zamore (2009) Nat Rev Genet 42
43 85 Ghildiyal & Zamore (2009) Nat Rev Genet Małe RNA niezależne od Dicer prirna primal small RNAs zidentyfikowane u S.pombe tworzenie/utrzymywanie heterochromatyny w rejonach centromerowych Triman: 3-5 egzorybonukleaza obróbka prekursorów prirna i sirna 86 43
44 Podsumowanie Wyciszanie genów za pośrednictwem małych RNA odgrywa istotną rolę zarówno w regulacji ekspresji, jak i w zachowaniu integralności genomów organizmów żywych. Specyficzność wyciszenia określonych genów zapewniona jest dzięki komplementarności zasad między małym wyciszającym RNA, a RNA ulegającym wyciszeniu. Za pośrednictwem sirna wyciszane są m.in. regiony bogatej w powtórzenia DNA heterochromatyny, sekwencje transpozonowe, wirusy i inne patogeny. mirna i tasirna regulują m.in. ekspresję genów związanych z zegarem biologicznym, rozwojem, reakcją na stres
Świat małych RNA. Monika Zakrzewska-Płaczek
 Świat małych RNA Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas) srna 21-30 nt wyciszanie ekspresji genów: (gene silencing, RNA silencing) post-transkrypcyjne wyciszanie genów
Świat małych RNA Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas) srna 21-30 nt wyciszanie ekspresji genów: (gene silencing, RNA silencing) post-transkrypcyjne wyciszanie genów
Endo-siRNA: 32 Ghildiyal & Zamore (2009) Nat Rev Genet
 Endo-siRNA sirna: 32 Ghildiyal & Zamore (2009) Nat Rev Genet Większość endogennych sirna powstaje z transpozonów i powtórzeń sekwencji DNA Chromosom Centromer małe RNA transpozony retrotranspozony Wysoko-przepustowe
Endo-siRNA sirna: 32 Ghildiyal & Zamore (2009) Nat Rev Genet Większość endogennych sirna powstaje z transpozonów i powtórzeń sekwencji DNA Chromosom Centromer małe RNA transpozony retrotranspozony Wysoko-przepustowe
Małe RNA.
 Małe RNA Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas) srna 21-30 nt wyciszanie ekspresji genów: (gene silencing, RNA silencing) post-transkrypcyjne wyciszanie genów (post-transcriptional
Małe RNA Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas) srna 21-30 nt wyciszanie ekspresji genów: (gene silencing, RNA silencing) post-transkrypcyjne wyciszanie genów (post-transcriptional
Rola chromatyny w regulacji ekspresji genów. Monika Zakrzewska-Płaczek
 Rola chromatyny w regulacji ekspresji genów Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Co oznacza epigenetyka? Epigenetyczna regulacja ekspresji genów = zmiana ekspresji genu, która zachodzi bez zmiany
Rola chromatyny w regulacji ekspresji genów Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Co oznacza epigenetyka? Epigenetyczna regulacja ekspresji genów = zmiana ekspresji genu, która zachodzi bez zmiany
Rola chromatyny w regulacji ekspresji genów. Monika Zakrzewska-Płaczek
 Rola chromatyny w regulacji ekspresji genów Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Epigenetyczna etyczna regulacja ekspresji genów zmiana ekspresji genu, która zachodzi bez zmiany sekwencji DNA
Rola chromatyny w regulacji ekspresji genów Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Epigenetyczna etyczna regulacja ekspresji genów zmiana ekspresji genu, która zachodzi bez zmiany sekwencji DNA
Małe RNA i mechanizmy epigenetycznej regulacji ekspresji genów. dr Monika Zakrzewska-Płaczek
 Małe RNA i mechanizmy epigenetycznej regulacji ekspresji genów dr Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas, smrnas) srna 21-30 nt wyciszanie ekspresji genów: Specyficzność
Małe RNA i mechanizmy epigenetycznej regulacji ekspresji genów dr Monika Zakrzewska-Płaczek monika.z@ibb.waw.pl Małe RNA (small RNAs, srnas, smrnas) srna 21-30 nt wyciszanie ekspresji genów: Specyficzność
TRANSLACJA II etap ekspresji genów
 TRANSLACJA II etap ekspresji genów Tłumaczenie informacji genetycznej zawartej w mrna (po transkrypcji z DNA) na aminokwasy budujące konkretne białko. trna Operon (wg. Jacob i Monod) Zgrupowane w jednym
TRANSLACJA II etap ekspresji genów Tłumaczenie informacji genetycznej zawartej w mrna (po transkrypcji z DNA) na aminokwasy budujące konkretne białko. trna Operon (wg. Jacob i Monod) Zgrupowane w jednym
Wykład 5. Remodeling chromatyny
 Wykład 5 Remodeling chromatyny 1 Plan wykładu: 1. Przebudowa chromatyny 2. Struktura, funkcje oraz mechanizm działania kompleksów remodelujących chromatynę 3. Charakterystyka kompleksów typu SWI/SNF 4.
Wykład 5 Remodeling chromatyny 1 Plan wykładu: 1. Przebudowa chromatyny 2. Struktura, funkcje oraz mechanizm działania kompleksów remodelujących chromatynę 3. Charakterystyka kompleksów typu SWI/SNF 4.
Wykład 3. Organizacja jądra komórkowego struktura chromatyny
 Wykład 3 Organizacja jądra komórkowego struktura chromatyny Struktura jądra komórkowego Matriks jądrowa - jądro komórkowe pozbawione chromatyny Nukleoplazma Heterochromatyna Euchromatyna Jąderko Otoczka
Wykład 3 Organizacja jądra komórkowego struktura chromatyny Struktura jądra komórkowego Matriks jądrowa - jądro komórkowe pozbawione chromatyny Nukleoplazma Heterochromatyna Euchromatyna Jąderko Otoczka
INICJACJA ELONGACJA TERMINACJA
 INICJACJA ELONGACJA TERMINACJA 2007 by National Academy of Sciences Kornberg R D PNAS 2007;104:12955-12961 Struktura chromatyny pozwala na różny sposób odczytania informacji zawartej w DNA. Możliwe staje
INICJACJA ELONGACJA TERMINACJA 2007 by National Academy of Sciences Kornberg R D PNAS 2007;104:12955-12961 Struktura chromatyny pozwala na różny sposób odczytania informacji zawartej w DNA. Możliwe staje
Dr. habil. Anna Salek International Bio-Consulting 1 Germany
 1 2 3 Drożdże są najprostszymi Eukariontami 4 Eucaryota Procaryota 5 6 Informacja genetyczna dla każdej komórki drożdży jest identyczna A zatem każda komórka koduje w DNA wszystkie swoje substancje 7 Przy
1 2 3 Drożdże są najprostszymi Eukariontami 4 Eucaryota Procaryota 5 6 Informacja genetyczna dla każdej komórki drożdży jest identyczna A zatem każda komórka koduje w DNA wszystkie swoje substancje 7 Przy
wykład dla studentów II roku biotechnologii Andrzej Wierzbicki
 Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ 1. Gen to odcinek DNA odpowiedzialny
Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ 1. Gen to odcinek DNA odpowiedzialny
ODKRYCIE POLIMERAZY RNA IV
 ODKRYCIE POLIMERAZY RNA IV Ewa Róg-ZieliR Zielińska POLIMERAZY RNA U eukariota występuj pują trzy podstawowe rodzaje polimeraz RNA zależnych od DNA Wszystkie te polimerazy są złożone one z około o 12-17
ODKRYCIE POLIMERAZY RNA IV Ewa Róg-ZieliR Zielińska POLIMERAZY RNA U eukariota występuj pują trzy podstawowe rodzaje polimeraz RNA zależnych od DNA Wszystkie te polimerazy są złożone one z około o 12-17
Epigenetyczna regulacja ekspresji genów w trakcie rozwoju zwierząt i roślin
 Epigenetyczna regulacja ekspresji genów w trakcie rozwoju zwierząt i roślin Rozwój jest z natury epigenetyczny te same geny w różnych tkankach i komórkach utrzymywane są w stanie aktywnym lub wyciszonym
Epigenetyczna regulacja ekspresji genów w trakcie rozwoju zwierząt i roślin Rozwój jest z natury epigenetyczny te same geny w różnych tkankach i komórkach utrzymywane są w stanie aktywnym lub wyciszonym
Chromatyna struktura i funkcja
 Chromatyna struktura i funkcja dr hab. Marta Koblowska dr Rafał Archacki http://www.accessexcellence.org/ab/gg/nucleosome.html GENETYKA FRIEDRICH MIESCHER (1844-1895) W 1869 roku wyizolował z jąder komórkowych
Chromatyna struktura i funkcja dr hab. Marta Koblowska dr Rafał Archacki http://www.accessexcellence.org/ab/gg/nucleosome.html GENETYKA FRIEDRICH MIESCHER (1844-1895) W 1869 roku wyizolował z jąder komórkowych
Badanie funkcji genu
 Badanie funkcji genu Funkcję genu można zbadać różnymi sposobami Przypadkowa analizy funkcji genu MUTACJA FENOTYP GEN Strategia ukierunkowanej analizy funkcji genu GEN 1. wprowadzenie mutacji w genie 2.
Badanie funkcji genu Funkcję genu można zbadać różnymi sposobami Przypadkowa analizy funkcji genu MUTACJA FENOTYP GEN Strategia ukierunkowanej analizy funkcji genu GEN 1. wprowadzenie mutacji w genie 2.
wykład dla studentów II roku biotechnologii Andrzej Wierzbicki
 Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Wykład 5 Droga od genu do
Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Wykład 5 Droga od genu do
Badanie funkcji genu
 Badanie funkcji genu Funkcję genu można zbadać różnymi sposobami Przypadkowa analizy funkcji genu MUTACJA FENOTYP GEN Strategia ukierunkowanej analizy funkcji genu GEN 1. wprowadzenie mutacji w genie 2.
Badanie funkcji genu Funkcję genu można zbadać różnymi sposobami Przypadkowa analizy funkcji genu MUTACJA FENOTYP GEN Strategia ukierunkowanej analizy funkcji genu GEN 1. wprowadzenie mutacji w genie 2.
Arabidopsis thaliana jako organizm modelowy
 Arabidopsis thaliana jako organizm modelowy Arabidopsis thaliana mały jednoroczny chwast z rodziny Brassicaceae (krzyżowe) odkryty w XVI w przez Johannesa Thala thaliana w 1873 zidentyfikowano pierwszego
Arabidopsis thaliana jako organizm modelowy Arabidopsis thaliana mały jednoroczny chwast z rodziny Brassicaceae (krzyżowe) odkryty w XVI w przez Johannesa Thala thaliana w 1873 zidentyfikowano pierwszego
wykład dla studentów II roku biotechnologii Andrzej Wierzbicki
 Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Budowa rybosomu Translacja
Genetyka ogólna wykład dla studentów II roku biotechnologii Andrzej Wierzbicki Uniwersytet Warszawski Wydział Biologii andw@ibb.waw.pl http://arete.ibb.waw.pl/private/genetyka/ Budowa rybosomu Translacja
Regulacja ekspresji genów. Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego.
 Regulacja ekspresji genów Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Problem: jak sprawić aby z jednej komórki powstał wielokomórkowy
Regulacja ekspresji genów Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Problem: jak sprawić aby z jednej komórki powstał wielokomórkowy
Budowa histonów rdzeniowych
 Histony rdzeniowe Budowa histonów rdzeniowych Histon H4 Silnie dodatnio naładowany N-koniec białka Globularna hydrofobowa domena odpowiedzialna za oddziaływania histon-histon oraz histon-dna Domena histonowa
Histony rdzeniowe Budowa histonów rdzeniowych Histon H4 Silnie dodatnio naładowany N-koniec białka Globularna hydrofobowa domena odpowiedzialna za oddziaływania histon-histon oraz histon-dna Domena histonowa
Wykład 14 Biosynteza białek
 BIOCHEMIA Kierunek: Technologia Żywności i Żywienie Człowieka semestr III Wykład 14 Biosynteza białek WYDZIAŁ NAUK O ŻYWNOŚCI I RYBACTWA CENTRUM BIOIMMOBILIZACJI I INNOWACYJNYCH MATERIAŁÓW OPAKOWANIOWYCH
BIOCHEMIA Kierunek: Technologia Żywności i Żywienie Człowieka semestr III Wykład 14 Biosynteza białek WYDZIAŁ NAUK O ŻYWNOŚCI I RYBACTWA CENTRUM BIOIMMOBILIZACJI I INNOWACYJNYCH MATERIAŁÓW OPAKOWANIOWYCH
Regulacja transkrypcji genów eukariotycznych
 Regulacja transkrypcji genów eukariotycznych Definicja genu Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko lub RNA. Zawiera całą funkcjonalną podjednostkę wraz
Regulacja transkrypcji genów eukariotycznych Definicja genu Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko lub RNA. Zawiera całą funkcjonalną podjednostkę wraz
The Role of Maf1 Protein in trna Processing and Stabilization / Rola białka Maf1 w dojrzewaniu i kontroli stabilności trna
 Streszczenie rozprawy doktorskiej pt. The Role of Maf1 Protein in trna Processing and Stabilization / Rola białka Maf1 w dojrzewaniu i kontroli stabilności trna mgr Tomasz Turowski, promotor prof. dr hab.
Streszczenie rozprawy doktorskiej pt. The Role of Maf1 Protein in trna Processing and Stabilization / Rola białka Maf1 w dojrzewaniu i kontroli stabilności trna mgr Tomasz Turowski, promotor prof. dr hab.
października 2013: Elementarz biologii molekularnej. Wykład nr 2 BIOINFORMATYKA rok II
 10 października 2013: Elementarz biologii molekularnej www.bioalgorithms.info Wykład nr 2 BIOINFORMATYKA rok II Komórka: strukturalna i funkcjonalne jednostka organizmu żywego Jądro komórkowe: chroniona
10 października 2013: Elementarz biologii molekularnej www.bioalgorithms.info Wykład nr 2 BIOINFORMATYKA rok II Komórka: strukturalna i funkcjonalne jednostka organizmu żywego Jądro komórkowe: chroniona
Komórka eukariotyczna
 Komórka eukariotyczna http://pl.wikipedia.org/w/index.php?title=plik:hela_cells_stained_with_hoechst_33258.jpg cytoplazma + jądro komórkowe = protoplazma W cytoplazmie odbywa się: cała przemiana materii,
Komórka eukariotyczna http://pl.wikipedia.org/w/index.php?title=plik:hela_cells_stained_with_hoechst_33258.jpg cytoplazma + jądro komórkowe = protoplazma W cytoplazmie odbywa się: cała przemiana materii,
Gen eukariotyczny. Działanie i regulacja etapy posttranskrypcyjne
 Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne Definicja genu Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko lub RNA. Zawiera całą funkcjonalną
Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne Definicja genu Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko lub RNA. Zawiera całą funkcjonalną
Gen eukariotyczny. Działanie i regulacja etapy posttranskrypcyjne
 Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne Literatura Allison, r. 13 Brown, r. 12.2 Definicja genu Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze
Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne Literatura Allison, r. 13 Brown, r. 12.2 Definicja genu Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze
Zmiany epigenetyczne a dieta
 GENO-centryczne teorie mają ograniczony zasięg Klasyczna genetyka nie może wytłumaczyć na przykład : Zmiany epigenetyczne a dieta 1. różnorodności fenotypowej w obrębie populacji; 2. dlaczego bliźniaki
GENO-centryczne teorie mają ograniczony zasięg Klasyczna genetyka nie może wytłumaczyć na przykład : Zmiany epigenetyczne a dieta 1. różnorodności fenotypowej w obrębie populacji; 2. dlaczego bliźniaki
TRANSKRYPCJA - I etap ekspresji genów
 Eksparesja genów TRANSKRYPCJA - I etap ekspresji genów Przepisywanie informacji genetycznej z makrocząsteczki DNA na mniejsze i bardziej funkcjonalne cząsteczki pre-mrna Polimeraza RNA ETAP I Inicjacja
Eksparesja genów TRANSKRYPCJA - I etap ekspresji genów Przepisywanie informacji genetycznej z makrocząsteczki DNA na mniejsze i bardziej funkcjonalne cząsteczki pre-mrna Polimeraza RNA ETAP I Inicjacja
TATA box. Enhancery. CGCG ekson intron ekson intron ekson CZĘŚĆ KODUJĄCA GENU TERMINATOR. Elementy regulatorowe
 Promotory genu Promotor bliski leży w odległości do 40 pz od miejsca startu transkrypcji, zawiera kasetę TATA. Kaseta TATA to silnie konserwowana sekwencja TATAAAA, występująca w większości promotorów
Promotory genu Promotor bliski leży w odległości do 40 pz od miejsca startu transkrypcji, zawiera kasetę TATA. Kaseta TATA to silnie konserwowana sekwencja TATAAAA, występująca w większości promotorów
Gen eukariotyczny. Działanie i regulacja etapy posttranskrypcyjne
 Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne Literatura Allison, r. 13 Brown, r. 12.2 Poziom RNA w komórce dojrzewanie/obróbka synteza degradacja Rys. dr Monika Zakrzewska-Płaczek,
Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne Literatura Allison, r. 13 Brown, r. 12.2 Poziom RNA w komórce dojrzewanie/obróbka synteza degradacja Rys. dr Monika Zakrzewska-Płaczek,
Gen eukariotyczny. Działanie i regulacja etapy posttranskrypcyjne
 Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne 1 Klasyczne wyobrażenie genu fragment DNA, który koduje funkcjonalny mrna Wszystkie transkrypty mrna mają niekodujące fragmenty 5 i 3 końcowe
Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne 1 Klasyczne wyobrażenie genu fragment DNA, który koduje funkcjonalny mrna Wszystkie transkrypty mrna mają niekodujące fragmenty 5 i 3 końcowe
Gen eukariotyczny. Działanie i regulacja etapy posttranskrypcyjne
 Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne 1 Definicja genu } Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko lub RNA. } Zawiera całą funkcjonalną
Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne 1 Definicja genu } Region DNA, który określa dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko lub RNA. } Zawiera całą funkcjonalną
Badanie funkcji genu
 Badanie funkcji genu Funkcję genu można zbadać różnymi sposobami Przypadkowa analizy funkcji genu MUTACJA FENOTYP GEN Strategia ukierunkowanej analizy funkcji genu GEN 1. wprowadzenie mutacji w genie 2.
Badanie funkcji genu Funkcję genu można zbadać różnymi sposobami Przypadkowa analizy funkcji genu MUTACJA FENOTYP GEN Strategia ukierunkowanej analizy funkcji genu GEN 1. wprowadzenie mutacji w genie 2.
WARUNKI ZALICZENIA PRZEDMIOTU- 5 ECTS
 WARUNKI ZALICZENIA PRZEDMIOTU- 5 ECTS KOLOKWIA; 15% KOLOKWIA-MIN; 21% WEJŚCIÓWKI; 6% WEJŚCIÓWKI-MIN; 5% EGZAMIN; 27% EGZAMIN-MIN; 26% WARUNKI ZALICZENIA PRZEDMIOTU- 5 ECTS kolokwium I 12% poprawa kolokwium
WARUNKI ZALICZENIA PRZEDMIOTU- 5 ECTS KOLOKWIA; 15% KOLOKWIA-MIN; 21% WEJŚCIÓWKI; 6% WEJŚCIÓWKI-MIN; 5% EGZAMIN; 27% EGZAMIN-MIN; 26% WARUNKI ZALICZENIA PRZEDMIOTU- 5 ECTS kolokwium I 12% poprawa kolokwium
Analizy wielkoskalowe w badaniach chromatyny
 Analizy wielkoskalowe w badaniach chromatyny Analizy wielkoskalowe wykorzystujące mikromacierze DNA Genotypowanie: zróżnicowane wewnątrz genów RNA Komórka eukariotyczna Ekspresja genów: Które geny? Poziom
Analizy wielkoskalowe w badaniach chromatyny Analizy wielkoskalowe wykorzystujące mikromacierze DNA Genotypowanie: zróżnicowane wewnątrz genów RNA Komórka eukariotyczna Ekspresja genów: Które geny? Poziom
Zgodnie z tzw. modelem interpunkcji trna, cząsteczki mt-trna wyznaczają miejsca
 Tytuł pracy: Autor: Promotor rozprawy: Recenzenci: Funkcje białek ELAC2 i SUV3 u ssaków i ryb Danio rerio. Praca doktorska wykonana w Instytucie Genetyki i Biotechnologii, Wydział Biologii UW Lien Brzeźniak
Tytuł pracy: Autor: Promotor rozprawy: Recenzenci: Funkcje białek ELAC2 i SUV3 u ssaków i ryb Danio rerio. Praca doktorska wykonana w Instytucie Genetyki i Biotechnologii, Wydział Biologii UW Lien Brzeźniak
Gen eukariotyczny. Działanie i regulacja etapy posttranskrypcyjne
 Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne 1 Geny eukariotyczne Procesy transkrypcji i translacji są rozdzielone w przestrzeni i czasie Każdy gen ma własny promotor, nie występują
Gen eukariotyczny Działanie i regulacja etapy posttranskrypcyjne 1 Geny eukariotyczne Procesy transkrypcji i translacji są rozdzielone w przestrzeni i czasie Każdy gen ma własny promotor, nie występują
Regulacja transkrypcji genów eukariotycznych
 Regulacja transkrypcji genów eukariotycznych Klasyczne wyobrażenie genu fragment DNA, który koduje mrna Definicja genu - klasyczna GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną
Regulacja transkrypcji genów eukariotycznych Klasyczne wyobrażenie genu fragment DNA, który koduje mrna Definicja genu - klasyczna GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną
Zmiany epigenetyczne a dieta
 Zmiany epigenetyczne a dieta Prof. dr hab. n. med. Małgorzata Milkiewicz Zmiany epigenetyczne a dieta 1 Chromatyna ulega kondensacji i dekondensacji podczas cyklu komórkowego stopień jej kondensacji wpływa
Zmiany epigenetyczne a dieta Prof. dr hab. n. med. Małgorzata Milkiewicz Zmiany epigenetyczne a dieta 1 Chromatyna ulega kondensacji i dekondensacji podczas cyklu komórkowego stopień jej kondensacji wpływa
Marek Figlerowicz 1, Agata Tyczewska 1, Magdalena Figlerowicz 2
 PRZEGL EPIDEMIOL 2006; 60: 693 700 Marek Figlerowicz 1, Agata Tyczewska 1, Magdalena Figlerowicz 2 Wpływ małych regulatorowych RNA na przebieg zakażeń wirusowych nowe strategie leczenia zakażeń HCV 1 Zespół
PRZEGL EPIDEMIOL 2006; 60: 693 700 Marek Figlerowicz 1, Agata Tyczewska 1, Magdalena Figlerowicz 2 Wpływ małych regulatorowych RNA na przebieg zakażeń wirusowych nowe strategie leczenia zakażeń HCV 1 Zespół
Regulacja transkrypcji genów eukariotycznych
 Regulacja transkrypcji genów eukariotycznych Klasyczne wyobrażenie genu fragment DNA, który koduje mrna Definicja genu GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną
Regulacja transkrypcji genów eukariotycznych Klasyczne wyobrażenie genu fragment DNA, który koduje mrna Definicja genu GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną
Nowoczesne systemy ekspresji genów
 Nowoczesne systemy ekspresji genów Ekspresja genów w organizmach żywych GEN - pojęcia podstawowe promotor sekwencja kodująca RNA terminator gen Gen - odcinek DNA zawierający zakodowaną informację wystarczającą
Nowoczesne systemy ekspresji genów Ekspresja genów w organizmach żywych GEN - pojęcia podstawowe promotor sekwencja kodująca RNA terminator gen Gen - odcinek DNA zawierający zakodowaną informację wystarczającą
DNA musi współdziałać z białkami!
 DNA musi współdziałać z białkami! Specyficzność oddziaływań między DNA a białkami wiążącymi DNA zależy od: zmian konformacyjnych wzdłuż cząsteczki DNA zróżnicowania struktury DNA wynikającego z sekwencji
DNA musi współdziałać z białkami! Specyficzność oddziaływań między DNA a białkami wiążącymi DNA zależy od: zmian konformacyjnych wzdłuż cząsteczki DNA zróżnicowania struktury DNA wynikającego z sekwencji
DNA superhelikalny eukariota DNA kolisty bakterie plazmidy mitochondria DNA liniowy wirusy otrzymywany in vitro
 DNA- kwas deoksyrybonukleinowy: DNA superhelikalny eukariota DNA kolisty bakterie plazmidy mitochondria DNA liniowy wirusy otrzymywany in vitro RNA- kwasy rybonukleinowe: RNA matrycowy (mrna) transkrybowany
DNA- kwas deoksyrybonukleinowy: DNA superhelikalny eukariota DNA kolisty bakterie plazmidy mitochondria DNA liniowy wirusy otrzymywany in vitro RNA- kwasy rybonukleinowe: RNA matrycowy (mrna) transkrybowany
THE UNFOLDED PROTEIN RESPONSE
 THE UNFOLDED PROTEIN RESPONSE Anna Czarnecka Źródło: Intercellular signaling from the endoplasmatic reticulum to the nucleus: the unfolded protein response in yeast and mammals Ch. Patil & P. Walter The
THE UNFOLDED PROTEIN RESPONSE Anna Czarnecka Źródło: Intercellular signaling from the endoplasmatic reticulum to the nucleus: the unfolded protein response in yeast and mammals Ch. Patil & P. Walter The
Składniki diety a stabilność struktury DNA
 Składniki diety a stabilność struktury DNA 1 DNA jedyna makrocząsteczka, której synteza jest ściśle kontrolowana, a powstałe błędy są naprawiane DNA jedyna makrocząsteczka naprawiana in vivo Replikacja
Składniki diety a stabilność struktury DNA 1 DNA jedyna makrocząsteczka, której synteza jest ściśle kontrolowana, a powstałe błędy są naprawiane DNA jedyna makrocząsteczka naprawiana in vivo Replikacja
Regulacja transkrypcji genów eukariotycznych
 Regulacja transkrypcji genów eukariotycznych Definicja genu GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko
Regulacja transkrypcji genów eukariotycznych Definicja genu GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną dziedziczoną cechę organizmu; zwykle koduje pojedyncze białko
Jak działają geny. Podstawy biologii molekularnej genu
 Jak działają geny Podstawy biologii molekularnej genu Uniwersalność życia Podstawowe mechanizmy są takie same u wszystkich znanych organizmów budowa DNA i RNA kod genetyczny repertuar aminokwasów budujących
Jak działają geny Podstawy biologii molekularnej genu Uniwersalność życia Podstawowe mechanizmy są takie same u wszystkich znanych organizmów budowa DNA i RNA kod genetyczny repertuar aminokwasów budujących
WYKŁAD: Klasyczny przepływ informacji ( Dogmat) Klasyczny przepływ informacji. Ekspresja genów realizacja informacji zawartej w genach
 WYKŁAD: Ekspresja genów realizacja informacji zawartej w genach Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej Klasyczny przepływ informacji ( Dogmat) Białka Retrowirusy Białka Klasyczny
WYKŁAD: Ekspresja genów realizacja informacji zawartej w genach Prof. hab. n. med. Małgorzata Milkiewicz Zakład Biologii Medycznej Klasyczny przepływ informacji ( Dogmat) Białka Retrowirusy Białka Klasyczny
transkrypcja chromatyny
 transkrypcja chromatyny problem: Jak to może e działać: dysocjacja histonów? kompleks pol II RNA >500 kd skip: split: nukleosom: 145 bp DNA = 100 kd rdzeń histonowy = 100 kd strip: + Skip czy Split czy
transkrypcja chromatyny problem: Jak to może e działać: dysocjacja histonów? kompleks pol II RNA >500 kd skip: split: nukleosom: 145 bp DNA = 100 kd rdzeń histonowy = 100 kd strip: + Skip czy Split czy
Komórka stuktura i funkcje. Bogusław Nedoszytko. WSZPIZU Wydział w Gdyni
 Komórka stuktura i funkcje Bogusław Nedoszytko WSZPIZU Wydział w Gdyni Jądro komórkowe Struktura i funkcje Podziały komórkowe Jądro komórkowe 46 chromosomów 2,6 metra DNA 3 miliardy par nukleotydów (A,T,G,C)
Komórka stuktura i funkcje Bogusław Nedoszytko WSZPIZU Wydział w Gdyni Jądro komórkowe Struktura i funkcje Podziały komórkowe Jądro komórkowe 46 chromosomów 2,6 metra DNA 3 miliardy par nukleotydów (A,T,G,C)
TECHNIKI ANALIZY RNA TECHNIKI ANALIZY RNA TECHNIKI ANALIZY RNA
 DNA 28SRNA 18/16S RNA 5SRNA mrna Ilościowa analiza mrna aktywność genów w zależności od wybranych czynników: o rodzaju tkanki o rodzaju czynnika zewnętrznego o rodzaju upośledzenia szlaku metabolicznego
DNA 28SRNA 18/16S RNA 5SRNA mrna Ilościowa analiza mrna aktywność genów w zależności od wybranych czynników: o rodzaju tkanki o rodzaju czynnika zewnętrznego o rodzaju upośledzenia szlaku metabolicznego
Regulacja transkrypcji genów eukariotycznych
 Regulacja transkrypcji genów eukariotycznych Klasyczne wyobrażenie genu fragment DNA, który koduje mrna Definicja genu GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną
Regulacja transkrypcji genów eukariotycznych Klasyczne wyobrażenie genu fragment DNA, który koduje mrna Definicja genu GEN- podstawowa jednostka dziedziczenia Region DNA, który określa charakterystyczną
Zarówno u organizmów eukariotycznych, jak i prokariotycznych proces replikacji ma charakter semikonserwatywny.
 HIPTEZY WYJAŚIAJĄCE MECHAIZM REPLIKACJI C. Model replikacji semikonserwatywnej zakłada on, że obie nici macierzystej cząsteczki DA są matrycą dla nowych, dosyntetyzowywanych nici REPLIKACJA każda z dwóch
HIPTEZY WYJAŚIAJĄCE MECHAIZM REPLIKACJI C. Model replikacji semikonserwatywnej zakłada on, że obie nici macierzystej cząsteczki DA są matrycą dla nowych, dosyntetyzowywanych nici REPLIKACJA każda z dwóch
Ekspresja informacji genetycznej
 Ekspresja informacji genetycznej Informacja o budowie i funkcjonowaniu organizmu jest zakodowana w sekwencji nukleotydów cząsteczek DNA i podzielona na dużą liczbę genów. Gen jest odcinkiem DNA kodującym
Ekspresja informacji genetycznej Informacja o budowie i funkcjonowaniu organizmu jest zakodowana w sekwencji nukleotydów cząsteczek DNA i podzielona na dużą liczbę genów. Gen jest odcinkiem DNA kodującym
cytoplazma + jądro komórkowe = protoplazma Jądro komórkowe
 Komórka eukariotyczna http://pl.wikipedia.org/w/index.php?title=plik:hela_cells_stained_with_hoechst_33258.jpg cytoplazma + jądro komórkowe = protoplazma W cytoplazmie odbywa się: cała przemiana materii,
Komórka eukariotyczna http://pl.wikipedia.org/w/index.php?title=plik:hela_cells_stained_with_hoechst_33258.jpg cytoplazma + jądro komórkowe = protoplazma W cytoplazmie odbywa się: cała przemiana materii,
Dr hab. Anna Bębenek Warszawa,
 Dr hab. Anna Bębenek Warszawa, 14.01. 2018 Instytut Biochemii i Biofizyki PAN Ul. Pawińskiego 5a 02-106 Warszawa Recenzja pracy doktorskiej Pana mgr Michała Płachty Pod Tytułem Regulacja funkcjonowania
Dr hab. Anna Bębenek Warszawa, 14.01. 2018 Instytut Biochemii i Biofizyki PAN Ul. Pawińskiego 5a 02-106 Warszawa Recenzja pracy doktorskiej Pana mgr Michała Płachty Pod Tytułem Regulacja funkcjonowania
Prokariota i Eukariota
 Prokariota i Eukariota W komórkach organizmów żywych ilość DNA jest zazwyczaj stała i charakterystyczna dla danego gatunku. ILOŚĆ DNA PRZYPADAJĄCA NA APARAT GENETYCZNY WZRASTA WRAZ Z BARDZIEJ FILOGENETYCZNIE
Prokariota i Eukariota W komórkach organizmów żywych ilość DNA jest zazwyczaj stała i charakterystyczna dla danego gatunku. ILOŚĆ DNA PRZYPADAJĄCA NA APARAT GENETYCZNY WZRASTA WRAZ Z BARDZIEJ FILOGENETYCZNIE
Mechanizmy kontroli rozwoju roślin. Rafał Archacki
 Mechanizmy kontroli rozwoju roślin Rafał Archacki Drzewo życia pozycja roślin i zwierząt http://5e.plantphys.net/article.php?ch=t&id=399 Ewolucja roślin ewolucja procesu rozmnażania i rozwoju http://5e.plantphys.net/article.php?ch=t&id=399
Mechanizmy kontroli rozwoju roślin Rafał Archacki Drzewo życia pozycja roślin i zwierząt http://5e.plantphys.net/article.php?ch=t&id=399 Ewolucja roślin ewolucja procesu rozmnażania i rozwoju http://5e.plantphys.net/article.php?ch=t&id=399
NUTRIGENOMIKA na co mają geny apetyt. Ewa Róg - Zielińska
 NUTRIGENOMIKA na co mają geny apetyt Ewa Róg - Zielińska NUTRIGENOMIKA badanie zależności między żywieniem a odpowiedzią organizmu na poziomie ekspresji genów dieta ma wpływ na każdy etap ekspresji - na
NUTRIGENOMIKA na co mają geny apetyt Ewa Róg - Zielińska NUTRIGENOMIKA badanie zależności między żywieniem a odpowiedzią organizmu na poziomie ekspresji genów dieta ma wpływ na każdy etap ekspresji - na
Badanie dynamiki białek jądrowych w żywych komórkach metodą mikroskopii konfokalnej
 Badanie dynamiki białek jądrowych w żywych komórkach metodą mikroskopii konfokalnej PRAKTIKUM Z BIOLOGII KOMÓRKI () ćwiczenie prowadzone we współpracy z Pracownią Biofizyki Komórki Badanie dynamiki białek
Badanie dynamiki białek jądrowych w żywych komórkach metodą mikroskopii konfokalnej PRAKTIKUM Z BIOLOGII KOMÓRKI () ćwiczenie prowadzone we współpracy z Pracownią Biofizyki Komórki Badanie dynamiki białek
srna: małe cząsteczki o dużych możliwościach
 Plan srna: małe cząsteczki o dużych możliwościach Marek Daniel Koter, Marcin Filipecki Katedra Genetyki, Hodowli i Biotechnologii Roślin Wydział Ogrodnictwa, Biotechnologii i Architektury Krajobrazu Szkoła
Plan srna: małe cząsteczki o dużych możliwościach Marek Daniel Koter, Marcin Filipecki Katedra Genetyki, Hodowli i Biotechnologii Roślin Wydział Ogrodnictwa, Biotechnologii i Architektury Krajobrazu Szkoła
Wykład 13. Regulacja cyklu komórkowego w odpowiedzi na uszkodzenia DNA. Mechanizmy powstawania nowotworów
 Wykład 13 Regulacja cyklu komórkowego w odpowiedzi na uszkodzenia DNA Mechanizmy powstawania nowotworów Uszkodzenie DNA Wykrycie uszkodzenia Naprawa DNA Zatrzymanie cyklu kom. Apoptoza Źródła uszkodzeń
Wykład 13 Regulacja cyklu komórkowego w odpowiedzi na uszkodzenia DNA Mechanizmy powstawania nowotworów Uszkodzenie DNA Wykrycie uszkodzenia Naprawa DNA Zatrzymanie cyklu kom. Apoptoza Źródła uszkodzeń
BIOINFORMATYKA. edycja 2016 / wykład 11 RNA. dr Jacek Śmietański
 BIOINFORMATYKA edycja 2016 / 2017 wykład 11 RNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Rola i rodzaje RNA 2. Oddziaływania wewnątrzcząsteczkowe i struktury
BIOINFORMATYKA edycja 2016 / 2017 wykład 11 RNA dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Rola i rodzaje RNA 2. Oddziaływania wewnątrzcząsteczkowe i struktury
Historia informacji genetycznej. Jak ewolucja tworzy nową informację (z ma ą dygresją).
 Historia informacji genetycznej. Jak ewolucja tworzy nową informację (z ma ą dygresją). Czym jest życie? metabolizm + informacja (replikacja) 2 Cząsteczki organiczne mog y powstać w atmosferze pierwotnej
Historia informacji genetycznej. Jak ewolucja tworzy nową informację (z ma ą dygresją). Czym jest życie? metabolizm + informacja (replikacja) 2 Cząsteczki organiczne mog y powstać w atmosferze pierwotnej
Translacja i proteom komórki
 Translacja i proteom komórki 1. Kod genetyczny 2. Budowa rybosomów 3. Inicjacja translacji 4. Elongacja translacji 5. Terminacja translacji 6. Potranslacyjne zmiany polipeptydów 7. Translacja a retikulum
Translacja i proteom komórki 1. Kod genetyczny 2. Budowa rybosomów 3. Inicjacja translacji 4. Elongacja translacji 5. Terminacja translacji 6. Potranslacyjne zmiany polipeptydów 7. Translacja a retikulum
Podstawy genetyki molekularnej
 Podstawy genetyki molekularnej Materiał genetyczny Materiałem genetycznym są kwasy nukleinowe Materiałem genetycznym organizmów komórkowych jest kwas deoksyrybonukleinowy (DNA) 5 DNA zbudowany jest z nukleotydów
Podstawy genetyki molekularnej Materiał genetyczny Materiałem genetycznym są kwasy nukleinowe Materiałem genetycznym organizmów komórkowych jest kwas deoksyrybonukleinowy (DNA) 5 DNA zbudowany jest z nukleotydów
Geny i działania na nich
 Metody bioinformatyki Geny i działania na nich prof. dr hab. Jan Mulawka Trzy królestwa w biologii Prokaryota organizmy, których komórki nie zawierają jądra, np. bakterie Eukaryota - organizmy, których
Metody bioinformatyki Geny i działania na nich prof. dr hab. Jan Mulawka Trzy królestwa w biologii Prokaryota organizmy, których komórki nie zawierają jądra, np. bakterie Eukaryota - organizmy, których
BUDOWA I FUNKCJA GENOMU LUDZKIEGO
 BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
Rzęski, wici - budowa Mikrotubule. rozmieszczenie organelli. Stabilne mikrotubule szkielet rzęsek i wici
 Mikrotubule dynamiczna niestabilność - stabilizacja rozmieszczenie organelli ER Golgi organizują wnętrze komórki - polaryzacja komórki Mt Mt organizacja ER, aparatu Golgiego przemieszczanie mitochondriów
Mikrotubule dynamiczna niestabilność - stabilizacja rozmieszczenie organelli ER Golgi organizują wnętrze komórki - polaryzacja komórki Mt Mt organizacja ER, aparatu Golgiego przemieszczanie mitochondriów
Modyfikacje histonów rdzeniowych
 Modyfikacje histonów rdzeniowych Fosforylacja (seryna, treonina) Acetylacja (lizyna) Modyfikacje histonów rdzeniowych Metylacja (lizyna, arginina) ADP-rybozylacja Ubikwitynylacja (lizyna) Sumoilacja (lizyna)
Modyfikacje histonów rdzeniowych Fosforylacja (seryna, treonina) Acetylacja (lizyna) Modyfikacje histonów rdzeniowych Metylacja (lizyna, arginina) ADP-rybozylacja Ubikwitynylacja (lizyna) Sumoilacja (lizyna)
Kwasy nukleinowe. Replikacja
 Kwasy nukleinowe Replikacja Białko Helikaza Prymaza SSB Funkcja w replikacji DNA Rozplata podwójną helisę Syntetyzuje starterowy odcinek RNA Stabilizuje regiony jednoniciowe Gyraza DNA Wprowadza ujemne
Kwasy nukleinowe Replikacja Białko Helikaza Prymaza SSB Funkcja w replikacji DNA Rozplata podwójną helisę Syntetyzuje starterowy odcinek RNA Stabilizuje regiony jednoniciowe Gyraza DNA Wprowadza ujemne
Nowe oblicze świata RNA
 NAUKA 2/2009 93-109 AGATA TYCZEWSKA 1, MAREK FIGLEROWICZ 2 Nowe oblicze świata RNA Wstęp Jednym z najistotniejszych osiągnięć biologii w drugiej połowie XX wieku było poznanie podstaw molekularnych procesu
NAUKA 2/2009 93-109 AGATA TYCZEWSKA 1, MAREK FIGLEROWICZ 2 Nowe oblicze świata RNA Wstęp Jednym z najistotniejszych osiągnięć biologii w drugiej połowie XX wieku było poznanie podstaw molekularnych procesu
mirna i zmiany faz wzrostu wegetatywnego
 mirna i zmiany faz wzrostu wegetatywnego mir156 reguluje ekspresję genów SPL (SQUAMOSA PROMOTER BINDING PROTEIN-LIKE) Defekty morfologiczne wywołane nadekspresją mirna w Arabidopsis" mirna156 mirna166
mirna i zmiany faz wzrostu wegetatywnego mir156 reguluje ekspresję genów SPL (SQUAMOSA PROMOTER BINDING PROTEIN-LIKE) Defekty morfologiczne wywołane nadekspresją mirna w Arabidopsis" mirna156 mirna166
WPROWADZENIE DO GENETYKI MOLEKULARNEJ
 WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
Wykład 1. Od atomów do komórek
 Wykład 1. Od atomów do komórek Skład chemiczny komórek roślinnych Składniki mineralne (nieorganiczne) - popiół Substancje organiczne (sucha masa) - węglowodany - lipidy - kwasy nukleinowe - białka Woda
Wykład 1. Od atomów do komórek Skład chemiczny komórek roślinnych Składniki mineralne (nieorganiczne) - popiół Substancje organiczne (sucha masa) - węglowodany - lipidy - kwasy nukleinowe - białka Woda
Transport makrocząsteczek
 Komórka eukariotyczna cytoplazma + jądro komórkowe = protoplazma W cytoplazmie odbywa się: cała przemiana materii, dzięki której organizm uzyskuje energię biosynteza białka i innych związków Transport
Komórka eukariotyczna cytoplazma + jądro komórkowe = protoplazma W cytoplazmie odbywa się: cała przemiana materii, dzięki której organizm uzyskuje energię biosynteza białka i innych związków Transport
WPROWADZENIE DO GENETYKI MOLEKULARNEJ
 WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
WPROWADZENIE DO GENETYKI MOLEKULARNEJ Replikacja organizacja widełek replikacyjnych Transkrypcja i biosynteza białek Operon regulacja ekspresji genów Prowadzący wykład: prof. dr hab. Jarosław Burczyk REPLIKACJA
mikrosatelitarne, minisatelitarne i polimorfizm liczby kopii
 Zawartość 139371 1. Wstęp zarys historii genetyki, czyli od genetyki klasycznej do genomiki 2. Chromosomy i podziały jądra komórkowego 2.1. Budowa chromosomu 2.2. Barwienie prążkowe chromosomów 2.3. Mitoza
Zawartość 139371 1. Wstęp zarys historii genetyki, czyli od genetyki klasycznej do genomiki 2. Chromosomy i podziały jądra komórkowego 2.1. Budowa chromosomu 2.2. Barwienie prążkowe chromosomów 2.3. Mitoza
Plan wykładu: Budowa chromatyny - nukleosomy. Wpływ nukleosomów na replikację i transkrypcję
 Nukleosomy 1 Plan wykładu: Budowa chromatyny - nukleosomy Wpływ nukleosomów na replikację i transkrypcję Metody pozwalające na wyznaczanie miejsc wiązania nukleosomów Charakterystyka obsadzenia nukleosomów
Nukleosomy 1 Plan wykładu: Budowa chromatyny - nukleosomy Wpływ nukleosomów na replikację i transkrypcję Metody pozwalające na wyznaczanie miejsc wiązania nukleosomów Charakterystyka obsadzenia nukleosomów
Transport makrocząsteczek (białek)
 Transport makrocząsteczek (białek) Transport makrocząsteczek sortowanie białek - sekwencje sygnałowe lata 70-te XX w. - Günter Blobel - hipoteza sygnałowa; 1999r - nagroda Nobla Sekwencja sygnałowa: A
Transport makrocząsteczek (białek) Transport makrocząsteczek sortowanie białek - sekwencje sygnałowe lata 70-te XX w. - Günter Blobel - hipoteza sygnałowa; 1999r - nagroda Nobla Sekwencja sygnałowa: A
WSPÓ ZALE NOŒÆ POMIÊDZY METYLACJ CYTOZYNY I MODYFIKACJAMI CHROMATYNY
 METYLACJA CYTOZYNY A STRUKTURA CHROMATYNY 679 POSTÊPY BIOLOGII KOMÓRKI TOM 32 2005 NR 4 (679 696) WSPÓ ZALE NOŒÆ POMIÊDZY METYLACJ CYTOZYNY I MODYFIKACJAMI CHROMATYNY INTERRELATIONSHIP BETWEEN CYTOSINE
METYLACJA CYTOZYNY A STRUKTURA CHROMATYNY 679 POSTÊPY BIOLOGII KOMÓRKI TOM 32 2005 NR 4 (679 696) WSPÓ ZALE NOŒÆ POMIÊDZY METYLACJ CYTOZYNY I MODYFIKACJAMI CHROMATYNY INTERRELATIONSHIP BETWEEN CYTOSINE
GENOM I JEGO STRUKTURA
 GENOM I JEGO STRUKTURA GENOM Ogół materiału genetycznego (kwasu nukleinowego niosącego informację genetyczną) zawartego w pojedynczej części składowej (komórce, cząstce wirusa) organizmu 1 Genom eukariotyczny
GENOM I JEGO STRUKTURA GENOM Ogół materiału genetycznego (kwasu nukleinowego niosącego informację genetyczną) zawartego w pojedynczej części składowej (komórce, cząstce wirusa) organizmu 1 Genom eukariotyczny
Transkrypcja i obróbka RNA. Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego.
 Transkrypcja i obróbka RNA Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Centralny dogmat biologii molekularnej: sekwencja DNA zostaje
Transkrypcja i obróbka RNA Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Centralny dogmat biologii molekularnej: sekwencja DNA zostaje
Całogenomowa analiza niskocząsteczkowych RNA, pochodzących z trna w Arabidopsis thaliana
 UNIWERSYTET IM. ADAMA MICKIEWICZA Wydział Biologii Całogenomowa analiza niskocząsteczkowych RNA, pochodzących z trna w Arabidopsis thaliana Agnieszka Thompson Instytut Biologii Molekularnej i Biotechnologii
UNIWERSYTET IM. ADAMA MICKIEWICZA Wydział Biologii Całogenomowa analiza niskocząsteczkowych RNA, pochodzących z trna w Arabidopsis thaliana Agnieszka Thompson Instytut Biologii Molekularnej i Biotechnologii
Fragment cząsteczki DNA stanowiący matrycę dla syntezy cząsteczki lub podjednostki białka nazywamy GENEM
 KONTROLA EKSPRESJI GENU PRZEKAZYWANIE INFORMACJI GENETYCZNEJ Informacja genetyczna - instrukcje kierujące wszystkimi funkcjami komórki lub organizmu zapisane jako określone, swoiste sekwencje nukleotydów
KONTROLA EKSPRESJI GENU PRZEKAZYWANIE INFORMACJI GENETYCZNEJ Informacja genetyczna - instrukcje kierujące wszystkimi funkcjami komórki lub organizmu zapisane jako określone, swoiste sekwencje nukleotydów
Regulacja Ekspresji Genów
 Regulacja Ekspresji Genów Wprowadzenie o Ekspresja genu jest to złożony proces jego transkrypcji do mrna, o Obróbki tego mrna, a następnie o Translacji do białka. 4/17/2019 2 4/17/2019 3 E 1 GEN 3 Promotor
Regulacja Ekspresji Genów Wprowadzenie o Ekspresja genu jest to złożony proces jego transkrypcji do mrna, o Obróbki tego mrna, a następnie o Translacji do białka. 4/17/2019 2 4/17/2019 3 E 1 GEN 3 Promotor
Metylacja DNA. Anna Fogtman Pracownia Analiz Mikromacierzy Uniwersytet Warszawski Polska Akademia Nauk
 Metylacja DNA Anna Fogtman Pracownia Analiz Mikromacierzy Uniwersytet Warszawski Polska Akademia Nauk Przykład symfoniczny Przykład symfoniczny Metylacja DNA O SAM-CH SAM NH 3 5 2 6 1 H DNMT Cytozyna
Metylacja DNA Anna Fogtman Pracownia Analiz Mikromacierzy Uniwersytet Warszawski Polska Akademia Nauk Przykład symfoniczny Przykład symfoniczny Metylacja DNA O SAM-CH SAM NH 3 5 2 6 1 H DNMT Cytozyna
białka wiążące specyficzne sekwencje DNA czynniki transkrypcyjne
 białka wiążące specyficzne sekwencje DNA czynniki transkrypcyjne http://www.umass.edu/molvis/bme3d/materials/jtat_080510/exploringdna/ch_flex/chapter.htm czynniki transkrypcyjne (aktywatory/represory)
białka wiążące specyficzne sekwencje DNA czynniki transkrypcyjne http://www.umass.edu/molvis/bme3d/materials/jtat_080510/exploringdna/ch_flex/chapter.htm czynniki transkrypcyjne (aktywatory/represory)
Replikacja DNA. Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego.
 Replikacja DNA Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Replikacja DNA jest bardzo złożonym procesem, w którym biorą udział setki
Replikacja DNA Materiały dydaktyczne współfinansowane ze środków Unii Europejskiej w ramach Europejskiego Funduszu Społecznego. Replikacja DNA jest bardzo złożonym procesem, w którym biorą udział setki
Regulacja transkrypcji genów eukariotycznych
 Regulacja transkrypcji genów eukariotycznych Dr hab. Marta Koblowska, prof. UW Zakład Biologii Systemów, Wydział Biologii UW Pracownia Analiz Mikromacierzy i Sekwencjonowania UW/IBB PAN Klasyczne wyobrażenie
Regulacja transkrypcji genów eukariotycznych Dr hab. Marta Koblowska, prof. UW Zakład Biologii Systemów, Wydział Biologii UW Pracownia Analiz Mikromacierzy i Sekwencjonowania UW/IBB PAN Klasyczne wyobrażenie
1. Na podanej sekwencji przeprowadź proces replikacji, oraz do obu nici proces transkrypcji i translacji, podaj zapis antykodonów.
 mrna 1. Na podanej sekwencji przeprowadź proces replikacji, oraz do obu nici proces transkrypcji i translacji, podaj zapis antykodonów. GGA CGC GCT replikacja CCT GCG CGA transkrypcja aminokwasy trna antykodony
mrna 1. Na podanej sekwencji przeprowadź proces replikacji, oraz do obu nici proces transkrypcji i translacji, podaj zapis antykodonów. GGA CGC GCT replikacja CCT GCG CGA transkrypcja aminokwasy trna antykodony
Prof. dr.hab. Andrzej Kaczanowski Instytut Zoologii Wydział Biologii Uniwersytetu Warszawskiego
 1 Prof. dr.hab. Andrzej Kaczanowski 26.10. 2017 Instytut Zoologii Wydział Biologii Uniwersytetu Warszawskiego kaczan@biol.uw.edu.pl Recenzja rozprawy doktorskiej Pani mgr. Julity Gruchoty p.t. Proces rearanżacji
1 Prof. dr.hab. Andrzej Kaczanowski 26.10. 2017 Instytut Zoologii Wydział Biologii Uniwersytetu Warszawskiego kaczan@biol.uw.edu.pl Recenzja rozprawy doktorskiej Pani mgr. Julity Gruchoty p.t. Proces rearanżacji
Jajko czy kura? czyli gdzie dwóch się bije, tam trzeci korzysta
 Jajko czy kura? czyli gdzie dwóch się bije, tam trzeci korzysta Jacek Śmietański IIMK UJ 21.10.2015 Jajko czy kura Pytanie tytułowe Co było na początku? Jajko czy kura? Rother M., 2012 Pytanie tytułowe
Jajko czy kura? czyli gdzie dwóch się bije, tam trzeci korzysta Jacek Śmietański IIMK UJ 21.10.2015 Jajko czy kura Pytanie tytułowe Co było na początku? Jajko czy kura? Rother M., 2012 Pytanie tytułowe
Metody bioinformatyki. Ekspresja genów. prof. dr hab. Jan Mulawka
 Metody bioinformatyki Ekspresja genów prof. dr hab. Jan Mulawka Genetyczny skład prawie wszystkich komórek somatycznych organizmów wielokomórkowych jest identyczny. Fenotyp (swoistość tkankowa lub komórkowa)
Metody bioinformatyki Ekspresja genów prof. dr hab. Jan Mulawka Genetyczny skład prawie wszystkich komórek somatycznych organizmów wielokomórkowych jest identyczny. Fenotyp (swoistość tkankowa lub komórkowa)
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii
 Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
