Krzysztof Spalik 1, Marcin Piwczyński 2
|
|
|
- Wojciech Stankiewicz
- 8 lat temu
- Przeglądów:
Transkrypt
1 Tom Numer 3 4 ( ) Strony Krzysztof Spalik 1, Marcin Piwczyński 2 1 Zakład Systematyki i Geografii Roślin Instytut Botaniki Uniwersytet Warszawski Aleje Ujazdowskie 4, Warszawa 2 Zakład Taksonomii i Geografii Roślin Instytut Ekologii i Ochrony Środowiska Uniwersytet Mikołaja Kopernika Gagarina 9, Toruń spalik@biol.uw.edu.pl piwczyn@umk.pl REKONSTRUKCJA FILOGENEZY I WNIOSKOWANIE FILOGENETYCZNE W BADANIACH EWOLUCYJNYCH DARWIN, HAECKEL I FILOGENEZA W lipcu 1837 r. Darwin naszkicował w swoim notatniku schematyczny graf relacji pokrewieństwa między gatunkami, obrazujący koncepcję drzewa życia. Ta idea, ubrana w daleko doskonalszą formę graficzną, pojawiła się także 22 lata później w jego rewolucyjnym dziele O powstawaniu gatunków, ale wciąż jako koncept, a nie konkretne drzewo filogenetyczne, przedstawiające zależności ewolucyjne między gatunkami. Nie ma w tym nic dziwnego Darwin zajmował się wyjaśnianiem mechanizmów ewolucji, nie zaś odtwarzaniem jej przebiegu. Pierwsze drzewo filogenetyczne pojawiło się w Generelle Morphologie der Organismen Ernsta Heackla w 1866 r. i tę datę można przyjąć jako oficjalny początek filogenetyki gałęzi biologii zajmującej się rekonstrukcją filogenezy organizmów. Drzewo filogenetyczne Haeckla było zapisem poglądów tego wybitnego uczonego na pochodzenie organizmów i podsumowaniem ówczesnego stanu wiedzy. Jednocześnie było hipotezą naukową, podlegającą weryfikacji w toku dalszych badań. Jeśli popatrzymy na zapisane na nim zależności filogenetyczne, to okaże się, że niewiele z nich zostało poprawnie odtworzonych, a obecny obraz drzewa życia jest zasadniczo odmienny. Jednak drzewo filogenetyczne nadal pozostaje jednocześnie podsumowaniem obecnego stanu wiedzy oraz hipotezą badawczą. W filogenetyce, podobnie jak w każdej nauce przyrodniczej, nie ma prawd absolutnych, a teorie i hipotezy uznajemy za prawdziwe, jeśli nikomu, mimo usilnych prób, nie udało się ich obalić. Warto pamiętać o tym zakresie niepewności, jaki towarzyszy wszystkim prawdom naukowym, a zwłaszcza dotyczącym odtwarzania przeszłości. W tym artykule chcielibyśmy skupić się na metodyce badań filogenetycznych, a także pokazać, w jaki sposób otrzymane filogenezy służą wnioskowaniu ewolucyjnemu. Chcemy pokazać, że analizy filogenetyczne bazujące na danych molekularnych, aczkolwiek obarczone, jak w wypadku wszystkich nauk historycznych, nieuniknioną niepewnością, wsparte są na solidnych podstawach naukowych, a wypływające z nich wnioski nie są gorszej jakości od wniosków z badań eksperymentalnych.
2 486 Krzysztof Spalik, Marcin Piwczyński DWA PODEJŚCIA DO KONSTRUOWANIA DRZEWA Istnieje zasadnicza różnica metodologiczna między drzewem Haeckla a współczesnymi przedstawieniami filogenezy. To pierwsze było po prostu wyrazem poglądów badacza, wspartych wprawdzie rzetelną wiedzą i wynikającą z niej intuicją, ale nie powstało ono w wyniku żadnych określonych procedur. Współczesne drzewa są natomiast usyskiwane za pomocą określonych algorytmów obliczeniowych. Wyniki są zobiektywizowane i powtarzalne, a tym samym weryfikowalne. Ten przełom w filogenetyce został dokonany dzięki rozwojowi metod numerycznych oraz wynalezieniu komputerów, a przyniósł go nurt taksonomii zwany fenetyką, której ojcami-założycielami byli P. H. A. Sneath i R. R. Sokal. Jest paradoksem, że fenetyka jednocześnie odrzuciła biologiczny sens odtwarzania drzewa życia, a skoncentrowała się na konstruowaniu zależności wszechstronnego podobieństwa między organizmami, zakładając, że drzewo filogenetyczne wyjdzie mimochodem. Nie czyniła ona żadnych założeń o przydatności cech, traktując je równocennie. Odmienne podejście prezentowała kladystyka, której prekursorem był Willi Hennig. Ostro krytykowała ona podejście fenetyczne wskazując, że o pochodzeniu od wspólnego przodka świadczą jedynie wspólne unikatowe cechy pochodne ewolucyjnie, czyli synapomorfie, nie zaś cechy homoplastyczne: pierwotne ewolucyjnie, odziedziczone po odległym przodku (symplezjomorfie) albo powstałe niezależnie (parallelizmy). Rozróżnia się synapomorfie od homoplazji posługując się zasadą parsymonii (oszczędności), czyli wybierając spośród wszystkich możliwych drzew takie, które wyjaśnia różnorodność cech na liściach drzewa za pomocą najmniejszej liczby zmian na gałęziach, minimalizując tym samym konflikty cech. Spór między fenetyką a kladystyką był niezwykle gwałtowny dziś emocje opadły, a w efekcie status obywatelstwa we współczesnej filogenetyce zyskały sobie koncepcje z obu nurtów. Nikt dzisiaj nie kwestionuje, że nadrzędnym problemem badawczym jest rekonstrukcja filogenezy, ale ten cel jest osiągany również za pomocą metod bazujących na podobieństwie. REWOLUCJA MOLEKULARNA W FILOGENETYCE Biologia molekularna, a przede wszystkim rozwój metod łańcuchowej reakcji polimerazy (PCR) oraz sekwencjonowania DNA, zrewolucjonizowała odtwarzanie drzewa rodowego organizmów. Dane z sekwencji okazały się daleko lepszymi znacznikami dla rekonstrukcji filogenezy niż tradycyjne cechy morfologiczne, anatomiczne czy biochemiczne. Składa się na to kilka powodów. Przede wszystkim, dane z sekwencji są genetyczne przedstawiają nam od razu zapis informacji w DNA (lub RNA), podczas gdy dane z budowy organizmów mówią nam o tym zapisie pośrednio. Co gorsza, fenotyp organizmu jest wypadkową informacji genetycznej oraz jego interakcji ze środowiskiem zewnętrznym, a określona cecha morfologiczna może być determinowana przez jeden albo przez wiele loci. Wnioskowanie o podłożu genetycznym określonej cechy morfologicznej na podstawie jej zmienności jest zatem obarczone dużym błędem. Co więcej, aby taką cechę wykorzystać w analizie komputerowej, musimy jej zmienność zakodować, czyli przedstawić w formie liczb lub znaków, a sposób tego kodowania jest z konieczności arbitralny natomiast dane z sekwencji nie wymagają kodowania, ponieważ są już zapisane jako ciąg znaków. Sekwencje DNA dają nam też niezwykłą możliwość porównywania ze sobą bardzo odległych ewolucyjnie organizmów. Przykładowo trudno na podstawie morfologii czy anatomii szacować odległość ewolucyjną między człowiekiem a bakterią Escherichia coli, ich budowa jest bowiem zbyt odmienna i trudno wskazać jakiekolwiek porównywalne cechy. Mają one jednak wiele podobnych genów, np. loci, w których są zapisane sekwencje rybosomalnego DNA. Dzięki takim genom możliwe jest stworzenie kompletnego drzewa życia. Dla odtwarzania filogenezy nie bez znaczenia jest sposób, w jaki utrwaliły się analizowane zmiany cech (mutacje). Procesy prowadzące do rozpowszechnienia się mutacji możemy podzielić na dwa rodzaje: deterministyczne oraz stochastyczne (losowe). Procesem deterministycznym jest dobór naturalny mutacje korzystne zwiększają swój udział w puli genowej, natomiast niekorzystne są z niej eliminowane (patrz rozdział Łomnickie-
3 Rekonstrukcja filogenezy 487 go Dobór naturalny w tym zeszycie KOSMO- SU). Dobór naturalny jest architektem ewolucji, odpowiedzialnym za różnorodność organizmów. Paradoksalnie jednak, cechy utrwalone wskutek działania doboru mogą być zawodne w odtwarzaniu przebiegu ewolucji, silny nacisk selekcyjny sprzyja bowiem zmianom homoplastycznym konwergencjom. W filogenetyce bardziej przydatne są cechy, które utrwaliły się przypadkowo, jest bowiem mało prawdopodobne, że taka sama przypadkowa zmiana utrwali się ponowne. Gdzie takich cech szukać? Fenotyp organizmu podlega silnej presji środowiska, a zatem zdecydowana większość cech fenotypowych musiała przejść przez sito doboru. Inaczej jest na poziomie genetycznym. Kiedy poznano sekwencje genów, zauważono dużą liczbę mutacji milczących, czyli niezmieniających sekwencji kodowanego białka; jeszcze więcej mutacji stwierdzono w sekwencjach niekodujących, np. w przestrzeniach międzygenowych albo w intronach. Spostrzeżenia te zaowocowały sformułowaniem neutralnej teorii ewolucji, której autorem był japoński badacz Motoo Kimura (patrz też artykuł Łomnickiego Dryf genetyczny w tym zeszycie KOSMOSU). Zakłada ona, że większość substytucji (mutacji punktowych) jest neutralna lub prawie neutralna dla organizmu oraz że ich utrwalenie w populacji jest procesem przypadkowym. Ponieważ procesy powstawania i utrwalania mutacji są stochastyczne, to różnice między sekwencjami tego samego odcinka DNA u różnych organizmów są funkcją czasu, jaki upłynął od rozejścia się prowadzących do nich linii filogenetycznych. Umożliwia to nie tylko samo oszacowanie filogenezy, ale także przy spełnieniu dodatkowych warunków na opisanie tej filogenezy za pomocą skali czasu (patrz artykuł Jerzmanowskiego w tym zeszycie KOSMOSU). FILOGENETYKA MOLEKULARNA A TRADYCYJNA TAKSONOMIA Inwazja metod molekularnych do taksonomii oraz tradycyjnej filogenetyki bazującej na cechach morfologicznych nie odbyła się bez oporów. Wnioski płynące z badań molekularnych były rewolucyjne, obalały bowiem wiele głęboko zakorzenionych poglądów na relacje pokrewieństwa między organizmami. Niekiedy tradycyjnym taksonomom trudno było się pogodzić z tymi wnioskami, a także z tym, że badania molekularne w krótkim czasie dały odpowiedź na pytania, nad którymi oni biedzili się przez całe życie. Nieufność do wyników badań molekularnych pogłębiały błędne oznaczenia gatunków w niektórych analizach (biolodzy molekularni nie zadali sobie trudu zweryfikowania użytego do badań materiału) oraz niestabilność kladów (gałęzi drzewa) spowodowana niedostatecznym próbkowaniem taksonomicznym (liczba taksonów) i genetycznym (reprezentatywność i długość sekwencji). Ponadto, dało się zauważyć pewną nonszalancję taksonomów molekularnych, połączoną z naiwną wiarą, że drzewo molekularne jest odpowiedzią na wszystkie pytania. Wkrótce jednak okazało się, że drzewo molekularne jest nie tyle końcem, co początkiem badań trzeba bowiem je zinterpretować i sprawdzić, czy istotnie odpowiada na jakiekolwiek pytania ewolucyjne. Dziś już oba nurty molekularny i morfologiczny zgodnie koegzystują w taksonomii i biologii ewolucyjnej, korzystając wzajemnie z uzupełniających się kompetencji. Do absolutnych wyjątków należy kwestionowanie wyników badań molekularnych, jak to ostatnio uczynili Grehan i Schwartz (2009), postulując na podstawie zaledwie kilkudziesięciu cech morfologicznych, a wbrew badaniom molekularnym, że najbliższym krewnym człowieka jest orangutan, a nie szympans. Ich krytyka filogenetyki molekularnej jest naiwna i świadczy o podstawowych brakach w wiedzy odrzucają oni bowiem wyniki analiz molekularnych twierdząc, że podobieństwo molekularne nie świadczy o pokrewieństwie, ustalenie homologii jest wątpliwe, a morfologia jest bardziej stabilna ewolucyjnie. Absolutne zdumienie budzi fakt, że artykuł ten został opublikowany w bardzo prestiżowym czasopiśmie, jakim jest Journal of Biogeography. Jednak towarzyszący mu komentarz od redakcji świadczy, że głównym powodem publikacji była raczej polityczna poprawność oddanie głosu zanikającej mniejszości a sami redaktorzy mają świadomość, iż dla każdego biologa molekularnego albo taksonoma lub antropologa choćby nieco obeznanego z filogenetyką molekularną wnioski autorów są nonsensowne. Filogenetyka molekularna to jednak coś więcej niż prosta analiza podobieństwa molekularnego, co oczywiście nie znaczy, że wnioskowanie filogenetyczne na podstawie danych molekularnych jest zawsze bezbłędne i nieobarczone niepewnością. Warto sobie uświadomić źródła tej niepewności.
4 488 Krzysztof Spalik, Marcin Piwczyński HOMOLOGIA SEKWENCJI I SYGNAŁ FILOGENETYCZNY Porównując te same sekwencje DNA otrzymane od osobników z różnych populacji lub z różnych gatunków możemy oczekiwać, że bliżej spokrewnione będą osobniki (gatunki), które różnią się mniejszą liczbą mutacji. Czy zatem wnioskowanie o pokrewieństwach między organizmami jest prostym zabiegiem polegającym na porównaniu sekwencji i obliczeniu liczby różniących je podstawień? Sytuacja nie jest tak prosta, a droga do odtworzenia filogenezy jest pełna pułapek. Po pierwsze, sekwencje wybrane do analizy powinny być homologiczne, czyli pochodzące od wspólnego przodka. Homologia na poziomie sekwencji ma jednak dwojakie oblicze. Sekwencje ortologiczne zajmują ten sam locus i ewoluują niezależnie od czasu rozejścia się linii filogenetycznych, czyli od specjacji. To one niosą sygnał filogenetyczny zapis historii ewolucyjnej danej linii ewolucyjnej. W trakcie ewolucji regularnie występują jednak także duplikacje loci (patrz artykuł Jerzmanowskiego w tym zeszycie KO- SMOSU), w wyniku czego powstają sekwencje paralogiczne. Pomieszanie sekwencji ortologicznych i paralogicznych uniemożliwia odtworzenie prawidłowej filogenezy, ponieważ sekwencje paralogiczne ewoluują niezależnie od momentu duplikacji locus, a nie od rozejścia się linii filogenetycznych. Wybór sekwencji ortologicznych nie gwarantuje jednak, że informacja o ich historii ewolucyjnej jest niezaburzona. Procesami, które powodują, że sekwencje są do siebie bardziej podobne, niżby to wynikało z czasu, który upłynął od ich rozejścia się, są: mutacje wsteczne (rewersje), czyli powrót do nukleotydu występującego w sekwencji u wspólnego przodka; wielokrotne podstawienia, czyli kilkukrotne zamiany nukleotydów w tym samym miejscu, wskutek czego obserwujemy mniej podstawień, niż ich w rzeczywistości było; podstawienia równoległe, czyli niezależne podstawienia w tej samej pozycji przez ten sam nukleotyd w obu porównywanych sekwencjach. Wszystkie te procesy zaburzają liniową zależność między czasem rozejścia się organizmów a liczbą obserwowanych mutacji oraz zacierają sygnał filogenetyczny, czyli mutacje synapomorficzne, dzięki którym można zidentyfikować pokrewieństwo gatunków. Bardzo istotnym problemem jest także zidentyfikowanie homologicznych pozycji w sekwencji, czyli dokonanie ich przyrównania. Nie zawsze jest to zadanie łatwe, ponieważ w trakcie ewolucji zachodzą nie tylko podstawienia nukleotydów, ale także ich insercje (wstawienia) i delecje (usunięcia). W wypadku sekwencji kodujących białka insercje i delecje są zazwyczaj usuwane przez dobór oczyszczający, albowiem wstawienie bądź usunięcie jednego lub dwóch nukleotydów zmienia odczyt, wskutek czego białko przestaje być funkcjonalne. Jedynie wstawienia trzech (albo wielokrotności trzech) nukleotydów mają szansę na przejście przez sito doboru. Natomiast w sekwencjach niekodujących, np. w intronach lub przestrzeniach międzygenowych, delecje i insercje zdarzają się często. Proces przyrównywania sekwencji jest kluczowy do właściwego oszacowania pokrewieństw między organizmami żywymi i obecnie istnieje wiele algorytmów umożliwiających dokonanie takiego przyrównania. UKORZENIANIE DRZEWA Przyjrzyjmy się strukturze drzewa filogenetycznego jako zapisowi relacji pokrewieństwa ewolucyjnego między organizmami. Drzewo to zbudowane jest z węzłów zewnętrznych i wewnętrznych i łączących je gałęzi (Ryc. 1). W drzewie w pełni rozwiązanym każdy węzeł wewnętrzny połączony jest z innymi węzłami za pomocą trzech gałęzi, zaś do węzłów zewnętrznych prowadzi tylko jedna. W drzewie nie w pełni rozwiązanym gałęzi wychodzących z jednego węzła może być więcej, czyli występują politomie. Węzły zewnętrzne nazywamy inaczej liśćmi; każdy z nich odpowiada badanemu organizmowi. Natomiast węzły wewnętrzne można przypisać hipotetycznym wspólnym przodkom określonych konarów (kladów) drzewa. Drzewo zrekonstruowane metodami filogenetycznymi jest zazwyczaj drzewem niezakorzenionym, a więc takim, w którym nieznany jest kierunek ewolucji. Innymi słowy, nie wiemy, która z tych trzech gałęzi wchodzi do dane-
5 Rekonstrukcja filogenezy 489 Rycina 1. Struktura drzewa niezakorzenionego (1) i zakorzenionego (2). Oba drzewa mają tę samą topologię. A, B, C, D oznaczają liście, czyli węzły zewnętrzne drzewa, zaś E, F i G węzły wewnętrzne, przy czym drzewo (2) jest ukorzenione w węźle G. Strzałki przy drzewie zakorzenionym wskazują kierunek ewolucji, w przeciwieństwie do drzewa niezakorzenionego, w którym kierunek ten jest nieznany. go węzła, a które z niego wychodzą (Ryc. 1). Ukorzenienie polega na dodaniu dodatkowego węzła na jednej z gałęzi, tożsamego ze wspólnym przodkiem wszystkich badanych organizmów. Innymi słowy, łamiemy tę gałąź na dwie oraz do miejsca złamania (węzła) dołączamy korzeń drzewa. Po ukorzenieniu drzewa możemy zauważyć, że zmienia się status gałęzi w węzłach. W drzewie niezakorzenionym wszystkie trzy gałęzie zbiegające się w węźle wewnętrznym są równocenne, natomiast w drzewie zakorzenionym jedna z nich jest gałęzią wchodzącą, a dwie wychodzącymi. Grupy wywodzące się z jednego węzła nazywamy siostrzanymi. Warto zauważyć, że bez ukorzenienia drzewa nie możemy wyciągać żadnych sensownych wniosków o ewolucji badanej grupy, np. o monofiletyzmie określonych taksonów albo o kierunku zmian morfologicznych. Najlepszym sposobem ukorzenienia drzewa jest uwzględnienie w analizie filogenetycznej nie tylko badanej grupy, ale także jej najbliższych krewnych, czyli tzw. grupy zewnętrznej. Odszukujemy na uzyskanym drzewie wspólny wewnętrzny węzeł dla badanej grupy i wspólny wewnętrzny węzeł dla grupy zewnętrznej, a następnie przełamujemy łączącą je gałąź. Przykładowo, wiemy z innych badań, że grupą siostrzaną roślin okrytozalążkowych są nagozalążkowe. Rekonstruując filogenezę okrytozalążkowych, wybieramy więc sekwencje jednego lub kilku nagozalążkowych, np. sosny, sagowca, miłorzębu lub welwiczji, jako przedstawicieli grupy zewnętrznej. Następnie na drzewie odszukujemy gałąź łączącą okryto- i nagozalążkowe i przełamujemy ją, dodając korzeń. Dzięki takiemu zabiegowi jesteśmy w stanie stwierdzić, w jakiej kolejności oddzielały się poszczególne linie rodowe okrytozalążkowych i która grupa współczesnych gatunków jest filogenetycznie najstarsza. Ukorzenianie drzewa za pomocą grupy zewnętrznej jest standardową procedurą w badaniach filogenetycznych. Warto jednak zauważyć, że błędne wybranie grupy zewnętrznej, a tym samym błędne zakorzenienie, sprawia, że błędnie odczytujemy na nim kierunek ewolucji. Wszystkie metody szacowania filogenezy dają możliwość obliczenia długości gałęzi łączących poszczególne węzły. Jeśli długość gałęzi jest proporcjonalna do liczby mutacji (obserwowanej lub oszacowanej), które zaszły między węzłami, to takie drzewo nazywamy filogramem. Natomiast jeśli długość gałęzi odpowiada czasowi względnemu lub absolutnemu, wtedy mówimy o chronogramie. Czasami interesuje nas tylko topologia drzewa (wzór rozgałęzień), a długość gałęzi jest nieistotna takie drzewo nazywamy kladogramem. DRZEWO GATUNKÓW I DRZEWO GENÓW Warto zwrócić uwagę, że drzewo filogenetyczne odzwierciedla relację między przyrównanymi sekwencjami, dlatego też jest ono zawsze drzewem genów. Nie zawsze historia ewolucyjna genów odpowiada historii gatunków. Mechanizmów prowadzących do takich niezgodności jest kilka. Najważniejsze to: rekombinacja genów paralogicznych lub rekrutacja pseudogenów, wskutek czego powstały rekombinowany allel zawiera sygnał filogenetyczny z dwóch lub więcej loci; horyzontalny przepływ genów, czyli przeskoczenie materiału genetycznego z jednej linii filogenetycznej do drugiej; zjawisko to jest powszechne u bakterii, ale stosunkowo rzadkie wśród eukariotów, choć u roślin kwiatowych, szczególnie w genomie mitochondrialnym, znaleziono geny pocho-
6 490 Krzysztof Spalik, Marcin Piwczyński Rycina 2. Konstrukcja genealogii genu dla dwóch gatunków. W pierwszym przypadku (1) drzewo gatunków jest identyczne z drzewem genów, zaś w drugim (2) część alleli nie jest całkowicie posortowana. W tym przypadku drzewo genów nie jest zgodne z drzewem gatunków. Sytuacja ta występuje szczególnie u gatunków, u których specjacja zaszła stosunkowo niedawno, a liczba alleli danego genu przed rozejściem się była wysoka. dzące od bakterii, mszaków lub innych roślin kwiatowych, zwłaszcza pasożytniczych; niepełne sortowanie linii genealogicznych po rozejściu się puli genowych; ponieważ proces rozdziału alleli w trakcie specjacji jest losowy, może się zdarzyć, że do jednej puli trafią dwa odległe genealogicznie allele, bliższe allelom z drugiej puli, a nie sobie nawzajem (Ryc. 2); silny dobór premiujący polimorfizm alleli w loci, którego najlepszym przykładem są allele genów głównego układu zgodności tkankowej; przykładowo, wszystkie naczelne odziedziczyły podobny polimorfizm alleli tego układu po wspólnym przodku, a tym samym w puli genowej człowieka znajdują się allele, które są bliżej spokrewnione z odpowiednimi allelami występującymi u szympansów niż z innymi allelami u człowieka; zauważmy, że efekt takiego doboru jest podobny, jak w wypadku niepełnego sortowania linii genealogicznych, inne są jednak przyczyny obu zjawisk stochastyczne w wypadku sortowania linii genealogicznych i deterministyczne w wypadku selekcji faworyzującej polimorfizm; hybrydyzacja i introgresja 1, wskutek czego zależności międzygatunkowe opisywane są raczej za pomocą topologii sieci 2, a nie drzewa; zjawisko hybrydyzacji wydaje się stosunkowo częste u roślin, zwłaszcza okrytozalążkowych, wśród których spotykamy wiele allopoliploidów 3, powstałych właśnie wskutek hybrydyzacji. ODTWARZANIE DRZEWA Rekonstrukcja drzewa filogenetycznego jest złożonym zagadnieniem statystycznym i algorytmicznym. Istnieje wiele metod rekonstrukcji filogenezy, odwołujących się do różnych założeń statystycznych i biologicznych. Warto więc wykonywać analizę filogenetyczną za pomocą różnych narzędzi, a następnie porównać wyniki i szukać przyczyn ewentualnych rozbieżności między nimi. Wyróżniamy cztery podstawowe metody rekonstrukcji filogenezy: największej parsymonii (ang. Maximum Parsimony, MP), 1 Introgresja to krzyżowanie się mieszańca międzygatunkowego z jednym z gatunków rodzicielskich, wskutek czego dochodzi do przepływu genów z jednej puli genowej do drugiej. 2 Sieć, w przeciwieństwie do drzewa, charakteryzuje się występowaniem tzw. cykli, czyli zamkniętych ścieżek łączących poszczególne węzły. 3 Wiele gatunków roślin powstało poprzez hybrydyzację, a następnie poliploidyzację, która przywróciła homologie między chromosomami (patrz artykuł Szymury w tym zeszycie KOSMOSU).
7 Rekonstrukcja filogenezy 491 odległościowe (np. ang. Neighbour-Joining, NJ), największej wiarygodności (ang. Maximum Likelihood, ML) bayesowskie (ang. Bayesian Phylogenetics, BP). Trzy ostatnie grupy metod bazują na modelach substytucji nukleotydów. METODA NAJWIĘKSZEJ PARSYMONII Metoda największej parsymonii jest jedną z najwcześniej zaproponowanych procedur rekonstrukcji filogenezy (Camin i Sokal 1965). Polega ona na poszukiwaniu w przestrzeni wszystkich możliwych drzew takiego, które najoszczędniej tłumaczy obserwowaną zmienność cech na liściach drzewa. W tym celu odtwarza się stany poszczególnych cech w węzłach wewnętrznych drzewa, przyporządkowując jednocześnie zmiany stanów gałęziom, czyli mapując je na gałęziach. Przykładowo, jeśli w dwóch sekwencjach siostrzanych występuje nukleotyd A, to według kryterium parsymonii ich wspólny przodek ma także adeninę w tej pozycji, ponieważ taki układ nie wymaga żadnej zmiany na gałęziach. Gdyby była tam cytozyna (albo jakikolwiek inny nukleotyd), to musielibyśmy założyć, że na obu gałęziach nastąpiło podstawienie cytozyny przez adeninę. Suma wszystkich zmian dla każdego miejsca w przyrównanych sekwencjach buduje długość drzewa. Zgodnie z kryterium parsymonii, drzewo najkrótsze uważane jest za najlepsze. Mimo swojej prostoty, metoda największej parsymonii w pewnych sytuacjach zawodzi. Wykazano, że w wypadkach silnie zróżnicowanego tempa ewolucji w poszczególnych gałęziach i intensywnej radiacji (krótkich odcinków czasu między rozgałęzieniami drzewa), metoda MP jest wrażliwa na homoplazje interpretuje je jako synapomorfie. Takich fałszywych synapomorfii jest więcej na długich gałęziach (wykazujących szybsze tempo podstawiania nukleotydów), a zatem takie gałęzie są mylnie łączone. Zjawisko to nazwano efektem przyciągania się długich gałęzi. Pomimo tej krytyki metoda MP pozostaje silnym narzędziem do wnioskowania filogenetycznego, szczególnie na niskim poziomie zmienności sekwencji, głównie ze względu na niewielkie wymagania obliczeniowe oraz dość dobrze zbadane właściwości, w przeciwieństwie to tak modnej obecnie analizy bayesowskiej (patrz niżej). MODELE SUBSTYTUCJI NUKLEOTYDÓW Sposobem na uniknięcie efektu przyciągania się długich gałęzi jest uwzględnienie w szacowaniu filogenezy całkowitej liczby zmian, które na danej gałęzi zaszły, uwzględniając podstawienia wielokrotne i rewersje. Wymaga to zastosowania określonego modelu ewolucji DNA, czyli modelu substytucji nukleotydów. Z modeli takich korzystają metody odległościowe, największej wiarygodności oraz bayesowska. Ewolucję sekwencji nukleotydowych można przedstawić w postaci modeli matematycznych, które mają uzasadnienie biologiczne oraz są możliwe do implementacji algorytmicznej. Od czasu publikacji pierwszego modelu Jukesa i Cantora (1969), zakładającego jednakowe prawdopodobieństwo substytucji między wszystkimi czterema nukleotydami, opisano wiele modeli, które odchodzą od tych mało realistycznych założeń. Doprowadziło to w konsekwencji do stworzenia kilkudziesięciu modeli ewolucji DNA. Najbardziej złożony model GTR+I+γ [ang. General Time Reversible + Invariant (positions) + Gamma (distribution)] posiada 12 wolnych parametrów. Dziesięć z nich pozwala na przyporządkowanie różnego prawdopodobieństwa podstawienia jednego nukleotydu drugim (przy czym prawdopodobieństwa substytucji np. A T i T A są identyczne, a więc macierz podstawień nukleotydów jest symetryczna) oraz określenie frekwencji poszczególnych nukleotydów. Pozostałe dwa parametry pozwalają na wprowadzenie do modelu procentu miejsc niezmiennych (I) oraz zróżnicowanego tempa substytucji w różnych częściach danej sekwencji, opisanego za pomocą rozkładu gamma (γ). Wiele modeli można wyprowadzić z GTR poprzez uproszczenie jego założeń. Duża liczba modeli o różnej liczbie parametrów umożliwia matematyczny opis sekwencji pełniących róż-
8 492 Krzysztof Spalik, Marcin Piwczyński norodne role w genomie. Warto wspomnieć, że istnieją także inne modele ewolucji, które wykorzystywane są do rekonstrukcji filogenezy na podstawie sekwencji specyficznych cząsteczek, takich jak RNA czy białka. W celu zobiektywizowania procesu wyboru odpowiedniego modelu substytucji, wykorzystuje się kilka metod: LRT (ang. Likelihood-Ratio Test), AIC (ang. Akaike Information Criterion), BIC (ang. Bayesian Information Criterion). Wszystkie one pozwalają na wybranie najprostszego modelu dobrze opisującego analizowane dane. Procedura ta jest standardowo wykonywana przed użyciem metody filogenetycznej, która wymaga modelu ewolucji. METODY ODLEGŁOŚCIOWE Szacowanie filogenezy metodami odległościowymi wymaga dwóch kroków: obliczenia odległości genetycznej pomiędzy parami sekwencji, a następnie rekonstrukcji drzewa na podstawie macierzy odległości za pomocą określonego algorytmu. Najczęściej stosowaną metodą odległościową jest metoda łączenia sąsiadów (ang. Neighbour-Joining, NJ). Jedną z podstawowych zalet tej techniki jest jej szybkość obliczeniowa, nawet dla setek przyrównanych sekwencji. Uzyskujemy jednak tylko jedno drzewo, podczas gdy może istnieć wiele innych, równie dobrych drzew (o równie prawdopodobnej topologii). Dlatego też wykorzystanie tej metody jest ograniczone głównie do szybkiego oszacowania suboptymalnej zazwyczaj filogenezy. Służy ona do zgrubnej analizy danych, znajduje też zastosowanie do obliczenia wartości funkcji wiarygodności w procedurze wyboru modelu substytucji (np. w programie ModelTest) albo dostarcza drzewa stanowiącego punkt startowy do dalszych przeszukiwań (np. w metodzie maksymalnej wiarygodności). METODA NAJWIĘKSZEJ WIARYGODNOŚCI Stosowana powszechnie w statystyce metoda największej wiarygodności pomaga oszacować prawdopodobieństwo obserwowanych danych (w naszym przypadku przyrównanych sekwencji), kiedy parametry modelu są znane. Zmieniając wartości parametrów możemy znaleźć taki ich zbiór, który daje nam najwyższą wiarygodność opisu naszych danych innymi słowy, poszukujemy parametrów, dla których funkcja wiarygodności osiąga maksimum. W przypadku rekonstrukcji drzew filogenetycznych poszukiwanymi wartościami są topologia drzewa filogenetycznego oraz parametry wybranego modelu ewolucji DNA, niezbędne dla oszacowania długości gałęzi. Drzewo o najwyższej wartości funkcji wiarygodności uważane jest za najlepsze. Jednym z podstawowych argumentów za użyciem tej metody jest możliwość elastycznego wprowadzania różnych założeń w postaci parametrów oraz znane własności statystyczne. Problemem jest jednak obliczeniowa czasochłonność. Spowodowane jest to dużą liczbą parametrów do optymalizacji oraz ogromną liczbą możliwych drzew do sprawdzenia. METODA BAYESOWSKA Metoda bayesowska stała się obecnie najczęściej stosowaną techniką rekonstrukcji drzew filogenetycznych. Aby zrozumieć zasady leżące u podstaw tej metody, należy poznać dwa wzory z rachunku prawdopodobieństwa: wzór na prawdopodobieństwo całkowite i wzór Bayesa. Warto tutaj posłużyć się przykładem niezwiązanym z filogenetyką. Wyobraźmy sobie dwie urny, jedna zawiera 4 białe kule i jedną czarną, zaś druga 2 białe i 3 czarne. Wiemy także, że szansa wylosowania pierwszej urny równa się 2/3, zaś urny drugiej 1/3. Jakie jest prawdopodobieństwo wylosowania kuli białej? Jak widać, mamy tutaj dwie tury losowań, pierwsza dotyczy wylosowania urny, a druga losowania kuli. Oznaczmy zdarzenie wylosowania kuli białej literą A, natomiast wybór urny literą H. Zdarzenie H jest rozbite na dwa wykluczające się zdarzenia wybór urny pierwszej (H 1 ) lub wybór urny drugiej (H 2 ). Na wartość prawdopodobieństwa wyboru kuli białej składać się
9 Rekonstrukcja filogenezy 493 będzie prawdopodobieństwo wylosowania kuli białej z pierwszej urny P(A H 1 ) ważone przez prawdopodobieństwo wylosowania tej urny P(H 1 ) oraz prawdopodobieństwo wylosowania kuli z drugiej urny P(A H 2 ) ważone przez prawdopodobieństwo wyboru tej urny P(H 2 ). Uogólniając na dowolną liczbę wykluczających się zdarzeń H i, uzyskujemy wzór na prawdopodobieństwo całkowite: P(A) = P(A H i )P(H i ). Prawdopodobieństwo całkowite obliczamy wtedy, kiedy znamy procedurę doświadczenia i pytamy o jego najbardziej prawdopodobny wynik. Możemy jednak problem odwrócić znamy wynik doświadczenia, a chcemy zapytać o jego przebieg. Przykładowo, wiemy, że została wylosowana kula biała. Jakie jest prawdopodobieństwo, że wylosowano ją z pierwszej urny, czyli jakie jest prawdopodobieństwo zdarzenia H 1, jeśli wiemy że zaszło A? Prawdopodobieństwo P(H 1 A) jest iloczynem prawdopodobieństwa wyboru pierwszej urny P(H 1 ) i wylosowania kuli białej z tej urny P(A H 1 ), podzielonym przez prawdopodobieństwo całkowite wylosowania kuli białej. Uogólniając dla dowolnej liczby zdarzeń, prawdopodobieństwo to można zapisać jako P(H j A) = P(A H j )P(H j ) / P(A). Jest to właśnie wzór Bayesa. Jeśli zdarzenie H jest naszą hipotezą badawczą, to wzór Bayesa pozwala nam obliczyć jej prawdopodobieństwo a posteriori, czyli po zajściu zdarzenia A, pod warunkiem że znamy P(H i ), czyli prawdopodobieństwo tej hipotezy a priori (przed doświadczeniem w naszym przypadku jest to wiedza o prawdopodobieństwie wylosowania poszczególnych urn). Aby przełożyć ten przykład na język filogenetyki, wystarczy za zdarzenie A podstawić nasze dane wyjściowe, czyli przyrównane sekwencje, zaś za hipotezę H drzewo filogenetyczne wraz z długościami gałęzi. Wtedy można zadać pytanie: jakie jest prawdopodobieństwo poszczególnych drzew filogenetycznych przy danym zestawie przyrównanych sekwencji. Mimo prostoty wzoru Bayesa, jego zastosowanie w filogenetyce napotyka na poważne problemy, a mianowicie na kwestię wyboru wartości prawdopodobieństwa a priori dla stawianej hipotezy, czyli drzew filogenetycznych, oraz na pytanie, jak sprawdzić wszystkie możliwe drzewa. W drzewie filogenetycznym można wyróżnić: topologię (kolejność rozgałęzień) oraz długości gałęzi, które określone są przez parametry modelu substytucji nukleotydów. Musimy więc nadać prawdopodobieństwo a priori wszystkim składnikom budującym filogenezę. Ponieważ zazwyczaj nie mamy żadnej wiedzy na ten temat, przyjmujemy często tzw. wartości nieinformacyjne a priori, które nie wpływają na prawdopodobieństwo a posteriori a przynajmniej nie powinny wpływać, co niestety nie jest do końca prawdą. Oprócz wybrania odpowiedniego rozkładu a priori, pojawia się także problem przeszukiwania kombinacji wszystkich parametrów. Przy bardziej skomplikowanych modelach, do których należy rekonstrukcja filogenezy, statystyka bayesowska posiłkuje się algorytmem Monte Carlo z wykorzystaniem łańcuchów Markowa (ang. Markov Chain Monte Carlo, MCMC). Algorytm ten działa w ten sposób, że przeszukuje przestrzeń wszystkich możliwych filogenez, pobierając z niej próby. Zatrzymuje się jednak najdłużej w tym miejscu przestrzeni, w którym drzewa filogenetyczne mają najwyższe prawdopodobieństwo a posteriori. Drzewa o najwyższym prawdopodobieństwie zostaną próbkowane wielokrotnie i właśnie stosunek liczby próbkowań, w których uzyskano dane drzewo, do ich ogólnej liczby, to właśnie prawdopodobieństwo a posteriori danego drzewa. Jeśli nasze dane niosą ze sobą dużo informacji, w wyniku działania algorytmu otrzymamy niewielką liczbę drzew o wysokim prawdopodobieństwie i niewiele różniących się od siebie. OSZACOWANIE WEWNĘTRZNEGO WSPARCIA WĘZŁÓW Metody rekonstrukcji drzew filogenetycznych, takie jak metoda największej parsymonii, największej wiarygodności oraz odległościowe traktowane są jako tzw. oszacowania punktowe. Oznacza to, że przy odpowiednio dużej liczbie danych (i silnym sygnale filogenetycznym) otrzymujemy tylko jedno drzewo, które jest najlepsze przy danym kryterium rekonstrukcji. Nasuwa się zatem pytanie, jak ocenić niepewność w oszacowaniu poszczególnych kladów na tym drzewie. Do tego celu najczęściej wykorzystuje się metodę bootstrap. Metoda ta polega na losowaniu ze zwracaniem poszczególnych miejsc w ma-
10 494 Krzysztof Spalik, Marcin Piwczyński Rycina 3. Konstrukcja próbki bootstrap polegająca na losowaniu ze zwracaniem z oryginalnej macierzy przyrównanych sekwencji. Powstałą macierz wykorzystuje się do rekonstrukcji filogenezy. Cała procedurę powtarza się setki lub tysiące razy. cierzy przyrównanych sekwencji do momentu utworzenia nowej macierzy o tej samej liczbie miejsc (kolumn w macierzy), jak w oryginalnej. Na podstawie tej nowej macierzy rekonstruowana jest filogeneza według takiego samego kryterium, jak w wypadku danych oryginalnych. Cały ten cykl próbkowania powtarza się setki lub tysiące razy, a następnie dla każdego kladu występującego w drzewie pierwotnym zlicza się procent drzew, w których dany klad wystąpił jest to właśnie wartość wsparcia bootstrap dla danego węzła (Ryc. 3). Warto zauważyć, że jednym z założeń tej metody jest to, że miejsca w przyrównanych sekwencjach są próbkami niezależnymi. Jednak bardzo często poszczególne miejsca są ze sobą skorelowane. Przykładowo, w sekwencjach kodujących skorelowane są miejsca należące do tego samego kodonu, natomiast w sekwencjach, które nie kodują białka, ale po transkrypcji przybierają określoną i funkcjonalnie ważną strukturę przestrzenną (rrna, introny, transkrybowane przestrzenie międzygenowe itd.), skorelowane są fragmenty tworzące struktury dwuniciowe (np. w tzw. spinkach do włosów ). W takim wypadku metoda bootstrap może prowadzić do błędnego oszacowania wsparcia węzłów. FILOGENEZA I CZAS EWOLUCYJNY Zaproponowana przez Zuckerkandla i Paulinga (1965) hipoteza zegara molekularnego zakłada, że tempo ewolucji jest stałe w czasie oraz pomiędzy gałęziami drzewa filogenetycznego. Do takich założeń doprowadziły autorów wcześniejsze obserwacje dotyczące badań nad cytochromem c (Margoliash 1963) oraz fibrynopeptydami (Doolittle i Blomback 1964), które sugerowały, że różnice między peptydami są mniej więcej proporcjonalne do czasu dywergencji między gatunkami. Hipoteza zegara molekularnego otrzymała także wsparcie w postaci neutralnej teorii ewolucji molekularnej Kimury (1983). Od początku jednak zdawano sobie sprawę, że każdy taki zegar odmierza czas w różnym tempie w różnych liniach filogenetycznych, a także może przyspieszać lub zwalniać. Założenie ścisłego zegara molekularnego jest w rzeczywistości wyjątkowo rzadko spełnione, zazwyczaj tylko dla niewielkich grup blisko spokrewnionych gatunków. Badania nad tempem ewolucji molekularnej pokazały, że jest ono skorelowane z czasem generacji im krótszy czas generacji, tym szybsze tempo substytucji. U roślin czas generacji związany jest z formą życiową (drzewa i krzewy żyją dłużej niż rośliny zielne), co przekłada się na związek między formą życiową a tempem ewolucji molekularnej (Smith i Donoghue 2008). Aby uwzględnić te zjawiska przy szacowaniu czasu rozejścia się organizmów, osłabiono założenia zegara, tworząc grupę metod określanych wspólną nazwą rozluźnionego zegara molekularnego (ang. relaxed molecular clock). Opracowano
11 Rekonstrukcja filogenezy 495 różne podejścia do tego zagadnienia, np. zakładając autokorelację tempa substytucji w liniach filogenetycznych (co ma uzasadnienie, jeśli tempo substytucji jest skorelowane z czasem generacji) albo przyjmując, że tempo to jest niezależne i próbkowane z rozkładu log-normalnego. Wszystkie te metody pozwalają na uzyskanie chronogramu, a zatem drzewa, w którym długości gałęzi są proporcjonalne do czasu. Aby przełożyć długości gałęzi drzewa filogenetycznego na czas absolutny potrzebujemy tzw. punktów kalibracyjnych. Musimy bowiem pamiętać, że na długość gałęzi wpływają dwa czynniki tempo substytucji nukleotydów oraz czas. Załóżmy na przykład, że dwie sekwencje DNA różnią się między sobą podstawieniami w 10% miejsc. Jeśli tempo substytucji wynosiło 1% miejsc (pozycji w sekwencji) na milion lat, to ich wspólny przodek żył pięć milionów lat temu, ale równie dobrze obie sekwencje mogły ewoluować pięć razy szybciej przez milion lat. Sytuację tę można porównać do próby oszacowania czasu jazdy, bazując tylko i wyłącznie na wskazaniu licznika przejechanych kilometrów. Aby wykalibrować zegar molekularny, potrzebujemy datowania jakiegoś zdarzenia w przeszłości. Najlepiej, jeśli jest to skamieniałość, którą można przypisać konkretnej gałęzi wewnętrznej na drzewie filogenetycznym. Umiejscawiamy ją w węźle, z którego dana gałąź wychodzi albo do którego wchodzi (to temat do osobnej dyskusji), dzięki czemu możemy datować pozostałe węzły. W ostatnich latach nastąpił duży postęp w rozwoju metod szacowania czasów dywergencji, w tym bazujących na wnioskowaniu bayesowskim. Umożliwiają one wprowadzenie niepewności datowania punktów kalibracyjnych w postaci odpowiedniego rozkładu prawdopodobieńtwa a priori, a w wyniku uzyskuje się nie tylko punktowe oszacowanie wieku poszczególnych węzłów, ale i rozkład gęstości prawdopodobieńtwa tego oszacowania. Różnice między datowaniem za pomocą ścisłego i rozluźnionego zegara molekularnego dobrze ilustruje przykład roślin kwiatowych. Wykorzystując różne sekwencje i ścisły zegar molekularny oszacowano ich wiek na mln lat, lub 200 mln lat, a zatem te datowania były nie tylko niezgodne ze sobą, ale i z danymi kopalnymi, albowiem sugerowały, że rośliny okrytozalążkowe powstały nie tylko znacznie wcześniej niż na to wskazują ich najstarsze skamieniałości, ale nawet wcześniej niż dotychczasowe oszacowania wieku roślin nasiennych, wynoszące około mln lat. Większość datowań korzystających z rozluźnionego zegara molekularnego waha się natomiast w granicach mln lat. Na podstawie danych kopalnych powstanie roślin kwiatowych szacowano na około mln lat temu, kiedy to pojawiają się charakterystyczny dla nich pyłek oraz Archaefructus najstarsze pozostałości rośliny zielnej. Trzeba jednak nadmienić, że szacowanie czasu dywergencji za pomocą zegara molekularnego ma także swoich zdecydowanych przeciwników. Wskazują oni na arbitralność wielu decyzji, które trzeba podjąć przy takim wnioskowaniu, jak np. przypisanie skamieniałości do określonego węzła oraz wybór rozkładu a priori w analizie bayesowskiej, które znacząco wpływają na końcowy wynik. Przykładowo, w naszych badaniach nad roślinami z plemienia Oenantheae z rodziny baldaszkowatych, zmieniając przypisany punktom kalibracyjnym typ rozkładu prawdopodobieństwa a priori z równomiernego na log-normalny uzyskaliśmy dramatycznie różne oszacowania 21 lub 45 mln lat dla tego samego zbioru danych. Pokazuje to, że do wyników szacowania bezwzględnego czasu ewolucyjnego należy podchodzić z dużą ostrożnością, zwłaszcza jeśli służą one dalszemu wnioskowaniu, np. biogeograficznemu. FILOGENEZA JAKO PODSTAWA BIOLOGII PORÓWNAWCZEJ I EWOLUCYJNEJ Drzewa filogenetyczne są wykorzystywane nie tylko do weryfikacji systemu klasyfikacji organizmów, ale także do rekonstrukcji ich ewolucji i właśnie takie zastosowanie jest najbardziej ekscytujące. Ze względu na niekompletność zapisu kopalnego, zwłaszcza w wypadku organizmów lądowych, często jedynym sposobem wnioskowania o historii ewolucyjnej organizmów jest właśnie drzewo filogenetyczne i współczesna różnorodność organizmów, czyli dane neontologiczne, nazywane tak dla odróżnienia od danych paleontologicznych. Analizując rozkład cech na liściach drzewa, możemy zrekonstruować sta-
12 496 Krzysztof Spalik, Marcin Piwczyński ny tych cech w jego wewnętrznych węzłach. Podobnie jak w wypadku nukleotydów, rekonstrukcji tych można dokonać za pomocą różnych metod, w tym największej parsymonii, największej wiarygodności lub analizy bayesowskiej. Do czego może się przydać taka analiza? Czasem chcemy po prostu dobrze wyjaśnić ewolucję danej grupy organizmów, pokazać kolejne etapy jej różnicowania się lub uzyskiwania określonych adaptacji. Czasem interesuje nas koewolucja określonych cech chcielibyśmy się na przykład dowiedzieć, czy istnieją pewne syndromy adaptacyjne do określonych warunków, czyli grupy współewoluujących cech. Innym razem chcemy sprawdzić, czy uzyskanie określonej nowości ewolucyjnej zbiega się na drzewie filogenetycznym z radiacją danej grupy organizmów. Możliwości wykorzystania wiedzy o ewolucji cech jest wiele. Badania porównawcze prowadzono już od dawna, ale przed rozwojem filogenetyki molekularnej miały one wątpliwą wartość. Biologia porównawcza kręciła się w błędnym kole, albowiem dysponując jedynie danymi fenotypowymi wykorzystywała je zarówno do szacowania filogenezy, jak i rekonstrukcji ewolucji cech. Takie podejście obarczone jest poważnym błędem. Jeśli bowiem podobieństwo fenetyczne jest wynikiem ewolucji zbieżnej, to uzyskamy błędną filogenezę i zjawiska konwergencji nie wyłapiemy. Jeśli badamy korelację ewolucyjną cech, to nie możemy jej badać na filogenezie uzyskanej z tych cech (albowiem metody filogenetyczne zakładają brak tej korelacji). Dopiero filogenetyka molekularna dostarczyła silnie wspartych drzew uzyskanych na podstawie niezależnych danych i w mniejszym stopniu podatnych na konwegencję. Przez wiele lat jedyną metodą wykorzystywaną do rekonstrukcji ewolucji cech była metoda największej parsymonii. Jest to stosunkowo prosta i dobra metoda, ale podobnie jak w wypadku rekonstrukcji stanów cech nukleotydów (patrz powyżej) czasem zawodzi, zwłaszcza w dużej skali czasowej. Dlatego też coraz częściej wykorzystywane są inne metody, np. maksymalnej wiarygodności lub bayesowskie. Podobnie jak w wypadku analiz sekwencji, metody te wymagają założenia określonego modelu. Jednym z podstawowych jest model bazujący na ruchach Browna (proces Wienera). Zakłada on, że cecha ewoluuje pod wpływem dryfu genetycznego lub pod wpływem doboru naturalnego, którego kierunek zmienia się w sposób nieprzewidywalny (nie ma doboru kierunkowego). Ponieważ procesy ewolucyjne nie są czysto losowe, poszukiwano także metod, które pozwoliłyby na modelowanie siły doboru i rozluźnienie założenia o czystej losowości. Taki jest np. model bazujący na procesie stochastycznym nazwanym od dwóch holenderskich fizyków procesem Ornsteina-Uhlenbecka. Model ten jest bardziej realistyczny od modelu ruchów Browna, ponieważ ma parametr pozwalający na ograniczenia w zmianach cechy, co pozwala symulować ewolucję pod wpływem doboru naturalnego. Bardzo ciekawy empiryczny test metod rekonstrukcji cech przodków przeprowadzili Webster i Purvis (2002). Ze względu na bardzo obszerny, niemalże kompletny zapis kopalny ewolucji otwornic (Foraminifera), znali oni wartości cech przodków dla węzłów zrekonstruowanego drzewa filogenetycznego współcześnie żyjących gatunków. Mogli więc porównać oszacowania tych węzłów za pomocą różnych metod ze stanem faktycznym. Okazało się, że najlepiej sprawdziła się metoda bazująca na modelu Ornsteina-Uhlenbecka. Warto wspomnieć, że rekonstrukcja cech przodków nie musi się ograniczać tylko do cech fenotypowych organizmu, ale może dotyczyć jego środowiska życia albo zasięgu geograficznego. Takie pytania rodzą się w badaniach biogeografii historycznej, paleoekologii lub uwarunkowań kladogenezy. Badając np. zmiany tempa dywersyfikacji czyli wypadkowej specjacji i wymierania pytamy, który z czynników odpowiada za to zjawisko. Najczęściej wymienia się dwa typy uwarunkowań, które mogą mieć wpływ na zmiany tempa dywersyfikacji: a) uwarunkowania wewnętrzne, jakimi są inherentne właściwości organizmów sprzyjające ewolucyjnemu różnicowaniu się; zwraca się szczególną uwagę na kluczowe innowacje adaptacyjne u roślin są to cechy związane z morfologią kwiatów, formą życiową oraz typem owocu i związanym z nim mechanizmem rozsiewania się; b) uwarunkowania zewnętrzne, jakimi są np. czynniki geograficzne i klimatyczne; powstawanie barier sprzyja specjacji, natomiast zanikanie barier ułatwia migracje; takie bariery mogą powstawać wskutek zjawisk geologicznych (wędrówki kontynentów, zanikanie i pojawianie się pomostów lądowych, zmiany poziomu mórz, orogeneza itd.) albo klimatycznych (bariery termiczne, zlodowacenia i ustępowanie gatunków do ostoi itd.); zmiany
13 Rekonstrukcja filogenezy 497 klimatyczne powodują wymieranie starych gatunków, a także powstawanie nowych. W obydwu przypadkach często nie mamy wiedzy paleontologicznej na temat warunków, w jakich występował, lub cech, jakie posiadał przodek badanych gatunków. Jeśli umiemy odpowiednio zakodować cechy, w tym ekologiczne, oraz wybrać odpowiedni model zmian wzdłuż gałęzi drzewa filogenetycznego, to można taką rekonstrukcję przeprowadzić. Pozwoli ona na ustalenie, ile razy i w którym momencie nastąpiło przejście do innych warunków ekologicznych. Na przykład, Hardy i Linder (2005) wykorzystując kilka metod, zrekonstruowali najbardziej prawdopodobne warunki ekologiczne, w jakich żył przodek rodzaju Thamnochortus z Afryki Południowej. Okazało się, że żył on w typie siedliska, jakie występuje dzisiaj w południowo-zachodniej, górzystej części florystycznego regionu przylądkowego w Afryce Południowej, a jego potomkowie skolonizowali siedliska o niższej amplitudzie opadów atmosferycznych i położone niżej, przystosowali się także do większego spektrum warunków glebowych. W POSZUKIWANIU DRZEWA ŻYCIA Rozwój metod molekularnych, w tym wysoko wydajnego sekwencjonowania, stwarzają filogenetyce molekularnej nowe, niezwykłe możliwości. Narodziła się filogenomika analizująca nie poszczególne sekwencje, ale całe genomy, np. mitochondrialne albo chloroplastowe. Dużym osiągnięciem było zsekwencjonowanie kompletnego genomu mitochondrialnego neandertalczyka oraz porównanie go z genomami współczesnych ludzi (Green i współaut. 2008). Pozwoliło to na oszacowanie czasu rozejścia się Homo sapiens i Homo neanderthalensis na 660 ± 140 tys. lat temu znacznie dokładniejsze i z mniejszym błędem niż poprzednie oszacowania, bazujące na pojedynczych sekwencjach. Warto zauważyć, że sygnał filogenetyczny zawarty w genomach to nie tylko sekwencje poszczególnych odcinków, ale także informacja o zmianach strukturalnych o duplikacjach i utracie genów, zmianach ich położenia, fuzjach, transferze poziomym itd. Niekwestionowane sukcesy filogenetyki molekularnej skłaniają do zadania pytania, czy poznamy kiedyś kompletne drzewo życia. Pomijając fakt, że nie znamy jeszcze wszystkich gatunków żyjących na Ziemi, a wiele z nich wyginie, zanim je opiszemy, to jest to przedsięwzięcie możliwe do wykonania. Pamiętajmy jednak, że będzie to drzewo przybliżone, albowiem jak to już zaznaczyliśmy nie zawsze w materiale genetycznym organizmów zachował się czytelny sygnał filogenetyczny, a metody rekonstrukcji filogenezy niekiedy zawodzą. Tym niemniej, warto próbować. PHYLOGENY ESTIMATION AND PHYLOGENETIC INFERENCE IN EVOLUTIONARY STUDIES Summary Modern phylogenetics, although rooted in Darwin s and Haeckel s ideas on evolutionary relationships among organisms, dates back to the second half of the 20 th century and the advance of numerical methods in taxonomy. Its beginnings were marked by a fierce debate between phenetics and cladistics but at present it incorporates a diverse array of methods including those based on distance and clustering algorithms, parsimony, maximum likelihood and Bayesian statistics. The phylogeny of extant organisms is usually inferred using molecular markers, because they are genetic, less arbitrary (do not require arbitrary coding), more additive, less prone to convergence and more universal than traditional morphological markers. Phylogenies inferred using molecular data are usually more stable and have better internal support than those obtained from morphology. However, the informed user of phylogenetics methods must be aware of their assumptions and caveats. The chosen sequences must be orthologous (resulting from a speciation event), as opposed to paralogous (resulting from a duplication event); choosing orthologous sequences does not guarantee that the phylogenetic signal is undisturbed. Reversals, multiple hits and parallel substitutions may result in a higher similarity of sequences than expected from their evolutionary history and therefore affect the phylogenetic reconstructions. Moreover, trees inferred from molecular data are usually gene trees rather than species trees. There are several processes that may result in discordance between a gene tree and an organism tree including interspecific hybridisation, horizontal gene transfer, incomplete lineage sorting and selection for allele polymorphism. The most commonly used phylogenetic methods include those based on parsimony, distance and clustering, maximum likelihood and Bayesian statistics. The last three employ nucleotide
14 498 Krzysztof Spalik, Marcin Piwczyński substitution models. Each method is based on certain evolutionary assumptions that may not necessarily apply to a given data set. Noteworthy are recent advances in methods of inferring divergence times using relaxed molecular clock. In evolutionary biology, molecular phylogenies are widely used in comparative studies, historical biogeography and for analysing character state evolution. LITERATURA Camin J. H., Sokal R. R., A method for deducing branching sequences in phylogeny. Evolution 19, Doolitte R. F., Blomback B., Amino-acid sequence investigations of fibrinopeptides from various mammals: evolutionary implications. Nature 202, Green R. E., Malaspinas A.-s., Krause J., Briggs A. W., Johnson P. L., Uhler C., Meyer M., Good J. M., Maricic T., Stenzel U., Prüfer K., Siebauer M., Burbano H. A., Ronan M., Rothberg J. M., Egholm M., Rudan P., Brajković D., Kućan Z., Gusić I., Wikström M., Laakkonen L., Kelso J., Slatkin M., Pääbo S., A complete Neandertal mitochondrial genome sequence determined by high-throughput sequencing. Cell 134, Grehan J. R., Schwartz J. H., Evolution of the second orangutan: phylogeny and biogeography of hominid origins. J. Biogeograph. doi: / j x. Hardy C. R., Linder H. P., Intraspecific variability and timing in ancestral ecology reconstruction: A test case from the Cape Flora. Systematic Biol. 54, Jukes T. h., Cantor C. R., Evolution of protein molecules. [W:] Mammalian protein metabolism. Munro H. N. (red.). Academic Press, New York, Kimura M., The Neutral Theory of Molecular Evolution. Cambridge University Press, Cambridge. Margoliash E., Primary structure and evolution of cytochrome C. Proc. Natl. Acad. Sci. USA 50, Smith S. A., Donoghue M. J, Rates of molecular evolution are linked to life history in flowering plants. Science 322, Webster A. j., Purvis A., Testing the accuracy of methods for reconstructing ancestral states of continuous characters. Proc. R. Soc. Lond. Series B 269, Zuckerkandl E., Pauling L., Evolutionary divergence and convergence in proteins. [W:] Evolving genes and proteins. Bryson V., Vogel H. J. (red.). Academic Press, New York,
Filogenetyka molekularna I
 2 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498 Filogenetyka molekularna I John C. Avise
2 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498 Filogenetyka molekularna I John C. Avise
Filogenetyka molekularna I. Krzysztof Spalik
 Filogenetyka molekularna I Krzysztof Spalik Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498
Filogenetyka molekularna I Krzysztof Spalik Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498
Filogenetyka molekularna I. Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji
 Filogenetyka molekularna I Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji 3 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w
Filogenetyka molekularna I Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji 3 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w
Genomika Porównawcza. Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski
 Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
Filogenetyka molekularna. Dr Anna Karnkowska Zakład Filogenetyki Molekularnej i Ewolucji
 Filogenetyka molekularna Dr Anna Karnkowska Zakład Filogenetyki Molekularnej i Ewolucji Co to jest filogeneza? Filogeneza=drzewo filogenetyczne=drzewo rodowe=drzewo to rozgałęziający się diagram, który
Filogenetyka molekularna Dr Anna Karnkowska Zakład Filogenetyki Molekularnej i Ewolucji Co to jest filogeneza? Filogeneza=drzewo filogenetyczne=drzewo rodowe=drzewo to rozgałęziający się diagram, który
Konstruowanie drzew filogenetycznych. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Teoria ewolucji. Podstawy wspólne pochodzenie.
 Teoria ewolucji. Podstawy wspólne pochodzenie. Ewolucja biologiczna } Znaczenie ogólne: } proces zmian informacji genetycznej (częstości i rodzaju alleli), } które to zmiany są przekazywane z pokolenia
Teoria ewolucji. Podstawy wspólne pochodzenie. Ewolucja biologiczna } Znaczenie ogólne: } proces zmian informacji genetycznej (częstości i rodzaju alleli), } które to zmiany są przekazywane z pokolenia
Teoria ewolucji. Podstawowe pojęcia. Wspólne pochodzenie.
 Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Ewolucja Znaczenie ogólne: zmiany zachodzące stopniowo w czasie W biologii ewolucja biologiczna W astronomii i kosmologii ewolucja gwiazd i wszechświata
Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Ewolucja Znaczenie ogólne: zmiany zachodzące stopniowo w czasie W biologii ewolucja biologiczna W astronomii i kosmologii ewolucja gwiazd i wszechświata
Urszula Poziomek, doradca metodyczny w zakresie biologii Materiał dydaktyczny przygotowany na konferencję z cyklu Na miarę Nobla, 14 stycznia 2010 r.
 Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Acknowledgement. Drzewa filogenetyczne
 Wykład 8 Drzewa Filogenetyczne Lokalizacja genów Some figures from: Acknowledgement M. Zvelebil, J.O. Baum, Introduction to Bioinformatics, Garland Science 2008 Tradycyjne drzewa pokrewieństwa Drzewa oparte
Wykład 8 Drzewa Filogenetyczne Lokalizacja genów Some figures from: Acknowledgement M. Zvelebil, J.O. Baum, Introduction to Bioinformatics, Garland Science 2008 Tradycyjne drzewa pokrewieństwa Drzewa oparte
Ewolucjonizm NEODARWINIZM. Dr Jacek Francikowski Uniwersyteckie Towarzystwo Naukowe Uniwersytet Śląski w Katowicach
 Ewolucjonizm NEODARWINIZM Dr Jacek Francikowski Uniwersyteckie Towarzystwo Naukowe Uniwersytet Śląski w Katowicach Główne paradygmaty biologii Wspólne początki życia Komórka jako podstawowo jednostka funkcjonalna
Ewolucjonizm NEODARWINIZM Dr Jacek Francikowski Uniwersyteckie Towarzystwo Naukowe Uniwersytet Śląski w Katowicach Główne paradygmaty biologii Wspólne początki życia Komórka jako podstawowo jednostka funkcjonalna
Dopasowanie sekwencji (sequence alignment)
 Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Co to jest alignment? Dopasowanie sekwencji (sequence alignment) Alignment jest sposobem dopasowania struktur pierwszorzędowych DNA, RNA lub białek do zidentyfikowanych regionów w celu określenia podobieństwa;
Teoria ewolucji. Podstawowe pojęcia. Wspólne pochodzenie.
 Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego 5A pgolik@igib.uw.edu.pl Informacje, materiały: http://www.igib.uw.edu.pl/
Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego 5A pgolik@igib.uw.edu.pl Informacje, materiały: http://www.igib.uw.edu.pl/
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Filogenetyka molekularna wykorzystuje informację zawartą w sekwencjach aminokwasów lub nukleotydów do kontrukcji drzew
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Filogenetyka molekularna wykorzystuje informację zawartą w sekwencjach aminokwasów lub nukleotydów do kontrukcji drzew
Badanie doboru naturalnego na poziomie molekularnym
 Badanie doboru naturalnego na poziomie molekularnym Podstawy ewolucji molekulanej Jak ewoluują sekwencje Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na
Badanie doboru naturalnego na poziomie molekularnym Podstawy ewolucji molekulanej Jak ewoluują sekwencje Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na
Mechanizmy zmienności ewolucyjnej. Podstawy ewolucji molekularnej.
 Mechanizmy zmienności ewolucyjnej Podstawy ewolucji molekularnej. Mechanizmy ewolucji } Generujące zmienność } mutacje } rearanżacje genomu } horyzontalny transfer genów } Działające na warianty wytworzone
Mechanizmy zmienności ewolucyjnej Podstawy ewolucji molekularnej. Mechanizmy ewolucji } Generujące zmienność } mutacje } rearanżacje genomu } horyzontalny transfer genów } Działające na warianty wytworzone
Ograniczenia środowiskowe nie budzą wielu kontrowersji, co nie znaczy że rozumiemy do końca proces powstawania adaptacji fizjologicznych.
 1 Ograniczenia środowiskowe nie budzą wielu kontrowersji, co nie znaczy że rozumiemy do końca proces powstawania adaptacji fizjologicznych. Wiadomo, że ściśle powiązane z zagadnieniem interakcji kompetencje
1 Ograniczenia środowiskowe nie budzą wielu kontrowersji, co nie znaczy że rozumiemy do końca proces powstawania adaptacji fizjologicznych. Wiadomo, że ściśle powiązane z zagadnieniem interakcji kompetencje
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
Zmienność ewolucyjna. Ewolucja molekularna
 Zmienność ewolucyjna Ewolucja molekularna Mechanizmy ewolucji Generujące zmienność mutacje rearanżacje genomu horyzontalny transfer genów! Działające na warianty wytworzone przez zmienność dobór naturalny
Zmienność ewolucyjna Ewolucja molekularna Mechanizmy ewolucji Generujące zmienność mutacje rearanżacje genomu horyzontalny transfer genów! Działające na warianty wytworzone przez zmienność dobór naturalny
Analizy filogenetyczne
 BIOINFORMATYKA edycja 2016 / 2017 wykład 6 Analizy filogenetyczne dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Cele i zastosowania 2. Podstawy ewolucyjne
BIOINFORMATYKA edycja 2016 / 2017 wykład 6 Analizy filogenetyczne dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Cele i zastosowania 2. Podstawy ewolucyjne
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Często dopasować chcemy nie dwie sekwencje ale kilkanaście lub więcej 2 Istnieją dokładne algorytmy, lecz są one niewydajne
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Często dopasować chcemy nie dwie sekwencje ale kilkanaście lub więcej 2 Istnieją dokładne algorytmy, lecz są one niewydajne
Wykład Bioinformatyka 2012-09-24. Bioinformatyka. Wykład 7. E. Banachowicz. Zakład Biofizyki Molekularnej IF UAM. Ewolucyjne podstawy Bioinformatyki
 Bioinformatyka Wykład 7 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Plan Bioinformatyka Ewolucyjne podstawy Bioinformatyki Filogenetyka Bioinformatyczne narzędzia
Bioinformatyka Wykład 7 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Plan Bioinformatyka Ewolucyjne podstawy Bioinformatyki Filogenetyka Bioinformatyczne narzędzia
plezjomorfie: podobieństwa dziedziczone po dalszych przodkach (c. atawistyczna)
 Podobieństwa pomiędzy organizmami - cechy homologiczne: podobieństwa wynikające z dziedziczenia - apomorfie: podobieństwa dziedziczone po najbliższym przodku lub pojawiająca się de novo (c. ewolucyjnie
Podobieństwa pomiędzy organizmami - cechy homologiczne: podobieństwa wynikające z dziedziczenia - apomorfie: podobieństwa dziedziczone po najbliższym przodku lub pojawiająca się de novo (c. ewolucyjnie
Mitochondrialna Ewa;
 Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Wnioskowanie bayesowskie
 Wnioskowanie bayesowskie W podejściu klasycznym wnioskowanie statystyczne oparte jest wyłącznie na podstawie pobranej próby losowej. Możemy np. estymować punktowo lub przedziałowo nieznane parametry rozkładów,
Wnioskowanie bayesowskie W podejściu klasycznym wnioskowanie statystyczne oparte jest wyłącznie na podstawie pobranej próby losowej. Możemy np. estymować punktowo lub przedziałowo nieznane parametry rozkładów,
Podstawy biologii. Informacja genetyczna. Co to jest ewolucja.
 Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Materiał genetyczny Materiałem genetycznym są kwasy nukleinowe Materiałem genetycznym organizmów komórkowych jest kwas deoksyrybonukleinowy
Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Materiał genetyczny Materiałem genetycznym są kwasy nukleinowe Materiałem genetycznym organizmów komórkowych jest kwas deoksyrybonukleinowy
Algorytm genetyczny (genetic algorithm)-
 Optymalizacja W praktyce inżynierskiej często zachodzi potrzeba znalezienia parametrów, dla których system/urządzenie będzie działać w sposób optymalny. Klasyczne podejście do optymalizacji: sformułowanie
Optymalizacja W praktyce inżynierskiej często zachodzi potrzeba znalezienia parametrów, dla których system/urządzenie będzie działać w sposób optymalny. Klasyczne podejście do optymalizacji: sformułowanie
Budowanie drzewa filogenetycznego
 Szkoła Festiwalu Nauki 134567 Wojciech Grajkowski Szkoła Festiwalu Nauki, ul. Ks. Trojdena 4, 02-109 Warszawa www.sfn.edu.pl sfn@iimcb.gov.pl Budowanie drzewa filogenetycznego Cel Ćwiczenie polega na budowaniu
Szkoła Festiwalu Nauki 134567 Wojciech Grajkowski Szkoła Festiwalu Nauki, ul. Ks. Trojdena 4, 02-109 Warszawa www.sfn.edu.pl sfn@iimcb.gov.pl Budowanie drzewa filogenetycznego Cel Ćwiczenie polega na budowaniu
Jak powstają nowe gatunki. Katarzyna Gontek
 Jak powstają nowe gatunki Katarzyna Gontek Powstawanie gatunków (specjacja) to proces biologiczny, w wyniku którego powstają nowe gatunki organizmów. Zachodzi na skutek wytworzenia się bariery rozrodczej
Jak powstają nowe gatunki Katarzyna Gontek Powstawanie gatunków (specjacja) to proces biologiczny, w wyniku którego powstają nowe gatunki organizmów. Zachodzi na skutek wytworzenia się bariery rozrodczej
Spis treści. Przedmowa... XI. Wprowadzenie i biologiczne bazy danych. 1 Wprowadzenie... 3. 2 Wprowadzenie do biologicznych baz danych...
 Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Przedmowa... XI Część pierwsza Wprowadzenie i biologiczne bazy danych 1 Wprowadzenie... 3 Czym jest bioinformatyka?... 5 Cele... 5 Zakres zainteresowań... 6 Zastosowania... 7 Ograniczenia... 8 Przyszłe
Nuttall przeprowadził testy precypitacyjne białek surowicy, aby wykazać związek filogenetyczny między różnymi grupami zwierząt.
 1904 Nuttall przeprowadził testy precypitacyjne białek surowicy, aby wykazać związek filogenetyczny między różnymi grupami zwierząt. M. Prakash 2007.Encyclopaedia of Gene Evolution Vol. 2, Molecular Genetics,
1904 Nuttall przeprowadził testy precypitacyjne białek surowicy, aby wykazać związek filogenetyczny między różnymi grupami zwierząt. M. Prakash 2007.Encyclopaedia of Gene Evolution Vol. 2, Molecular Genetics,
Statystyka i opracowanie danych Podstawy wnioskowania statystycznego. Prawo wielkich liczb. Centralne twierdzenie graniczne. Estymacja i estymatory
 Statystyka i opracowanie danych Podstawy wnioskowania statystycznego. Prawo wielkich liczb. Centralne twierdzenie graniczne. Estymacja i estymatory Dr Anna ADRIAN Paw B5, pok 407 adrian@tempus.metal.agh.edu.pl
Statystyka i opracowanie danych Podstawy wnioskowania statystycznego. Prawo wielkich liczb. Centralne twierdzenie graniczne. Estymacja i estymatory Dr Anna ADRIAN Paw B5, pok 407 adrian@tempus.metal.agh.edu.pl
Teoria ewolucji. Losy gatunków: specjacja i wymieranie. Podstawy ewolucji molekularnej
 Teoria ewolucji. Losy gatunków: specjacja i wymieranie. Podstawy ewolucji molekularnej Specjacja } Pojawienie się bariery reprodukcyjnej między populacjami dające początek gatunkom } Specjacja allopatryczna
Teoria ewolucji. Losy gatunków: specjacja i wymieranie. Podstawy ewolucji molekularnej Specjacja } Pojawienie się bariery reprodukcyjnej między populacjami dające początek gatunkom } Specjacja allopatryczna
Podstawy ewolucji molekularnej. Ewolucja sekwencji DNA i białek
 Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji } Mutacje } tworzą nowe allele genów } Inwersje } zmieniają układ genów na chromosomach } mogą uniemożliwić rekombinację
Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji } Mutacje } tworzą nowe allele genów } Inwersje } zmieniają układ genów na chromosomach } mogą uniemożliwić rekombinację
Porównywanie i dopasowywanie sekwencji
 Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
Wykład Centralne twierdzenie graniczne. Statystyka matematyczna: Estymacja parametrów rozkładu
 Wykład 11-12 Centralne twierdzenie graniczne Statystyka matematyczna: Estymacja parametrów rozkładu Centralne twierdzenie graniczne (CTG) (Central Limit Theorem - CLT) Centralne twierdzenie graniczne (Lindenberga-Levy'ego)
Wykład 11-12 Centralne twierdzenie graniczne Statystyka matematyczna: Estymacja parametrów rozkładu Centralne twierdzenie graniczne (CTG) (Central Limit Theorem - CLT) Centralne twierdzenie graniczne (Lindenberga-Levy'ego)
Podstawy ewolucji molekularnej. Ewolucja sekwencji DNA i białek
 Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na chromosomach mogą uniemożliwić rekombinację
Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na chromosomach mogą uniemożliwić rekombinację
Podstawy biologii. Informacja genetyczna. Co to jest ewolucja.
 Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Zarys biologii molekularnej genu Podstawowe procesy genetyczne Replikacja powielanie informacji Ekspresja wyrażanie (realizowanie funkcji)
Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Zarys biologii molekularnej genu Podstawowe procesy genetyczne Replikacja powielanie informacji Ekspresja wyrażanie (realizowanie funkcji)
Recenzja rozprawy doktorskiej. mgr Marcina Jana Kamińskiego. pt. Grupa rodzajowa Ectateus (Coleoptera: Tenebrionidae) filogeneza i klasyfikacja.
 Dr hab. Grzegorz Paśnik Instytut Systematyki i Ewolucji Zwierząt Polska Akademia Nauk ul. Sławkowska 17, 31-016 Kraków Recenzja rozprawy doktorskiej mgr Marcina Jana Kamińskiego pt. Grupa rodzajowa Ectateus
Dr hab. Grzegorz Paśnik Instytut Systematyki i Ewolucji Zwierząt Polska Akademia Nauk ul. Sławkowska 17, 31-016 Kraków Recenzja rozprawy doktorskiej mgr Marcina Jana Kamińskiego pt. Grupa rodzajowa Ectateus
klasyfikacja fenetyczna (numeryczna)
 Teorie klasyfikacji klasyfikacja fenetyczna (numeryczna) systematyka powinna być wolna od wszelkiej teorii (a zwłaszcza od teorii ewolucji) filogeneza jako ciąg zdarzeń jest niepoznawalna opiera się na
Teorie klasyfikacji klasyfikacja fenetyczna (numeryczna) systematyka powinna być wolna od wszelkiej teorii (a zwłaszcza od teorii ewolucji) filogeneza jako ciąg zdarzeń jest niepoznawalna opiera się na
Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna
 Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna Spis treści Przedmowa................................. Podziękowania............................... XIII XIV 1 Metody genetyki molekularnej w badaniach
Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna Spis treści Przedmowa................................. Podziękowania............................... XIII XIV 1 Metody genetyki molekularnej w badaniach
Ekologia wyk. 1. wiedza z zakresu zarówno matematyki, biologii, fizyki, chemii, rozumienia modeli matematycznych
 Ekologia wyk. 1 wiedza z zakresu zarówno matematyki, biologii, fizyki, chemii, rozumienia modeli matematycznych Ochrona środowiska Ekologia jako dziedzina nauki jest nauką o zależnościach decydujących
Ekologia wyk. 1 wiedza z zakresu zarówno matematyki, biologii, fizyki, chemii, rozumienia modeli matematycznych Ochrona środowiska Ekologia jako dziedzina nauki jest nauką o zależnościach decydujących
Klasyfikatory: k-nn oraz naiwny Bayesa. Agnieszka Nowak Brzezińska Wykład IV
 Klasyfikatory: k-nn oraz naiwny Bayesa Agnieszka Nowak Brzezińska Wykład IV Naiwny klasyfikator Bayesa Naiwny klasyfikator bayesowski jest prostym probabilistycznym klasyfikatorem. Zakłada się wzajemną
Klasyfikatory: k-nn oraz naiwny Bayesa Agnieszka Nowak Brzezińska Wykład IV Naiwny klasyfikator Bayesa Naiwny klasyfikator bayesowski jest prostym probabilistycznym klasyfikatorem. Zakłada się wzajemną
Spis treści. Przedmowa... XI. Rozdział 1. Pomiar: jednostki miar... 1. Rozdział 2. Pomiar: liczby i obliczenia liczbowe... 16
 Spis treści Przedmowa.......................... XI Rozdział 1. Pomiar: jednostki miar................. 1 1.1. Wielkości fizyczne i pozafizyczne.................. 1 1.2. Spójne układy miar. Układ SI i jego
Spis treści Przedmowa.......................... XI Rozdział 1. Pomiar: jednostki miar................. 1 1.1. Wielkości fizyczne i pozafizyczne.................. 1 1.2. Spójne układy miar. Układ SI i jego
Prawdopodobieństwo warunkowe Twierdzenie o prawdopodobieństwie całkowitym
 Edward Stachowski Prawdopodobieństwo warunkowe Twierdzenie o prawdopodobieństwie całkowitym W podstawie programowej obowiązującej na egzaminie maturalnym od 05r pojawiły się nowe treści programowe Wśród
Edward Stachowski Prawdopodobieństwo warunkowe Twierdzenie o prawdopodobieństwie całkowitym W podstawie programowej obowiązującej na egzaminie maturalnym od 05r pojawiły się nowe treści programowe Wśród
E: Rekonstrukcja ewolucji. Algorytmy filogenetyczne
 E: Rekonstrukcja ewolucji. Algorytmy filogenetyczne Przypominajka: 152 drzewo filogenetyczne to drzewo, którego liśćmi są istniejące gatunki, a węzły wewnętrzne mają stopień większy niż jeden i reprezentują
E: Rekonstrukcja ewolucji. Algorytmy filogenetyczne Przypominajka: 152 drzewo filogenetyczne to drzewo, którego liśćmi są istniejące gatunki, a węzły wewnętrzne mają stopień większy niż jeden i reprezentują
Analiza składowych głównych. Wprowadzenie
 Wprowadzenie jest techniką redukcji wymiaru. Składowe główne zostały po raz pierwszy zaproponowane przez Pearsona(1901), a następnie rozwinięte przez Hotellinga (1933). jest zaliczana do systemów uczących
Wprowadzenie jest techniką redukcji wymiaru. Składowe główne zostały po raz pierwszy zaproponowane przez Pearsona(1901), a następnie rozwinięte przez Hotellinga (1933). jest zaliczana do systemów uczących
ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH
 1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
1 ALGORYTMICZNA I STATYSTYCZNA ANALIZA DANYCH WFAiS UJ, Informatyka Stosowana II stopień studiów 2 Wnioskowanie statystyczne dla zmiennych numerycznych Porównywanie dwóch średnich Boot-strapping Analiza
Algorytm Genetyczny. zastosowanie do procesów rozmieszczenia stacji raportujących w sieciach komórkowych
 Algorytm Genetyczny zastosowanie do procesów rozmieszczenia stacji raportujących w sieciach komórkowych Dlaczego Algorytmy Inspirowane Naturą? Rozwój nowych technologii: złożone problemy obliczeniowe w
Algorytm Genetyczny zastosowanie do procesów rozmieszczenia stacji raportujących w sieciach komórkowych Dlaczego Algorytmy Inspirowane Naturą? Rozwój nowych technologii: złożone problemy obliczeniowe w
Agnieszka Nowak Brzezińska Wykład III
 Agnieszka Nowak Brzezińska Wykład III Naiwny klasyfikator bayesowski jest prostym probabilistycznym klasyfikatorem. Zakłada się wzajemną niezależność zmiennych niezależnych (tu naiwność) Bardziej opisowe
Agnieszka Nowak Brzezińska Wykład III Naiwny klasyfikator bayesowski jest prostym probabilistycznym klasyfikatorem. Zakłada się wzajemną niezależność zmiennych niezależnych (tu naiwność) Bardziej opisowe
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii
 Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Możliwości współczesnej inżynierii genetycznej w obszarze biotechnologii 1. Technologia rekombinowanego DNA jest podstawą uzyskiwania genetycznie zmodyfikowanych organizmów 2. Medycyna i ochrona zdrowia
Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi.
 Ryciny 193 Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi. Na fioletowo zaznaczone zostały populacje (nr 1 14)
Ryciny 193 Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi. Na fioletowo zaznaczone zostały populacje (nr 1 14)
Algorytmy ewolucyjne NAZEWNICTWO
 Algorytmy ewolucyjne http://zajecia.jakubw.pl/nai NAZEWNICTWO Algorytmy ewolucyjne nazwa ogólna, obejmująca metody szczegółowe, jak np.: algorytmy genetyczne programowanie genetyczne strategie ewolucyjne
Algorytmy ewolucyjne http://zajecia.jakubw.pl/nai NAZEWNICTWO Algorytmy ewolucyjne nazwa ogólna, obejmująca metody szczegółowe, jak np.: algorytmy genetyczne programowanie genetyczne strategie ewolucyjne
Agnieszka Nowak Brzezińska Wykład III
 Agnieszka Nowak Brzezińska Wykład III Naiwny klasyfikator bayesowski jest prostym probabilistycznym klasyfikatorem. Zakłada się wzajemną niezależność zmiennych niezależnych (tu naiwność) Bardziej opisowe
Agnieszka Nowak Brzezińska Wykład III Naiwny klasyfikator bayesowski jest prostym probabilistycznym klasyfikatorem. Zakłada się wzajemną niezależność zmiennych niezależnych (tu naiwność) Bardziej opisowe
o cechach dziedziczonych decyduje środowisko, a gatunki mogą łatwo i spontanicznie przechodzić jedne w drugie
 Iwan Miczurin (1855-1935) Trofim Denisowicz Łysenko (1898-1976) przy interwencji człowieka możliwe jest zmuszenie każdej formy zwierzęcia lub rośliny do znacznie szybszych zmian, w kierunku pożądanym przez
Iwan Miczurin (1855-1935) Trofim Denisowicz Łysenko (1898-1976) przy interwencji człowieka możliwe jest zmuszenie każdej formy zwierzęcia lub rośliny do znacznie szybszych zmian, w kierunku pożądanym przez
Szczegółowy program kursu Statystyka z programem Excel (30 godzin lekcyjnych zajęć)
 Szczegółowy program kursu Statystyka z programem Excel (30 godzin lekcyjnych zajęć) 1. Populacja generalna a losowa próba, parametr rozkładu cechy a jego ocena z losowej próby, miary opisu statystycznego
Szczegółowy program kursu Statystyka z programem Excel (30 godzin lekcyjnych zajęć) 1. Populacja generalna a losowa próba, parametr rozkładu cechy a jego ocena z losowej próby, miary opisu statystycznego
Mikroekonometria 6. Mikołaj Czajkowski Wiktor Budziński
 Mikroekonometria 6 Mikołaj Czajkowski Wiktor Budziński Metody symulacyjne Monte Carlo Metoda Monte-Carlo Wykorzystanie mocy obliczeniowej komputerów, aby poznać charakterystyki zmiennych losowych poprzez
Mikroekonometria 6 Mikołaj Czajkowski Wiktor Budziński Metody symulacyjne Monte Carlo Metoda Monte-Carlo Wykorzystanie mocy obliczeniowej komputerów, aby poznać charakterystyki zmiennych losowych poprzez
Wszystkie wyniki w postaci ułamków należy podawać z dokładnością do czterech miejsc po przecinku!
 Pracownia statystyczno-filogenetyczna Liczba punktów (wypełnia KGOB) / 30 PESEL Imię i nazwisko Grupa Nr Czas: 90 min. Łączna liczba punktów do zdobycia: 30 Czerwona Niebieska Zielona Żółta Zaznacz znakiem
Pracownia statystyczno-filogenetyczna Liczba punktów (wypełnia KGOB) / 30 PESEL Imię i nazwisko Grupa Nr Czas: 90 min. Łączna liczba punktów do zdobycia: 30 Czerwona Niebieska Zielona Żółta Zaznacz znakiem
Statystyki: miary opisujące rozkład! np. : średnia, frakcja (procent), odchylenie standardowe, wariancja, mediana itd.
 Wnioskowanie statystyczne obejmujące metody pozwalające na uogólnianie wyników z próby na nieznane wartości parametrów oraz szacowanie błędów tego uogólnienia. Przewidujemy nieznaną wartości parametru
Wnioskowanie statystyczne obejmujące metody pozwalające na uogólnianie wyników z próby na nieznane wartości parametrów oraz szacowanie błędów tego uogólnienia. Przewidujemy nieznaną wartości parametru
Sposoby prezentacji problemów w statystyce
 S t r o n a 1 Dr Anna Rybak Instytut Informatyki Uniwersytet w Białymstoku Sposoby prezentacji problemów w statystyce Wprowadzenie W artykule zostaną zaprezentowane podstawowe zagadnienia z zakresu statystyki
S t r o n a 1 Dr Anna Rybak Instytut Informatyki Uniwersytet w Białymstoku Sposoby prezentacji problemów w statystyce Wprowadzenie W artykule zostaną zaprezentowane podstawowe zagadnienia z zakresu statystyki
Algorytmy genetyczne
 Algorytmy genetyczne Motto: Zamiast pracowicie poszukiwać najlepszego rozwiązania problemu informatycznego lepiej pozwolić, żeby komputer sam sobie to rozwiązanie wyhodował! Algorytmy genetyczne służą
Algorytmy genetyczne Motto: Zamiast pracowicie poszukiwać najlepszego rozwiązania problemu informatycznego lepiej pozwolić, żeby komputer sam sobie to rozwiązanie wyhodował! Algorytmy genetyczne służą
Statystyki: miary opisujące rozkład! np. : średnia, frakcja (procent), odchylenie standardowe, wariancja, mediana itd.
 Wnioskowanie statystyczne obejmujące metody pozwalające na uogólnianie wyników z próby na nieznane wartości parametrów oraz szacowanie błędów tego uogólnienia. Przewidujemy nieznaną wartości parametru
Wnioskowanie statystyczne obejmujące metody pozwalające na uogólnianie wyników z próby na nieznane wartości parametrów oraz szacowanie błędów tego uogólnienia. Przewidujemy nieznaną wartości parametru
Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej
 Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Mikroekonometria 5. Mikołaj Czajkowski Wiktor Budziński
 Mikroekonometria 5 Mikołaj Czajkowski Wiktor Budziński Zadanie 1. Wykorzystując dane me.medexp3.dta przygotuj model regresji kwantylowej 1. Przygotuj model regresji kwantylowej w którym logarytm wydatków
Mikroekonometria 5 Mikołaj Czajkowski Wiktor Budziński Zadanie 1. Wykorzystując dane me.medexp3.dta przygotuj model regresji kwantylowej 1. Przygotuj model regresji kwantylowej w którym logarytm wydatków
166 Wstęp do statystyki matematycznej
 166 Wstęp do statystyki matematycznej Etap trzeci realizacji procesu analizy danych statystycznych w zasadzie powinien rozwiązać nasz zasadniczy problem związany z identyfikacją cechy populacji generalnej
166 Wstęp do statystyki matematycznej Etap trzeci realizacji procesu analizy danych statystycznych w zasadzie powinien rozwiązać nasz zasadniczy problem związany z identyfikacją cechy populacji generalnej
Uczeń potrafi. Dział Rozdział Temat lekcji
 Plan wynikowy z biologii- zakres podstawowy, dla klasy III LO i III i IV Technikum LO im.ks. Jerzego Popiełuszki oraz Technikum w Suchowoli Nauczyciel: Katarzyna Kotiuk Nr programu: DKOS-4015-5/02 Dział
Plan wynikowy z biologii- zakres podstawowy, dla klasy III LO i III i IV Technikum LO im.ks. Jerzego Popiełuszki oraz Technikum w Suchowoli Nauczyciel: Katarzyna Kotiuk Nr programu: DKOS-4015-5/02 Dział
Zmienność. środa, 23 listopada 11
 Zmienność http://ggoralski.com Zmienność Zmienność - rodzaje Zmienność obserwuje się zarówno między poszczególnymi osobnikami jak i między populacjami. Różnice te mogą mieć jednak różne podłoże. Mogą one
Zmienność http://ggoralski.com Zmienność Zmienność - rodzaje Zmienność obserwuje się zarówno między poszczególnymi osobnikami jak i między populacjami. Różnice te mogą mieć jednak różne podłoże. Mogą one
Ewolucja człowieka. Ostatnie 5 milionów lat
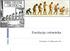 Ewolucja człowieka Ostatnie 5 milionów lat 1 Złożone zagadki } } Odnaleziono wiele skamieniałości naczelnych, różne gatunki w tym samym czasie Trudno ustalić relacje między nimi } Przodkowie, czy boczne
Ewolucja człowieka Ostatnie 5 milionów lat 1 Złożone zagadki } } Odnaleziono wiele skamieniałości naczelnych, różne gatunki w tym samym czasie Trudno ustalić relacje między nimi } Przodkowie, czy boczne
Testowanie hipotez statystycznych. Wnioskowanie statystyczne
 Testowanie hipotez statystycznych Wnioskowanie statystyczne Hipoteza statystyczna to dowolne przypuszczenie co do rozkładu populacji generalnej (jego postaci funkcyjnej lub wartości parametrów). Hipotezy
Testowanie hipotez statystycznych Wnioskowanie statystyczne Hipoteza statystyczna to dowolne przypuszczenie co do rozkładu populacji generalnej (jego postaci funkcyjnej lub wartości parametrów). Hipotezy
Zadania ze statystyki cz. 8 I rok socjologii. Zadanie 1.
 Zadania ze statystyki cz. 8 I rok socjologii Zadanie 1. W potocznej opinii pokutuje przekonanie, że lepsi z matematyki są chłopcy niż dziewczęta. Chcąc zweryfikować tę opinię, przeprowadzono badanie w
Zadania ze statystyki cz. 8 I rok socjologii Zadanie 1. W potocznej opinii pokutuje przekonanie, że lepsi z matematyki są chłopcy niż dziewczęta. Chcąc zweryfikować tę opinię, przeprowadzono badanie w
Biologia medyczna, materiały dla studentów
 Jaka tam ewolucja. Zanim trafię na jednego myślącego, muszę stoczyć bitwę zdziewięcioma orangutanami Carlos Ruis Zafon Wierzbownica drobnokwiatowa Fitosterole, garbniki, flawonoidy Właściwości przeciwzapalne,
Jaka tam ewolucja. Zanim trafię na jednego myślącego, muszę stoczyć bitwę zdziewięcioma orangutanami Carlos Ruis Zafon Wierzbownica drobnokwiatowa Fitosterole, garbniki, flawonoidy Właściwości przeciwzapalne,
Metody probabilistyczne klasyfikatory bayesowskie
 Konwersatorium Matematyczne Metody Ekonomii narzędzia matematyczne w eksploracji danych First Prev Next Last Go Back Full Screen Close Quit Metody probabilistyczne klasyfikatory bayesowskie Wykład 8 Marcin
Konwersatorium Matematyczne Metody Ekonomii narzędzia matematyczne w eksploracji danych First Prev Next Last Go Back Full Screen Close Quit Metody probabilistyczne klasyfikatory bayesowskie Wykład 8 Marcin
Kontekstowe wskaźniki efektywności nauczania - warsztaty
 Kontekstowe wskaźniki efektywności nauczania - warsztaty Przygotowała: Aleksandra Jasińska (a.jasinska@ibe.edu.pl) wykorzystując materiały Zespołu EWD Czy dobrze uczymy? Metody oceny efektywności nauczania
Kontekstowe wskaźniki efektywności nauczania - warsztaty Przygotowała: Aleksandra Jasińska (a.jasinska@ibe.edu.pl) wykorzystując materiały Zespołu EWD Czy dobrze uczymy? Metody oceny efektywności nauczania
Dobór parametrów algorytmu ewolucyjnego
 Dobór parametrów algorytmu ewolucyjnego 1 2 Wstęp Algorytm ewolucyjny posiada wiele parametrów. Przykładowo dla algorytmu genetycznego są to: prawdopodobieństwa stosowania operatorów mutacji i krzyżowania.
Dobór parametrów algorytmu ewolucyjnego 1 2 Wstęp Algorytm ewolucyjny posiada wiele parametrów. Przykładowo dla algorytmu genetycznego są to: prawdopodobieństwa stosowania operatorów mutacji i krzyżowania.
Podstawy ewolucji molekularnej. Ewolucja sekwencji DNA i białek
 Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Egzamin: 29.01.2018 16:00, sala 9B Pierwsza synteza Ewolucja jako zmiany częstości alleli w populacji Mutacje jako źródło nowych alleli Dobór
Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Egzamin: 29.01.2018 16:00, sala 9B Pierwsza synteza Ewolucja jako zmiany częstości alleli w populacji Mutacje jako źródło nowych alleli Dobór
PRZYRÓWNANIE SEKWENCJI
 http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO
 SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO. Rzeczywistość (istniejąca lub projektowana).. Model fizyczny. 3. Model matematyczny (optymalizacyjny): a. Zmienne projektowania
SCHEMAT ROZWIĄZANIA ZADANIA OPTYMALIZACJI PRZY POMOCY ALGORYTMU GENETYCZNEGO. Rzeczywistość (istniejąca lub projektowana).. Model fizyczny. 3. Model matematyczny (optymalizacyjny): a. Zmienne projektowania
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych???
 Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Analizy DNA in silico - czyli czego można szukać i co można znaleźć w sekwencjach nukleotydowych??? Alfabet kwasów nukleinowych jest stosunkowo ubogi!!! Dla sekwencji DNA (RNA) stosuje się zasadniczo*
Rachunek prawdopodobieństwa- wykład 2
 Rachunek prawdopodobieństwa- wykład 2 Pojęcie dyskretnej przestrzeni probabilistycznej i określenie prawdopodobieństwa w tej przestrzeni dr Marcin Ziółkowski Instytut Matematyki i Informatyki Uniwersytet
Rachunek prawdopodobieństwa- wykład 2 Pojęcie dyskretnej przestrzeni probabilistycznej i określenie prawdopodobieństwa w tej przestrzeni dr Marcin Ziółkowski Instytut Matematyki i Informatyki Uniwersytet
Zbigniew S. Szewczak Uniwersytet Mikołaja Kopernika Wydział Matematyki i Informatyki. Graniczne własności łańcuchów Markowa
 Zbigniew S. Szewczak Uniwersytet Mikołaja Kopernika Wydział Matematyki i Informatyki Graniczne własności łańcuchów Markowa Toruń, 2003 Co to jest łańcuch Markowa? Każdy skończony, jednorodny łańcuch Markowa
Zbigniew S. Szewczak Uniwersytet Mikołaja Kopernika Wydział Matematyki i Informatyki Graniczne własności łańcuchów Markowa Toruń, 2003 Co to jest łańcuch Markowa? Każdy skończony, jednorodny łańcuch Markowa
Wstęp do Biologii Obliczeniowej
 Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Adam Kirpsza Zastosowanie regresji logistycznej w studiach nad Unią Europejska. Anna Stankiewicz Izabela Słomska
 Adam Kirpsza Zastosowanie regresji logistycznej w studiach nad Unią Europejska Anna Stankiewicz Izabela Słomska Wstęp- statystyka w politologii Rzadkie stosowanie narzędzi statystycznych Pisma Karla Poppera
Adam Kirpsza Zastosowanie regresji logistycznej w studiach nad Unią Europejska Anna Stankiewicz Izabela Słomska Wstęp- statystyka w politologii Rzadkie stosowanie narzędzi statystycznych Pisma Karla Poppera
Szczegółowy program kursu Statystyka z programem Excel (30 godzin lekcyjnych zajęć)
 Szczegółowy program kursu Statystyka z programem Excel (30 godzin lekcyjnych zajęć) 1. Populacja generalna a losowa próba, parametr rozkładu cechy a jego ocena z losowej próby, miary opisu statystycznego
Szczegółowy program kursu Statystyka z programem Excel (30 godzin lekcyjnych zajęć) 1. Populacja generalna a losowa próba, parametr rozkładu cechy a jego ocena z losowej próby, miary opisu statystycznego
1. Liczby naturalne, podzielność, silnie, reszty z dzielenia
 1. Liczby naturalne, podzielność, silnie, reszty z dzielenia kwadratów i sześcianów przez małe liczby, cechy podzielności przez 2, 4, 8, 5, 25, 125, 3, 9. 26 września 2009 r. Uwaga: Przyjmujemy, że 0 nie
1. Liczby naturalne, podzielność, silnie, reszty z dzielenia kwadratów i sześcianów przez małe liczby, cechy podzielności przez 2, 4, 8, 5, 25, 125, 3, 9. 26 września 2009 r. Uwaga: Przyjmujemy, że 0 nie
1 Genetykapopulacyjna
 1 Genetykapopulacyjna Genetyka populacyjna zajmuje się badaniem częstości występowania poszczególnych alleli oraz genotypów w populacji. Bada także zmiany tych częstości spowodowane doborem naturalnym
1 Genetykapopulacyjna Genetyka populacyjna zajmuje się badaniem częstości występowania poszczególnych alleli oraz genotypów w populacji. Bada także zmiany tych częstości spowodowane doborem naturalnym
Ewolucja molekularna człowieka okiem bioinformatyka. Justyna Wojtczak Jarosław Jeleniewicz
 Ewolucja molekularna człowieka okiem bioinformatyka Justyna Wojtczak Jarosław Jeleniewicz Informatyka w biologii - bioinformatyka Jest to szeroka dziedzina zajmująca się tworzeniem zaawansowanych baz danych,
Ewolucja molekularna człowieka okiem bioinformatyka Justyna Wojtczak Jarosław Jeleniewicz Informatyka w biologii - bioinformatyka Jest to szeroka dziedzina zajmująca się tworzeniem zaawansowanych baz danych,
6.4 Podstawowe metody statystyczne
 156 Wstęp do statystyki matematycznej 6.4 Podstawowe metody statystyczne Spóbujemy teraz w dopuszczalnym uproszczeniu przedstawić istotę analizy statystycznej. W szczególności udzielimy odpowiedzi na postawione
156 Wstęp do statystyki matematycznej 6.4 Podstawowe metody statystyczne Spóbujemy teraz w dopuszczalnym uproszczeniu przedstawić istotę analizy statystycznej. W szczególności udzielimy odpowiedzi na postawione
Metody systemowe i decyzyjne w informatyce
 Metody systemowe i decyzyjne w informatyce Laboratorium JAVA Zadanie nr 2 Rozpoznawanie liter autorzy: A. Gonczarek, J.M. Tomczak Cel zadania Celem zadania jest zapoznanie się z problemem klasyfikacji
Metody systemowe i decyzyjne w informatyce Laboratorium JAVA Zadanie nr 2 Rozpoznawanie liter autorzy: A. Gonczarek, J.M. Tomczak Cel zadania Celem zadania jest zapoznanie się z problemem klasyfikacji
Podstawy teorii ewolucji. Informacja i ewolucja
 Podstawy teorii ewolucji Informacja i ewolucja Podręczniki 2 Dla zainteresowanych http://wps.prenhall.com/esm_freeman_evol_4/ 3 Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego
Podstawy teorii ewolucji Informacja i ewolucja Podręczniki 2 Dla zainteresowanych http://wps.prenhall.com/esm_freeman_evol_4/ 3 Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego
Pobieranie prób i rozkład z próby
 Pobieranie prób i rozkład z próby Marcin Zajenkowski Marcin Zajenkowski () Pobieranie prób i rozkład z próby 1 / 15 Populacja i próba Populacja dowolnie określony zespół przedmiotów, obserwacji, osób itp.
Pobieranie prób i rozkład z próby Marcin Zajenkowski Marcin Zajenkowski () Pobieranie prób i rozkład z próby 1 / 15 Populacja i próba Populacja dowolnie określony zespół przedmiotów, obserwacji, osób itp.
BUDOWA I FUNKCJA GENOMU LUDZKIEGO
 BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
Filogeneza: problem konstrukcji grafu (drzewa) zależności pomiędzy gatunkami.
 181 Filogeneza: problem konstrukcji grafu (drzewa) zależności pomiędzy gatunkami. 3. D T(D) poprzez algorytm łączenia sąsiadów 182 D D* : macierz łącząca sąsiadów n Niech TotDist i = k=1 D i,k Definiujemy
181 Filogeneza: problem konstrukcji grafu (drzewa) zależności pomiędzy gatunkami. 3. D T(D) poprzez algorytm łączenia sąsiadów 182 D D* : macierz łącząca sąsiadów n Niech TotDist i = k=1 D i,k Definiujemy
Zadania ze statystyki, cz.7 - hipotezy statystyczne, błąd standardowy, testowanie hipotez statystycznych
 Zadania ze statystyki, cz.7 - hipotezy statystyczne, błąd standardowy, testowanie hipotez statystycznych Zad. 1 Średnia ocen z semestru letniego w populacji studentów socjologii w roku akademickim 2011/2012
Zadania ze statystyki, cz.7 - hipotezy statystyczne, błąd standardowy, testowanie hipotez statystycznych Zad. 1 Średnia ocen z semestru letniego w populacji studentów socjologii w roku akademickim 2011/2012
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
Aproksymacja funkcji a regresja symboliczna
 Aproksymacja funkcji a regresja symboliczna Problem aproksymacji funkcji polega na tym, że funkcję F(x), znaną lub określoną tablicą wartości, należy zastąpić inną funkcją, f(x), zwaną funkcją aproksymującą
Aproksymacja funkcji a regresja symboliczna Problem aproksymacji funkcji polega na tym, że funkcję F(x), znaną lub określoną tablicą wartości, należy zastąpić inną funkcją, f(x), zwaną funkcją aproksymującą
1. Symulacje komputerowe Idea symulacji Przykład. 2. Metody próbkowania Jackknife Bootstrap. 3. Łańcuchy Markova. 4. Próbkowanie Gibbsa
 BIOINFORMATYKA 1. Wykład wstępny 2. Bazy danych: projektowanie i struktura 3. Równowaga Hardyego-Weinberga, wsp. rekombinacji 4. Analiza asocjacyjna 5. Analiza asocjacyjna 6. Sekwencjonowanie nowej generacji
BIOINFORMATYKA 1. Wykład wstępny 2. Bazy danych: projektowanie i struktura 3. Równowaga Hardyego-Weinberga, wsp. rekombinacji 4. Analiza asocjacyjna 5. Analiza asocjacyjna 6. Sekwencjonowanie nowej generacji
METODY STATYSTYCZNE W BIOLOGII
 METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
METODY STATYSTYCZNE W BIOLOGII 1. Wykład wstępny 2. Populacje i próby danych 3. Testowanie hipotez i estymacja parametrów 4. Planowanie eksperymentów biologicznych 5. Najczęściej wykorzystywane testy statystyczne
Statystyka w pracy badawczej nauczyciela Wykład 4: Analiza współzależności. dr inż. Walery Susłow walery.suslow@ie.tu.koszalin.pl
 Statystyka w pracy badawczej nauczyciela Wykład 4: Analiza współzależności dr inż. Walery Susłow walery.suslow@ie.tu.koszalin.pl Statystyczna teoria korelacji i regresji (1) Jest to dział statystyki zajmujący
Statystyka w pracy badawczej nauczyciela Wykład 4: Analiza współzależności dr inż. Walery Susłow walery.suslow@ie.tu.koszalin.pl Statystyczna teoria korelacji i regresji (1) Jest to dział statystyki zajmujący
Teoria ewolucji. Ślady wspólnego pochodzenia. Dobór sztuczny i naturalny.
 Teoria ewolucji. Ślady wspólnego pochodzenia. Dobór sztuczny i naturalny. Dowody wspierające wspólne pochodzenie Skamieniałości Homologia Cechy szczątkowe Hierarchiczna klasyfikacja Zgodność drzew dla
Teoria ewolucji. Ślady wspólnego pochodzenia. Dobór sztuczny i naturalny. Dowody wspierające wspólne pochodzenie Skamieniałości Homologia Cechy szczątkowe Hierarchiczna klasyfikacja Zgodność drzew dla
