Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi.
|
|
|
- Beata Urbaniak
- 7 lat temu
- Przeglądów:
Transkrypt
1 Ryciny 193
2 Rycina 1. Zasięg i zagęszczenie łosi (liczba osobników/1000 ha) w Polsce w roku 2010 oraz rozmieszczenie 29 analizowanych populacji łosi. Na fioletowo zaznaczone zostały populacje (nr 1 14) uwzględnione w analizach zróżnicowania genetycznego, na niebiesko populacje (nr 15 29), które wraz z populacjami 1 14 posłużyły do uzyskania ogólnego obrazu zmienności genetycznej u łosi. 194
3 H1 H2 H3 H4 H6 H10 H11 H12 H13 H17 H20 H22 Rycina 2. Frekwencja 12 haplotypów regionu kontrolnego mtdna badanego odcinka o długości 607 par zasad w 27 populacjach łosi z Polski, jednej z Litwy oraz jednej z Niemiec. 195
4 Rycina 3. Drzewo filogenetyczne skonstruowane metodą łączenia sąsiadów (ang. neighbor-joining; NJ) odzwierciedlające pokrewieństwa filogenetyczne między haplotypami mtdna-cr (607 pz) łosi. Jako grupę zewnętrzną wykorzystałam sekwencje mtdna jelonka błotnego, Hydropotes inermis i renifera, Rangifer tarandus. Pozostałe numery haplotypów mtdna-cr łosi pochodzą z GenBanku i zostały objaśnione w rozdziale Materiały i Metody. Haplotypy łosi stwierdzone w Polsce oznaczyłam symbolem H oraz odpowiednim numerem. Ocenę istotności statystycznej drzewa uzyskałam wykonując test samopróbkowania (ang. bootstrap, 1000 replikacji). Procent poparcia bootstrap (ang. bootstrap suport, BS) > 70% pozwala przypisać otrzymanemu węzłowi duże prawdopodobieństwo. Nad kreską znajdują się wartości poparcia bootstrap uzyskane dla drzewa NJ, natomiast pod kreską wartości poparcia bootstrap dla drzewa filogenetycznego skonstruowanego metodą maksymalnego prawdopodobieństwa (ang. maximum likelihood, ML) zgodnie z modelem sekwencji ewolucji HKY +I. 196
5 Rycina 4. Drzewo filogenetyczne skonstruowane metodą łączenia sąsiadów (ang. neighbor-joining; NJ) odzwierciedlające pokrewieństwa filogenetyczne między haplotypami mtdna-cr (351 pz) łosi. Jako grupę zewnętrzną wykorzystałam sekwencje mtdna jelonka błotnego, Hydropotes inermis i renifera, Rangifer tarandus. Pozostałe numery haplotypów mtdna-cr łosi pochodzą z GenBanku i zostały objaśnione w rozdziale Materiały i Metody. Haplotypy łosi stwierdzone w Polsce oznaczyłam symbolem H oraz odpowiednim numerem. Ocenę istotności statystycznej drzewa uzyskałam wykonując test samopróbkowania (ang. bootstrap, 1000 replikacji). Procent poparcia bootstrap (ang. bootstrap suport, BS) > 70% pozwala przypisać otrzymanemu węzłowi duże prawdopodobieństwo. 197
6 Rycina 5. Drzewo filogenetyczne skonstruowane metodą łączenia sąsiadów (ang. neighbor-joining; NJ) odzwierciedlające pokrewieństwa filogenetyczne między haplotypami cytochromu b mtdna (1140 pz) łosi. Jako grupę zewnętrzną wykorzystałam sekwencje mtdna jelonka błotnego, Hydropotes inermis i renifera, Rangifer tarandus. Haplotypy łosi cytochromu b mtdna z haplogrupy Azjatyckiej pochodzą z GenBanku i zostały objaśnione w rozdziale Materiały i Metody. Haplotypy łosi stwierdzone w Polsce oznaczyłam symbolem H oraz odpowiednim numerem. Ocenę istotności statystycznej drzewa uzyskałam wykonując test samopróbkowania (ang. bootstrap, 1000 replikacji). Procent poparcia bootstrap (ang. bootstrap suport, BS) > 70% pozwala przypisać otrzymanemu węzłowi duże prawdopodobieństwo. Nad kreską znajdują się wartości poparcia bootstrap uzyskane dla drzewa NJ, natomiast pod kreską wartości poparcia bootstrap dla drzewa filogenetycznego skonstruowanego metodą maksymalnego prawdopodobieństwa (ang. maximum likelihood, ML) zgodnie z modelem sekwencji ewolucji HKY. 198
7 Rycina 6. Drzewo filogenetyczne skonstruowane metodą łączenia sąsiadów (ang. neighbor-joining; NJ) odzwierciedlające pokrewieństwa filogenetyczne między haplotypami mtdna (mtdna-cr + cytb, 1747 pz) łosi. Jako grupę zewnętrzną wykorzystałam sekwencje mtdna jelonka błotnego, Hydropotes inermis i renifera, Rangifer tarandus. Haplotypy łosi stwierdzone w Polsce oznaczyłam symbolem H oraz odpowiednim numerem. Do konstrukcji drzew wykorzystałam również sekwencję mtdna łosia o numerze GenBank JN należącego do haplogrupy Azjatyckiej. Ocenę istotności statystycznej drzewa uzyskałam wykonując test samopróbkowania (ang. bootstrap, 1000 replikacji). Procent poparcia bootstrap (ang. bootstrap suport, BS) > 70% pozwala przypisać otrzymanemu węzłowi duże prawdopodobieństwo. Nad kreską znajdują się wartości poparcia bootstrap uzyskane dla drzewa NJ, natomiast pod kreską wartości poparcia bootstrap dla drzewa filogenetycznego skonstruowanego metodą maksymalnego prawdopodobieństwa (ang. maximum likelihood, ML) zgodnie z modelem sekwencji ewolucji HKY +G + I. 199
8 Rycina 7. Sieć ilustrująca pokrewieństwa filogenetyczne między haplotypami mtdna (mtdna-cr + cytb, 1747 pz) u łosi w Europie. Haplotypy łosi stwierdzone w Polsce oznaczyłam symbolem H oraz odpowiednim numerem. Do konstrukcji sieci wykorzystałam również sekwencję mtdna łosia o numerze GenBank JN należącego do haplogrupy azjatyckiej. Małe czarne kółka oznaczają brakujące haplotypy, natomiast krótkie odcinki oznaczają mutacje różniące poszczególne haplotypy. 200
9 Rycina 8. Sieć ilustrująca pokrewieństwa filogenetyczne między haplotypami genu DBY14 (129 pz) u łosi (haplotypy H1 H4), sarny europejskiej, Capreolus capreolus i syberyjskiej, C. pygargus (H5), bawołu domowego, Bubalus bubalis (H6; GenBank nr AY936552), bizona, Bison bison (H7; GenBank nr AY936553), gaura, Bos gaurus (H8; GenBank nr AY936554) i bydła, Bos taurus (H9; GenBank nr AY936555). Małe czarne kółka oznaczają brakujące haplotypy, natomiast krótkie odcinki oznaczają mutacje różniące poszczególne haplotypy. 201
10 Rycina 9. Ilustracja graficzna wyników analizy składowych głównych (PCA) wykonanej na bazie macierzy wartości Φ ST w mtdna-cr dla 13 populacji łosi z Polski i jednej populacji z Litwy. Symbole populacji zostały wyjaśnione w Tabeli
11 Rycina 10. Ilustracja graficzna wyników analizy składowych głównych (PCA) wykonanej na bazie macierzy wartości F ST w mtdna-cr dla 13 populacji łosi z Polski i jednej populacji z Litwy. Symbole populacji zostały wyjaśnione w Tabeli
12 Rycina 11. Ilustracja graficzna wyników analizy składowych głównych (PCA) wykonanej na bazie macierzy wartości R ST w 11 loci mikrosatelitarnego DNA dla 13 populacji łosi z Polski i populacji z Litwy. Symbole populacji zostały wyjaśnione w Tabeli
13 Rycina 12. Ilustracja graficzna wyników analizy składowych głównych (PCA) wykonanej na bazie macierzy wartości F ST w MHC II DRB dla 7 populacji łosi z Polski i jednej populacji z Litwy. Symbole populacji zostały wyjaśnione w Tabeli
14 Rycina 13. Wykres rozkładu liczby niedopasowań nukleotydowych par porównywanych sekwencji (ang. mismatch distribution) wyznaczone z analizy haplotypów mtdna-cr (607 pz) dla 13 populacji łosi z Polski. 206
15 Rycina 14. Wykres rozkładu liczby niedopasowań nukleotydowych par porównywanych sekwencji (ang. mismatch distribution) wyznaczone z analizy haplotypów mtdna-cr (607 pz) należących do kladu Centralna Europa. 207
16 Rycina 15. Wykres rozkładu liczby niedopasowań nukleotydowych par porównywanych sekwencji (ang. mismatch distribution) wyznaczone z analizy haplotypów mtdna-cr (607 pz) należących do gałęzi Biebrza. 208
17 Rycina 16. Wykres rozkładu liczby niedopasowań nukleotydowych par porównywanych sekwencji (ang. mismatch distribution) wyznaczone z analizy haplotypów mtdna-cr (607 pz) należących do gałęzi Fennoscandia. 209
18 Rycina 17. Wpływ doboru na gen MHC II DRB przy wykorzystaniu 11 loci mikrosatelitarnych jako neutralne tło genetyczne, na które z założenia nie działa dobór naturalny. Analizy, które umożliwiły wygenerowanie przedstawionej ryciny zostały wykonane w programie Lositan, który pozwala wyznaczyć związek pomiędzy współczynnikiem F ST Wright a i heterozygotycznością oczekiwaną (H E ). 210
Wpływ struktury krajobrazu na przestrzenną zmienność genetyczną populacji myszy leśnej Apodemus flavicollis w północno wschodniej Polsce
 Uniwersytet Warszawski Wydział Biologii Sylwia Czarnomska Wpływ struktury krajobrazu na przestrzenną zmienność genetyczną populacji myszy leśnej Apodemus flavicollis w północno wschodniej Polsce Autoreferat
Uniwersytet Warszawski Wydział Biologii Sylwia Czarnomska Wpływ struktury krajobrazu na przestrzenną zmienność genetyczną populacji myszy leśnej Apodemus flavicollis w północno wschodniej Polsce Autoreferat
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
PODSTAWY BIOINFORMATYKI WYKŁAD 5 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa 4. Etapy konstrukcji drzewa filogenetycznego
Polimorfizm genu mitochondrialnej polimerazy gamma (pol γ) w populacjach ludzkich Europy
 Polimorfizm genu mitochondrialnej polimerazy gamma (pol γ) w populacjach ludzkich Europy Praca wykonana pod kierunkiem dr hab. Tomasza Grzybowskiego w Katedrze Medycyny Sądowej w Zakładzie Genetyki Molekularnej
Polimorfizm genu mitochondrialnej polimerazy gamma (pol γ) w populacjach ludzkich Europy Praca wykonana pod kierunkiem dr hab. Tomasza Grzybowskiego w Katedrze Medycyny Sądowej w Zakładzie Genetyki Molekularnej
Mitochondrialna Ewa;
 Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Mitochondrialna Ewa; jej sprzymierzeńcy i wrogowie Lien Dybczyńska Zakład genetyki, Uniwersytet Warszawski 01.05.2004 Milion lat temu Ale co dalej??? I wtedy wkracza biologia molekularna Analiza różnic
Filogenetyka molekularna I. Krzysztof Spalik
 Filogenetyka molekularna I Krzysztof Spalik Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498
Filogenetyka molekularna I Krzysztof Spalik Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498
Badania genetyczne nad populacją jelenia w północno-wschodniej Polsce
 Badania genetyczne nad populacją jelenia w północno-wschodniej Polsce Magdalena Niedziałkowska, Bogumiła Jędrzejewska, Jan Marek Wójcik Instytut Biologii Ssaków PAN w Białowieży Cele badań 1) Poznanie
Badania genetyczne nad populacją jelenia w północno-wschodniej Polsce Magdalena Niedziałkowska, Bogumiła Jędrzejewska, Jan Marek Wójcik Instytut Biologii Ssaków PAN w Białowieży Cele badań 1) Poznanie
Genomika Porównawcza. Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski
 Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
Genomika Porównawcza Agnieszka Rakowska Instytut Informatyki i Matematyki Komputerowej Uniwersytet Jagiellooski 1 Plan prezentacji 1. Rodzaje i budowa drzew filogenetycznych 2. Metody ukorzeniania drzewa
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA
 PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
PODSTAWY BIOINFORMATYKI 6 ANALIZA FILOGENETYCZNA ANALIZA FILOGENETYCZNA 1. Wstęp - filogenetyka 2. Struktura drzewa filogenetycznego 3. Metody konstrukcji drzewa - przykłady 4. Etapy konstrukcji drzewa
Konstruowanie drzew filogenetycznych. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Konstruowanie drzew filogenetycznych Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Drzewa filogenetyczne ukorzenione i nieukorzenione binarność konstrukcji topologia (sposób rozgałęziana
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Filogenetyka molekularna wykorzystuje informację zawartą w sekwencjach aminokwasów lub nukleotydów do kontrukcji drzew
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Filogenetyka molekularna wykorzystuje informację zawartą w sekwencjach aminokwasów lub nukleotydów do kontrukcji drzew
klasyfikacja fenetyczna (numeryczna)
 Teorie klasyfikacji klasyfikacja fenetyczna (numeryczna) systematyka powinna być wolna od wszelkiej teorii (a zwłaszcza od teorii ewolucji) filogeneza jako ciąg zdarzeń jest niepoznawalna opiera się na
Teorie klasyfikacji klasyfikacja fenetyczna (numeryczna) systematyka powinna być wolna od wszelkiej teorii (a zwłaszcza od teorii ewolucji) filogeneza jako ciąg zdarzeń jest niepoznawalna opiera się na
Filogenetyka molekularna I. Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji
 Filogenetyka molekularna I Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji 3 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w
Filogenetyka molekularna I Krzysztof Spalik Zakład Filogenetyki Molekularnej i Ewolucji 3 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w
Urszula Poziomek, doradca metodyczny w zakresie biologii Materiał dydaktyczny przygotowany na konferencję z cyklu Na miarę Nobla, 14 stycznia 2010 r.
 Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Ćwiczenie 1 1 Wstęp Rozważając możliwe powiązania filogenetyczne gatunków, systematyka porównuje dane molekularne. Najskuteczniejszym sposobem badania i weryfikacji różnych hipotez filogenetycznych jest
Przyczyny i konsekwencje struktury genetycznej zwierzyny
 Przyczyny i konsekwencje struktury genetycznej zwierzyny Wanda Olech, Zuza Nowak, Zbigniew Borowski - Wydział Nauk o Zwierzętach SGGW - Instytut Badawczy Leśnictwa Łowiectwo w zrównowaŝonej gospodarce
Przyczyny i konsekwencje struktury genetycznej zwierzyny Wanda Olech, Zuza Nowak, Zbigniew Borowski - Wydział Nauk o Zwierzętach SGGW - Instytut Badawczy Leśnictwa Łowiectwo w zrównowaŝonej gospodarce
Białowieża, Prof. dr hab. Jan M. Wójcik Instytut Biologii Ssaków PAN w Białowieży
 Białowieża, 05.04.2017 Prof. dr hab. Jan M. Wójcik Instytut Biologii Ssaków PAN w Białowieży e-mail: jwojcik@ibs.bialowieza.pl Recenzja rozprawy doktorskiej mgr. Macieja Matosiuka pt. Hybridization and
Białowieża, 05.04.2017 Prof. dr hab. Jan M. Wójcik Instytut Biologii Ssaków PAN w Białowieży e-mail: jwojcik@ibs.bialowieza.pl Recenzja rozprawy doktorskiej mgr. Macieja Matosiuka pt. Hybridization and
Filogenetyka molekularna I
 2 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498 Filogenetyka molekularna I John C. Avise
2 Literatura Krzysztof Spalik, Marcin Piwczyński (2009), Rekonstrukcja filogenezy i wnioskowanie filogenetyczne w badaniach ewolucyjnych, Kosmos 58(3-4): 485-498 Filogenetyka molekularna I John C. Avise
Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna
 Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna Spis treści Przedmowa................................. Podziękowania............................... XIII XIV 1 Metody genetyki molekularnej w badaniach
Księgarnia PWN: Joanna R. Freeland - Ekologia molekularna Spis treści Przedmowa................................. Podziękowania............................... XIII XIV 1 Metody genetyki molekularnej w badaniach
Ćwiczenia nr 4. Programy komputerowe stosowane do analizy danych molekularnych
 Techniki molekularne ćw. 4 1 z 6 Ćwiczenia nr 4. Programy komputerowe stosowane do analizy danych molekularnych Istnieje ogromna liczba programów służących do analizy danych molekularnych. Wiele z nich
Techniki molekularne ćw. 4 1 z 6 Ćwiczenia nr 4. Programy komputerowe stosowane do analizy danych molekularnych Istnieje ogromna liczba programów służących do analizy danych molekularnych. Wiele z nich
Filogenetyka molekularna. Dr Anna Karnkowska Zakład Filogenetyki Molekularnej i Ewolucji
 Filogenetyka molekularna Dr Anna Karnkowska Zakład Filogenetyki Molekularnej i Ewolucji Co to jest filogeneza? Filogeneza=drzewo filogenetyczne=drzewo rodowe=drzewo to rozgałęziający się diagram, który
Filogenetyka molekularna Dr Anna Karnkowska Zakład Filogenetyki Molekularnej i Ewolucji Co to jest filogeneza? Filogeneza=drzewo filogenetyczne=drzewo rodowe=drzewo to rozgałęziający się diagram, który
Mechanizmy ewolucji. SYLABUS A. Informacje ogólne
 Mechanizmy ewolucji A. Informacje ogólne Elementy sylabusu Nazwa jednostki prowadzącej kierunek Nazwa kierunku studiów Poziom kształcenia Profil studiów Forma studiów Język Rodzaj Rok studiów /semestr
Mechanizmy ewolucji A. Informacje ogólne Elementy sylabusu Nazwa jednostki prowadzącej kierunek Nazwa kierunku studiów Poziom kształcenia Profil studiów Forma studiów Język Rodzaj Rok studiów /semestr
Przykładowa analiza danych
 Przykładowa analiza danych W analizie wykorzystano dane pochodzące z publicznego repozytorium ArrayExpress udostępnionego na stronach Europejskiego Instytutu Bioinformatyki (http://www.ebi.ac.uk/). Zbiór
Przykładowa analiza danych W analizie wykorzystano dane pochodzące z publicznego repozytorium ArrayExpress udostępnionego na stronach Europejskiego Instytutu Bioinformatyki (http://www.ebi.ac.uk/). Zbiór
Porównywanie i dopasowywanie sekwencji
 Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
Porównywanie i dopasowywanie sekwencji Związek bioinformatyki z ewolucją Wraz ze wzrostem dostępności sekwencji DNA i białek pojawiła się nowa możliwość śledzenia ewolucji na poziomie molekularnym Ewolucja
MARKERY MIKROSATELITARNE
 MARKERY MIKROSATELITARNE Badania laboratoryjne prowadzone w Katedrze Genetyki i Ogólnej Hodowli Zwierząt SGGW w ramach monitoringu genetycznego wykorzystują analizę genetyczną markerów mikrosatelitarnych.
MARKERY MIKROSATELITARNE Badania laboratoryjne prowadzone w Katedrze Genetyki i Ogólnej Hodowli Zwierząt SGGW w ramach monitoringu genetycznego wykorzystują analizę genetyczną markerów mikrosatelitarnych.
Struktura genetyczna populacji łosia (Alces alces) w dolinie Biebrzy
 Uniwersytet w Białymstoku Wydział Biologiczno-Chemiczny Instytut Biologii Magdalena Świsłocka Struktura genetyczna populacji łosia (Alces alces) w dolinie Biebrzy Rozprawa doktorska Wykonana w Zakładzie
Uniwersytet w Białymstoku Wydział Biologiczno-Chemiczny Instytut Biologii Magdalena Świsłocka Struktura genetyczna populacji łosia (Alces alces) w dolinie Biebrzy Rozprawa doktorska Wykonana w Zakładzie
Monitoring genetyczny populacji wilka (Canis lupus) jako nowy element monitoringu stanu populacji dużych drapieżników
 Monitoring genetyczny populacji wilka (Canis lupus) jako nowy element monitoringu stanu populacji dużych drapieżników Wojciech Śmietana Co to jest monitoring genetyczny? Monitoring genetyczny to regularnie
Monitoring genetyczny populacji wilka (Canis lupus) jako nowy element monitoringu stanu populacji dużych drapieżników Wojciech Śmietana Co to jest monitoring genetyczny? Monitoring genetyczny to regularnie
Badanie doboru naturalnego na poziomie molekularnym
 Badanie doboru naturalnego na poziomie molekularnym Podstawy ewolucji molekulanej Jak ewoluują sekwencje Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na
Badanie doboru naturalnego na poziomie molekularnym Podstawy ewolucji molekulanej Jak ewoluują sekwencje Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na
Podstawy ewolucji molekularnej. Ewolucja sekwencji DNA i białek
 Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji } Mutacje } tworzą nowe allele genów } Inwersje } zmieniają układ genów na chromosomach } mogą uniemożliwić rekombinację
Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji } Mutacje } tworzą nowe allele genów } Inwersje } zmieniają układ genów na chromosomach } mogą uniemożliwić rekombinację
Anna Szewczyk. Wydział Geodezji Górniczej i InŜynierii środowiska AGH
 Anna Szewczyk Wydział Geodezji Górniczej i InŜynierii środowiska AGH Zastosowania biblioteki Genetics programu R The genetics Package Tytuł: Populacja genetyczna Wersja:1.2.0 Data utworzenia: 2005-11-09
Anna Szewczyk Wydział Geodezji Górniczej i InŜynierii środowiska AGH Zastosowania biblioteki Genetics programu R The genetics Package Tytuł: Populacja genetyczna Wersja:1.2.0 Data utworzenia: 2005-11-09
Podstawy ewolucji molekularnej. Ewolucja sekwencji DNA i białek
 Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Egzamin: 29.01.2018 16:00, sala 9B Pierwsza synteza Ewolucja jako zmiany częstości alleli w populacji Mutacje jako źródło nowych alleli Dobór
Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Egzamin: 29.01.2018 16:00, sala 9B Pierwsza synteza Ewolucja jako zmiany częstości alleli w populacji Mutacje jako źródło nowych alleli Dobór
Podstawy ewolucji molekularnej. Ewolucja sekwencji DNA i białek
 Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na chromosomach mogą uniemożliwić rekombinację
Podstawy ewolucji molekularnej Ewolucja sekwencji DNA i białek Zmiany genetyczne w ewolucji Mutacje tworzą nowe allele genów Inwersje zmieniają układ genów na chromosomach mogą uniemożliwić rekombinację
Wszystkie wyniki w postaci ułamków należy podawać z dokładnością do czterech miejsc po przecinku!
 Pracownia statystyczno-filogenetyczna Liczba punktów (wypełnia KGOB) / 30 PESEL Imię i nazwisko Grupa Nr Czas: 90 min. Łączna liczba punktów do zdobycia: 30 Czerwona Niebieska Zielona Żółta Zaznacz znakiem
Pracownia statystyczno-filogenetyczna Liczba punktów (wypełnia KGOB) / 30 PESEL Imię i nazwisko Grupa Nr Czas: 90 min. Łączna liczba punktów do zdobycia: 30 Czerwona Niebieska Zielona Żółta Zaznacz znakiem
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
Teoria ewolucji. Podstawowe pojęcia. Wspólne pochodzenie.
 Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego 5A pgolik@igib.uw.edu.pl Informacje, materiały: http://www.igib.uw.edu.pl/
Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego 5A pgolik@igib.uw.edu.pl Informacje, materiały: http://www.igib.uw.edu.pl/
RECENZJA rozprawy doktorskiej mgr Magdaleny Herdegen pt. "Phenotypic traits, genetic variation and gene flow between guppy populations in Venezuela"
 UNIWERSYTET W BIAŁYMSTOKU Instytut Biologii Świerkowa 20B, 15-950 Białystok tel: 085 745 73 04, fax: 085 745 73 01 Dr hab. Mirosław Ratkiewicz prof. UwB Białystok, 27.04. 2014r. e-mail: ermi@uwb.edu.pl
UNIWERSYTET W BIAŁYMSTOKU Instytut Biologii Świerkowa 20B, 15-950 Białystok tel: 085 745 73 04, fax: 085 745 73 01 Dr hab. Mirosław Ratkiewicz prof. UwB Białystok, 27.04. 2014r. e-mail: ermi@uwb.edu.pl
Teoria ewolucji. Podstawy wspólne pochodzenie.
 Teoria ewolucji. Podstawy wspólne pochodzenie. Ewolucja biologiczna } Znaczenie ogólne: } proces zmian informacji genetycznej (częstości i rodzaju alleli), } które to zmiany są przekazywane z pokolenia
Teoria ewolucji. Podstawy wspólne pochodzenie. Ewolucja biologiczna } Znaczenie ogólne: } proces zmian informacji genetycznej (częstości i rodzaju alleli), } które to zmiany są przekazywane z pokolenia
Bioinformatyka Laboratorium, 30h. Michał Bereta
 Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Metoda NJ (przyłączania sąsiadów) umożliwia tworzenie drzewa addytywnego: odległości ewolucyjne między sekwencjami
Bioinformatyka Laboratorium, 30h Michał Bereta mbereta@pk.edu.pl www.michalbereta.pl 1 Metoda NJ (przyłączania sąsiadów) umożliwia tworzenie drzewa addytywnego: odległości ewolucyjne między sekwencjami
WSTĘP. Copyright 2011, Joanna Szyda
 BIOINFORMATYKA 1. Wykład wstępny 2. Struktury danych w badaniach bioinformatycznych 3. Bazy danych: projektowanie i struktura 4. Bazy danych: projektowanie i struktura 5. Równowaga Hardyego-Weinberga,
BIOINFORMATYKA 1. Wykład wstępny 2. Struktury danych w badaniach bioinformatycznych 3. Bazy danych: projektowanie i struktura 4. Bazy danych: projektowanie i struktura 5. Równowaga Hardyego-Weinberga,
Acknowledgement. Drzewa filogenetyczne
 Wykład 8 Drzewa Filogenetyczne Lokalizacja genów Some figures from: Acknowledgement M. Zvelebil, J.O. Baum, Introduction to Bioinformatics, Garland Science 2008 Tradycyjne drzewa pokrewieństwa Drzewa oparte
Wykład 8 Drzewa Filogenetyczne Lokalizacja genów Some figures from: Acknowledgement M. Zvelebil, J.O. Baum, Introduction to Bioinformatics, Garland Science 2008 Tradycyjne drzewa pokrewieństwa Drzewa oparte
Ekologia molekularna. wykład 6
 Ekologia molekularna wykład 6 Tempo mutacji Tempo błędu polimerazy: 10-4 pomyłka polimerazy 10-8 po naprawie błędów Faktyczne tempo mutacji: 10-9/zasadę/pokolenie W genomie człowieka jest 3 x 109 zasad
Ekologia molekularna wykład 6 Tempo mutacji Tempo błędu polimerazy: 10-4 pomyłka polimerazy 10-8 po naprawie błędów Faktyczne tempo mutacji: 10-9/zasadę/pokolenie W genomie człowieka jest 3 x 109 zasad
Ocena zmienności genetycznej jesionu wyniosłego w Polsce. Jarosław Burczyk
 Ocena zmienności genetycznej jesionu wyniosłego w Polsce Jarosław Burczyk Zmienność genetyczna W naturalnych zbiorowiskach, zróżnicowanie genetyczne jest wynikiem doboru naturalnego i historii demograficznej
Ocena zmienności genetycznej jesionu wyniosłego w Polsce Jarosław Burczyk Zmienność genetyczna W naturalnych zbiorowiskach, zróżnicowanie genetyczne jest wynikiem doboru naturalnego i historii demograficznej
Przyrównanie sekwencji. Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu
 Przyrównanie sekwencji Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych),
Przyrównanie sekwencji Magda Mielczarek Katedra Genetyki Uniwersytet Przyrodniczy we Wrocławiu Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych),
Ewolucja molekularna człowieka okiem bioinformatyka. Justyna Wojtczak Jarosław Jeleniewicz
 Ewolucja molekularna człowieka okiem bioinformatyka Justyna Wojtczak Jarosław Jeleniewicz Informatyka w biologii - bioinformatyka Jest to szeroka dziedzina zajmująca się tworzeniem zaawansowanych baz danych,
Ewolucja molekularna człowieka okiem bioinformatyka Justyna Wojtczak Jarosław Jeleniewicz Informatyka w biologii - bioinformatyka Jest to szeroka dziedzina zajmująca się tworzeniem zaawansowanych baz danych,
Teoria ewolucji. Podstawowe pojęcia. Wspólne pochodzenie.
 Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Ewolucja Znaczenie ogólne: zmiany zachodzące stopniowo w czasie W biologii ewolucja biologiczna W astronomii i kosmologii ewolucja gwiazd i wszechświata
Teoria ewolucji Podstawowe pojęcia. Wspólne pochodzenie. Ewolucja Znaczenie ogólne: zmiany zachodzące stopniowo w czasie W biologii ewolucja biologiczna W astronomii i kosmologii ewolucja gwiazd i wszechświata
ZARZĄDZANIE POPULACJAMI ZWIERZĄT
 ZARZĄDZANIE POPULACJAMI ZWIERZĄT Ćwiczenia 1 mgr Magda Kaczmarek-Okrój magda_kaczmarek_okroj@sggw.pl 1 ZAGADNIENIA struktura genetyczna populacji obliczanie frekwencji genotypów obliczanie frekwencji alleli
ZARZĄDZANIE POPULACJAMI ZWIERZĄT Ćwiczenia 1 mgr Magda Kaczmarek-Okrój magda_kaczmarek_okroj@sggw.pl 1 ZAGADNIENIA struktura genetyczna populacji obliczanie frekwencji genotypów obliczanie frekwencji alleli
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI
 PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
PODSTAWY BIOINFORMATYKI WYKŁAD 4 DOPASOWANIE SEKWENCJI DOPASOWANIE SEKWENCJI 1. Dopasowanie sekwencji - definicja 2. Wizualizacja dopasowania sekwencji 3. Miary podobieństwa sekwencji 4. Przykłady programów
Filogeneza: problem konstrukcji grafu (drzewa) zależności pomiędzy gatunkami.
 181 Filogeneza: problem konstrukcji grafu (drzewa) zależności pomiędzy gatunkami. 3. D T(D) poprzez algorytm łączenia sąsiadów 182 D D* : macierz łącząca sąsiadów n Niech TotDist i = k=1 D i,k Definiujemy
181 Filogeneza: problem konstrukcji grafu (drzewa) zależności pomiędzy gatunkami. 3. D T(D) poprzez algorytm łączenia sąsiadów 182 D D* : macierz łącząca sąsiadów n Niech TotDist i = k=1 D i,k Definiujemy
Ekologia molekularna. wykład 4
 Ekologia molekularna wykład 4 Zróżnicowanie między populacjami Przyczyny odchyleń od HWE Czynniki demograficzne nielosowe kojarzenie wsobność (inbred) struktura genetyczna populacji (subpopulacje) migracje
Ekologia molekularna wykład 4 Zróżnicowanie między populacjami Przyczyny odchyleń od HWE Czynniki demograficzne nielosowe kojarzenie wsobność (inbred) struktura genetyczna populacji (subpopulacje) migracje
Podstawy biologii. Informacja genetyczna. Co to jest ewolucja.
 Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Materiał genetyczny Materiałem genetycznym są kwasy nukleinowe Materiałem genetycznym organizmów komórkowych jest kwas deoksyrybonukleinowy
Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Materiał genetyczny Materiałem genetycznym są kwasy nukleinowe Materiałem genetycznym organizmów komórkowych jest kwas deoksyrybonukleinowy
OCENA ROZPRAWY NA STOPIEŃ NAUKOWY DOKTORA NAUK MEDYCZNYCH LEKARZA KRZYSZTOFA WIKTORA. pt. Analiza sekwencji mitochondrialnego DNA u
 Prof. dr hab. n. med. Paweł Kamiński Klinika Ginekologii i Ginekologii Onkologicznej Wojskowego Instytutu Medycznego OCENA ROZPRAWY NA STOPIEŃ NAUKOWY DOKTORA NAUK MEDYCZNYCH LEKARZA KRZYSZTOFA WIKTORA
Prof. dr hab. n. med. Paweł Kamiński Klinika Ginekologii i Ginekologii Onkologicznej Wojskowego Instytutu Medycznego OCENA ROZPRAWY NA STOPIEŃ NAUKOWY DOKTORA NAUK MEDYCZNYCH LEKARZA KRZYSZTOFA WIKTORA
Best for Biodiversity
 W tym miejscu realizowany jest projekt LIFE + Ochrona różnorodności biologicznej na obszarach leśnych, w tym w ramach sieci Natura 2000 promocja najlepszych praktyk Best for Biodiversity NAJLEPSZE PRAKTYKI
W tym miejscu realizowany jest projekt LIFE + Ochrona różnorodności biologicznej na obszarach leśnych, w tym w ramach sieci Natura 2000 promocja najlepszych praktyk Best for Biodiversity NAJLEPSZE PRAKTYKI
RECENZJA Rozprawy doktorskiej mgr Emilii Rabiniak pt. Analiza kopalnego DNA rodzajów Lepus i Ochotona
 Wstęp RECENZJA Rozprawy doktorskiej mgr Emilii Rabiniak pt. Analiza kopalnego DNA rodzajów Lepus i Ochotona (Lagomorpha, Mammalia) z plejstocenu i holocenu Europy Środkowej Przedstawiona do oceny praca
Wstęp RECENZJA Rozprawy doktorskiej mgr Emilii Rabiniak pt. Analiza kopalnego DNA rodzajów Lepus i Ochotona (Lagomorpha, Mammalia) z plejstocenu i holocenu Europy Środkowej Przedstawiona do oceny praca
Konstrukcja drzew filogenetycznych podstawy teoretyczne.
 Dorota Rogalla Urszula Rogalla Konstrukcja drzew filogenetycznych podstawy teoretyczne. Streszczenie Wśród wyzwań stojących przed genetyką wymienić można nie tylko kojarzone klasycznie poznawanie struktury
Dorota Rogalla Urszula Rogalla Konstrukcja drzew filogenetycznych podstawy teoretyczne. Streszczenie Wśród wyzwań stojących przed genetyką wymienić można nie tylko kojarzone klasycznie poznawanie struktury
Budowanie drzewa filogenetycznego
 Szkoła Festiwalu Nauki 134567 Wojciech Grajkowski Szkoła Festiwalu Nauki, ul. Ks. Trojdena 4, 02-109 Warszawa www.sfn.edu.pl sfn@iimcb.gov.pl Budowanie drzewa filogenetycznego Cel Ćwiczenie polega na budowaniu
Szkoła Festiwalu Nauki 134567 Wojciech Grajkowski Szkoła Festiwalu Nauki, ul. Ks. Trojdena 4, 02-109 Warszawa www.sfn.edu.pl sfn@iimcb.gov.pl Budowanie drzewa filogenetycznego Cel Ćwiczenie polega na budowaniu
GENETYKA POPULACJI NA PRZESTRZENI WIEKU
 Wszechświat, t. 115, nr 7 9/2014 ARTYKUŁY 183 substratem do produkcji serotoniny, będącej hormonem tkankowym w znacznym stopniu odpowiadającym za nasz nastrój serotonina zwana jest również hormonem szczęścia.
Wszechświat, t. 115, nr 7 9/2014 ARTYKUŁY 183 substratem do produkcji serotoniny, będącej hormonem tkankowym w znacznym stopniu odpowiadającym za nasz nastrój serotonina zwana jest również hormonem szczęścia.
Ćwiczenie 12. Diagnostyka molekularna. Poszukiwanie SNPs Odczytywanie danych z sekwencjonowania. Prof. dr hab. Roman Zieliński
 Ćwiczenie 12 Diagnostyka molekularna. Poszukiwanie SNPs Odczytywanie danych z sekwencjonowania Prof. dr hab. Roman Zieliński 1. Diagnostyka molekularna 1.1. Pytania i zagadnienia 1.1.1. Jak definiujemy
Ćwiczenie 12 Diagnostyka molekularna. Poszukiwanie SNPs Odczytywanie danych z sekwencjonowania Prof. dr hab. Roman Zieliński 1. Diagnostyka molekularna 1.1. Pytania i zagadnienia 1.1.1. Jak definiujemy
( x) Równanie regresji liniowej ma postać. By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : Gdzie:
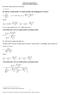 ma postać y = ax + b Równanie regresji liniowej By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : xy b = a = b lub x Gdzie: xy = też a = x = ( b ) i to dane empiryczne, a ilość
ma postać y = ax + b Równanie regresji liniowej By obliczyć współczynniki a i b należy posłużyć się następującymi wzorami 1 : xy b = a = b lub x Gdzie: xy = też a = x = ( b ) i to dane empiryczne, a ilość
PORÓWNYWANIE POPULACJI POD WZGLĘDEM STRUKTURY
 PORÓWNYWANIE POPULACJI POD WZGLĘDEM STRUKTURY obliczanie dystansu dzielącego grupy (subpopulacje) wyrażonego za pomocą indeksu F Wrighta (fixation index) w modelu jednego locus 1 Ćwiczenia III Mgr Kaczmarek-Okrój
PORÓWNYWANIE POPULACJI POD WZGLĘDEM STRUKTURY obliczanie dystansu dzielącego grupy (subpopulacje) wyrażonego za pomocą indeksu F Wrighta (fixation index) w modelu jednego locus 1 Ćwiczenia III Mgr Kaczmarek-Okrój
Algorytmy genetyczne. Materiały do laboratorium PSI. Studia stacjonarne i niestacjonarne
 Algorytmy genetyczne Materiały do laboratorium PSI Studia stacjonarne i niestacjonarne Podstawowy algorytm genetyczny (PAG) Schemat blokowy algorytmu genetycznego Znaczenia, pochodzących z biologii i genetyki,
Algorytmy genetyczne Materiały do laboratorium PSI Studia stacjonarne i niestacjonarne Podstawowy algorytm genetyczny (PAG) Schemat blokowy algorytmu genetycznego Znaczenia, pochodzących z biologii i genetyki,
BIOLOGIA EGZAMIN KLASYFIKACYJNY 2015/16. KLASA III Gimnazjum. Imię:... Nazwisko:... Data:...
 BIOLOGIA EGZAMIN KLASYFIKACYJNY 2015/16 KLASA III Gimnazjum Imię:... Nazwisko:... Data:... 1. Ponumeruj poziomy organizacji materiału genetycznego, rozpoczynając od poziomu najniższego: - chromatyna -
BIOLOGIA EGZAMIN KLASYFIKACYJNY 2015/16 KLASA III Gimnazjum Imię:... Nazwisko:... Data:... 1. Ponumeruj poziomy organizacji materiału genetycznego, rozpoczynając od poziomu najniższego: - chromatyna -
Polimorfizm. sekwencji mitochondrialnego DNA gatunku Canis familiaris - aspekty filogenetyczne i identyfikacyjne. Anna Czarnecka
 Uniwersytet Mikołaja Kopernika w Toruniu Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Polimorfizm sekwencji mitochondrialnego DNA gatunku Canis familiaris - aspekty filogenetyczne i identyfikacyjne
Uniwersytet Mikołaja Kopernika w Toruniu Collegium Medicum im. Ludwika Rydygiera w Bydgoszczy Polimorfizm sekwencji mitochondrialnego DNA gatunku Canis familiaris - aspekty filogenetyczne i identyfikacyjne
Algorytmy genetyczne. Materiały do laboratorium PSI. Studia niestacjonarne
 Algorytmy genetyczne Materiały do laboratorium PSI Studia niestacjonarne Podstawowy algorytm genetyczny (PAG) Schemat blokowy algorytmu genetycznego Znaczenia, pochodzących z biologii i genetyki, pojęć
Algorytmy genetyczne Materiały do laboratorium PSI Studia niestacjonarne Podstawowy algorytm genetyczny (PAG) Schemat blokowy algorytmu genetycznego Znaczenia, pochodzących z biologii i genetyki, pojęć
ZARZĄDZANIE POPULACJAMI ZWIERZĄT 1. RÓWNOWAGA GENETYCZNA POPULACJI. Prowadzący: dr Wioleta Drobik Katedra Genetyki i Ogólnej Hodowli Zwierząt
 ZARZĄDZANIE POPULACJAMI ZWIERZĄT 1. RÓWNOWAGA GENETYCZNA POPULACJI Fot. W. Wołkow Prowadzący: dr Wioleta Drobik Katedra Genetyki i Ogólnej Hodowli Zwierząt POPULACJA Zbiór organizmów żywych, które łączy
ZARZĄDZANIE POPULACJAMI ZWIERZĄT 1. RÓWNOWAGA GENETYCZNA POPULACJI Fot. W. Wołkow Prowadzący: dr Wioleta Drobik Katedra Genetyki i Ogólnej Hodowli Zwierząt POPULACJA Zbiór organizmów żywych, które łączy
Wstęp do Biologii Obliczeniowej
 Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
Wstęp do Biologii Obliczeniowej Zagadnienia na kolokwium Bartek Wilczyński 5. czerwca 2018 Sekwencje DNA i grafy Sekwencje w biologii, DNA, RNA, białka, alfabety, transkrypcja DNA RNA, translacja RNA białko,
ALGORYTM RANDOM FOREST
 SKRYPT PRZYGOTOWANY NA ZAJĘCIA INDUKOWANYCH REGUŁ DECYZYJNYCH PROWADZONYCH PRZEZ PANA PAWŁA WOJTKIEWICZA ALGORYTM RANDOM FOREST Katarzyna Graboś 56397 Aleksandra Mańko 56699 2015-01-26, Warszawa ALGORYTM
SKRYPT PRZYGOTOWANY NA ZAJĘCIA INDUKOWANYCH REGUŁ DECYZYJNYCH PROWADZONYCH PRZEZ PANA PAWŁA WOJTKIEWICZA ALGORYTM RANDOM FOREST Katarzyna Graboś 56397 Aleksandra Mańko 56699 2015-01-26, Warszawa ALGORYTM
WSTĘP DO REGRESJI LOGISTYCZNEJ. Dr Wioleta Drobik-Czwarno
 WSTĘP DO REGRESJI LOGISTYCZNEJ Dr Wioleta Drobik-Czwarno REGRESJA LOGISTYCZNA Zmienna zależna jest zmienną dychotomiczną (dwustanową) przyjmuje dwie wartości, najczęściej 0 i 1 Zmienną zależną może być:
WSTĘP DO REGRESJI LOGISTYCZNEJ Dr Wioleta Drobik-Czwarno REGRESJA LOGISTYCZNA Zmienna zależna jest zmienną dychotomiczną (dwustanową) przyjmuje dwie wartości, najczęściej 0 i 1 Zmienną zależną może być:
Dziedziczenie poligenowe
 Dziedziczenie poligenowe Dziedziczenie cech ilościowych Dziedziczenie wieloczynnikowe Na wartość cechy wpływa Komponenta genetyczna - wspólne oddziaływanie wielu (najczęściej jest to liczba nieznana) genów,
Dziedziczenie poligenowe Dziedziczenie cech ilościowych Dziedziczenie wieloczynnikowe Na wartość cechy wpływa Komponenta genetyczna - wspólne oddziaływanie wielu (najczęściej jest to liczba nieznana) genów,
Recenzja rozprawy doktorskiej mgr Marty Heleny Czernik pt. Analiza molekularna zimowej diety łosia, jelenia szlachetnego i sarny europejskiej w Polsce
 Białowieża, 23.05.2017 Recenzja rozprawy doktorskiej mgr Marty Heleny Czernik pt. Analiza molekularna zimowej diety łosia, jelenia szlachetnego i sarny europejskiej w Polsce Uwagi formalne Rozprawa doktorska
Białowieża, 23.05.2017 Recenzja rozprawy doktorskiej mgr Marty Heleny Czernik pt. Analiza molekularna zimowej diety łosia, jelenia szlachetnego i sarny europejskiej w Polsce Uwagi formalne Rozprawa doktorska
Dopasowywanie sekwencji (ang. sequence alignment) Metody dopasowywania sekwencji. Homologia a podobieństwo sekwencji. Rodzaje dopasowania
 Wprowadzenie do Informatyki Biomedycznej Wykład 2: Metody dopasowywania sekwencji Wydział Informatyki PB Dopasowywanie sekwencji (ang. sequence alignment) Dopasowywanie (przyrównywanie) sekwencji polega
Wprowadzenie do Informatyki Biomedycznej Wykład 2: Metody dopasowywania sekwencji Wydział Informatyki PB Dopasowywanie sekwencji (ang. sequence alignment) Dopasowywanie (przyrównywanie) sekwencji polega
Jak sprawdzić normalność rozkładu w teście dla prób zależnych?
 Jak sprawdzić normalność rozkładu w teście dla prób zależnych? W pliku zalezne_10.sta znajdują się dwie zmienne: czasu biegu przed rozpoczęciem cyklu treningowego (zmienna 1) oraz czasu biegu po zakończeniu
Jak sprawdzić normalność rozkładu w teście dla prób zależnych? W pliku zalezne_10.sta znajdują się dwie zmienne: czasu biegu przed rozpoczęciem cyklu treningowego (zmienna 1) oraz czasu biegu po zakończeniu
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI
 ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI Joanna Szyda Magdalena Frąszczak Magda Mielczarek WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
ANALIZA DANYCH POCHODZĄCYCH Z SEKWENCJONOWANIA NASTĘPNEJ GENERACJI Joanna Szyda Magdalena Frąszczak Magda Mielczarek WSTĘP 1. Katedra Genetyki 2. Pracownia biostatystyki 3. Projekty NGS 4. Charakterystyka
Ewolucjonizm NEODARWINIZM. Dr Jacek Francikowski Uniwersyteckie Towarzystwo Naukowe Uniwersytet Śląski w Katowicach
 Ewolucjonizm NEODARWINIZM Dr Jacek Francikowski Uniwersyteckie Towarzystwo Naukowe Uniwersytet Śląski w Katowicach Główne paradygmaty biologii Wspólne początki życia Komórka jako podstawowo jednostka funkcjonalna
Ewolucjonizm NEODARWINIZM Dr Jacek Francikowski Uniwersyteckie Towarzystwo Naukowe Uniwersytet Śląski w Katowicach Główne paradygmaty biologii Wspólne początki życia Komórka jako podstawowo jednostka funkcjonalna
Korelacja oznacza współwystępowanie, nie oznacza związku przyczynowo-skutkowego
 Korelacja oznacza współwystępowanie, nie oznacza związku przyczynowo-skutkowego Współczynnik korelacji opisuje siłę i kierunek związku. Jest miarą symetryczną. Im wyższa korelacja tym lepiej potrafimy
Korelacja oznacza współwystępowanie, nie oznacza związku przyczynowo-skutkowego Współczynnik korelacji opisuje siłę i kierunek związku. Jest miarą symetryczną. Im wyższa korelacja tym lepiej potrafimy
Testowanie hipotez dla dwóch zmiennych zależnych. Moc testu. Minimalna liczność próby; Regresja prosta; Korelacja Pearsona;
 LABORATORIUM 4 Testowanie hipotez dla dwóch zmiennych zależnych. Moc testu. Minimalna liczność próby; Regresja prosta; Korelacja Pearsona; dwie zmienne zależne mierzalne małe próby duże próby rozkład normalny
LABORATORIUM 4 Testowanie hipotez dla dwóch zmiennych zależnych. Moc testu. Minimalna liczność próby; Regresja prosta; Korelacja Pearsona; dwie zmienne zależne mierzalne małe próby duże próby rozkład normalny
PRZYRÓWNANIE SEKWENCJI
 http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
http://theta.edu.pl/ Podstawy Bioinformatyki III PRZYRÓWNANIE SEKWENCJI 1 Sequence alignment - przyrównanie sekwencji Poszukiwanie ciągów znaków (zasad nukleotydowych lub reszt aminokwasowych), które posiadają
Analizy filogenetyczne
 BIOINFORMATYKA edycja 2016 / 2017 wykład 6 Analizy filogenetyczne dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Cele i zastosowania 2. Podstawy ewolucyjne
BIOINFORMATYKA edycja 2016 / 2017 wykład 6 Analizy filogenetyczne dr Jacek Śmietański jacek.smietanski@ii.uj.edu.pl http://jaceksmietanski.net Plan wykładu 1. Cele i zastosowania 2. Podstawy ewolucyjne
Strefa pokrycia radiowego wokół stacji bazowych. Zasięg stacji bazowych Zazębianie się komórek
 Problem zapożyczania kanałów z wykorzystaniem narzędzi optymalizacji Wprowadzenie Rozwiązanie problemu przydziału częstotliwości prowadzi do stanu, w którym każdej stacji bazowej przydzielono żądaną liczbę
Problem zapożyczania kanałów z wykorzystaniem narzędzi optymalizacji Wprowadzenie Rozwiązanie problemu przydziału częstotliwości prowadzi do stanu, w którym każdej stacji bazowej przydzielono żądaną liczbę
SYMULACJE NUMERYCZNE W OCENIE RYZYKA
 SYMULACJE NUMERYCZNE W OCENIE RYZYKA Dr Marek Biesiada Instytut Medycyny Pracy i Zdrowia Środowiskowego, Sosnowiec Główną trudnością metodologiczną w procesie ocen ryzyka zdrowotnego jest złożoność oddziaływań
SYMULACJE NUMERYCZNE W OCENIE RYZYKA Dr Marek Biesiada Instytut Medycyny Pracy i Zdrowia Środowiskowego, Sosnowiec Główną trudnością metodologiczną w procesie ocen ryzyka zdrowotnego jest złożoność oddziaływań
Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV
 Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV Cel ćwiczenia Określenie podatności na zakażenie wirusem HIV poprzez detekcję homo lub heterozygotyczności
Ćwiczenie 3. Amplifikacja genu ccr5 Homo sapiens wykrywanie delecji Δ32pz warunkującej oporność na wirusa HIV Cel ćwiczenia Określenie podatności na zakażenie wirusem HIV poprzez detekcję homo lub heterozygotyczności
Metoda dokładnej rekonstrukcji drzew filogenetycznych genów. współczynników substytucji dla genów i gatunków
 Metoda dokładnej rekonstrukcji drzew filogenetycznych genów z zastosowaniem współczynników substytucji dla genów i gatunków Na podstawie artykułu Accurate gene-tree reconstruction by learning gene- and
Metoda dokładnej rekonstrukcji drzew filogenetycznych genów z zastosowaniem współczynników substytucji dla genów i gatunków Na podstawie artykułu Accurate gene-tree reconstruction by learning gene- and
a) Zapisz genotyp tego mężczyzny... oraz zaznacz poniżej (A, B, C lub D), jaki procent gamet tego mężczyzny będzie miało genotyp ax b.
 W tomie 2 zbioru zadań z biologii z powodu nieprawidłowego wprowadzenia komendy przenoszenia spójników i przyimków do następnej linii wystąpiła zamiana samotnych dużych liter (A, I, W, U) na małe litery.
W tomie 2 zbioru zadań z biologii z powodu nieprawidłowego wprowadzenia komendy przenoszenia spójników i przyimków do następnej linii wystąpiła zamiana samotnych dużych liter (A, I, W, U) na małe litery.
Pamiętając o komplementarności zasad azotowych, dopisz sekwencję nukleotydów brakującej nici DNA. A C C G T G C C A A T C G A...
 1. Zadanie (0 2 p. ) Porównaj mitozę i mejozę, wpisując do tabeli podane określenia oraz cyfry. ta sama co w komórce macierzystej, o połowę mniejsza niż w komórce macierzystej, gamety, komórki budujące
1. Zadanie (0 2 p. ) Porównaj mitozę i mejozę, wpisując do tabeli podane określenia oraz cyfry. ta sama co w komórce macierzystej, o połowę mniejsza niż w komórce macierzystej, gamety, komórki budujące
ALGORYTMY KONSTRUOWANIA DENDROGRAMÓW STOSOWANYCH PRZY ANALIZIE FILOGENETYCZNEJ MIKROORGANIZMÓW
 ALGORYTMY KONSTRUOWANIA DENDROGRAMÓW STOSOWANYCH PRZY ANALIZIE FILOGENETYCZNEJ MIKROORGANIZMÓW Filip Zdziennicki, Anna Misiewicz Instytut Biotechnologii Przemysłu Rolno-Spożywczego im. prof. Wacława Dąbrowskiego
ALGORYTMY KONSTRUOWANIA DENDROGRAMÓW STOSOWANYCH PRZY ANALIZIE FILOGENETYCZNEJ MIKROORGANIZMÓW Filip Zdziennicki, Anna Misiewicz Instytut Biotechnologii Przemysłu Rolno-Spożywczego im. prof. Wacława Dąbrowskiego
Ocena wartości hodowlanej. Dr Agnieszka Suchecka
 Ocena wartości hodowlanej Dr Agnieszka Suchecka Wartość hodowlana genetycznie uwarunkowane możliwości zwierzęcia do ujawnienia określonej produkcyjności oraz zdolność przekazywania ich potomstwu (wartość
Ocena wartości hodowlanej Dr Agnieszka Suchecka Wartość hodowlana genetycznie uwarunkowane możliwości zwierzęcia do ujawnienia określonej produkcyjności oraz zdolność przekazywania ich potomstwu (wartość
Podstawy biologii. Informacja genetyczna. Co to jest ewolucja.
 Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Zarys biologii molekularnej genu Podstawowe procesy genetyczne Replikacja powielanie informacji Ekspresja wyrażanie (realizowanie funkcji)
Podstawy biologii Informacja genetyczna. Co to jest ewolucja. Zarys biologii molekularnej genu Podstawowe procesy genetyczne Replikacja powielanie informacji Ekspresja wyrażanie (realizowanie funkcji)
MACIERZE MUTACYJNE W ANALIZIE GENOMÓW czy możliwa jest rekonstrukcja filogenetyczna? Aleksandra Nowicka
 MAIERZE MUTAYJNE W ANALIZIE GENOMÓW czy możliwa jest rekonstrukcja filogenetyczna? Aleksandra Nowicka Zadaniem FILOGENETYKI jest : zrekonstruowanie ewolucyjnej historii wszystkich organizmów odkrycie przodka
MAIERZE MUTAYJNE W ANALIZIE GENOMÓW czy możliwa jest rekonstrukcja filogenetyczna? Aleksandra Nowicka Zadaniem FILOGENETYKI jest : zrekonstruowanie ewolucyjnej historii wszystkich organizmów odkrycie przodka
Programowanie genetyczne - gra SNAKE
 PRACOWNIA Z ALGORYTMÓW EWOLUCYJNYCH Tomasz Kupczyk, Tomasz Urbański Programowanie genetyczne - gra SNAKE II UWr Wrocław 2009 Spis treści 1. Wstęp 3 1.1. Ogólny opis.....................................
PRACOWNIA Z ALGORYTMÓW EWOLUCYJNYCH Tomasz Kupczyk, Tomasz Urbański Programowanie genetyczne - gra SNAKE II UWr Wrocław 2009 Spis treści 1. Wstęp 3 1.1. Ogólny opis.....................................
Generator testów bioinformatyka wer / Strona: 1
 Przedmiot: wyklad monograficzny Nazwa testu: bioinformatyka wer. 1.0.6 Nr testu 10469906 Klasa: 5 IBOS Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Aminokwas jest to związek organiczny zawierający A) grupę
Przedmiot: wyklad monograficzny Nazwa testu: bioinformatyka wer. 1.0.6 Nr testu 10469906 Klasa: 5 IBOS Odpowiedzi zaznaczamy TYLKO w tabeli! 1. Aminokwas jest to związek organiczny zawierający A) grupę
PODSTAWY BIOINFORMATYKI 11 BAZA DANYCH HAPMAP
 PODSTAWY BIOINFORMATYKI 11 BAZA DANYCH HAPMAP WSTĘP 1. SNP 2. haplotyp 3. równowaga sprzężeń 4. zawartość bazy HapMap 5. przykłady zastosowań Copyright 2013, Joanna Szyda HAPMAP BAZA DANYCH HAPMAP - haplotypy
PODSTAWY BIOINFORMATYKI 11 BAZA DANYCH HAPMAP WSTĘP 1. SNP 2. haplotyp 3. równowaga sprzężeń 4. zawartość bazy HapMap 5. przykłady zastosowań Copyright 2013, Joanna Szyda HAPMAP BAZA DANYCH HAPMAP - haplotypy
STATYSTYKA - PRZYKŁADOWE ZADANIA EGZAMINACYJNE
 STATYSTYKA - PRZYKŁADOWE ZADANIA EGZAMINACYJNE 1 W trakcie badania obliczono wartości średniej (15,4), mediany (13,6) oraz dominanty (10,0). Określ typ asymetrii rozkładu. 2 Wymień 3 cechy rozkładu Gauss
STATYSTYKA - PRZYKŁADOWE ZADANIA EGZAMINACYJNE 1 W trakcie badania obliczono wartości średniej (15,4), mediany (13,6) oraz dominanty (10,0). Określ typ asymetrii rozkładu. 2 Wymień 3 cechy rozkładu Gauss
Uczeń potrafi. Dział Rozdział Temat lekcji
 Plan wynikowy z biologii- zakres podstawowy, dla klasy III LO i III i IV Technikum LO im.ks. Jerzego Popiełuszki oraz Technikum w Suchowoli Nauczyciel: Katarzyna Kotiuk Nr programu: DKOS-4015-5/02 Dział
Plan wynikowy z biologii- zakres podstawowy, dla klasy III LO i III i IV Technikum LO im.ks. Jerzego Popiełuszki oraz Technikum w Suchowoli Nauczyciel: Katarzyna Kotiuk Nr programu: DKOS-4015-5/02 Dział
BUDOWA I FUNKCJA GENOMU LUDZKIEGO
 BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
BUDOWA I FUNKCJA GENOMU LUDZKIEGO Magdalena Mayer Katedra i Zakład Genetyki Medycznej UM w Poznaniu 1. Projekt poznania genomu człowieka: Cele programu: - skonstruowanie szczegółowych map fizycznych i
plezjomorfie: podobieństwa dziedziczone po dalszych przodkach (c. atawistyczna)
 Podobieństwa pomiędzy organizmami - cechy homologiczne: podobieństwa wynikające z dziedziczenia - apomorfie: podobieństwa dziedziczone po najbliższym przodku lub pojawiająca się de novo (c. ewolucyjnie
Podobieństwa pomiędzy organizmami - cechy homologiczne: podobieństwa wynikające z dziedziczenia - apomorfie: podobieństwa dziedziczone po najbliższym przodku lub pojawiająca się de novo (c. ewolucyjnie
Wykład Bioinformatyka 2012-09-24. Bioinformatyka. Wykład 7. E. Banachowicz. Zakład Biofizyki Molekularnej IF UAM. Ewolucyjne podstawy Bioinformatyki
 Bioinformatyka Wykład 7 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Plan Bioinformatyka Ewolucyjne podstawy Bioinformatyki Filogenetyka Bioinformatyczne narzędzia
Bioinformatyka Wykład 7 E. Banachowicz Zakład Biofizyki Molekularnej IF UAM http://www.amu.edu.pl/~ewas 1 Plan Bioinformatyka Ewolucyjne podstawy Bioinformatyki Filogenetyka Bioinformatyczne narzędzia
Ograniczenia środowiskowe nie budzą wielu kontrowersji, co nie znaczy że rozumiemy do końca proces powstawania adaptacji fizjologicznych.
 1 Ograniczenia środowiskowe nie budzą wielu kontrowersji, co nie znaczy że rozumiemy do końca proces powstawania adaptacji fizjologicznych. Wiadomo, że ściśle powiązane z zagadnieniem interakcji kompetencje
1 Ograniczenia środowiskowe nie budzą wielu kontrowersji, co nie znaczy że rozumiemy do końca proces powstawania adaptacji fizjologicznych. Wiadomo, że ściśle powiązane z zagadnieniem interakcji kompetencje
Dyspersja wybranych gatunków dużych ssaków RYŚ, WILK i ŁOŚ uwarunkowania środowiskowe i behawioralne
 Dyspersja wybranych gatunków dużych ssaków RYŚ, WILK i ŁOŚ uwarunkowania środowiskowe i behawioralne mgr MARCIN GÓRNY 1, mgr WOJCIECH LEWANDOWSKI 2, dr hab. RAFAŁ KOWALCZYK 1 1 INSTYTUT BIOLOGII SSAKÓW
Dyspersja wybranych gatunków dużych ssaków RYŚ, WILK i ŁOŚ uwarunkowania środowiskowe i behawioralne mgr MARCIN GÓRNY 1, mgr WOJCIECH LEWANDOWSKI 2, dr hab. RAFAŁ KOWALCZYK 1 1 INSTYTUT BIOLOGII SSAKÓW
Podstawy teorii ewolucji. Informacja i ewolucja
 Podstawy teorii ewolucji Informacja i ewolucja Podręczniki 2 Dla zainteresowanych http://wps.prenhall.com/esm_freeman_evol_4/ 3 Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego
Podstawy teorii ewolucji Informacja i ewolucja Podręczniki 2 Dla zainteresowanych http://wps.prenhall.com/esm_freeman_evol_4/ 3 Informacje Kontakt: Paweł Golik Instytut Genetyki i Biotechnologii, Pawińskiego
Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej
 Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Seminarium 1 część 1 Konspekt do zajęć z przedmiotu Genetyka dla kierunku Położnictwo dr Anna Skorczyk-Werner Katedra i Zakład Genetyki Medycznej Genom człowieka Genomem nazywamy całkowitą ilość DNA jaka
Strategie ewolucyjne (ang. evolu4on strategies)
 Strategie ewolucyjne (ang. evolu4on strategies) Strategia ewolucyjna (1+1) W Strategii Ewolucyjnej(1 + 1), populacja złożona z jednego osobnika generuje jednego potomka. Kolejne (jednoelementowe) populacje
Strategie ewolucyjne (ang. evolu4on strategies) Strategia ewolucyjna (1+1) W Strategii Ewolucyjnej(1 + 1), populacja złożona z jednego osobnika generuje jednego potomka. Kolejne (jednoelementowe) populacje
Statystyka i Analiza Danych
 Warsztaty Statystyka i Analiza Danych Gdańsk, 20-22 lutego 2014 Zastosowania analizy wariancji w opracowywaniu wyników badań empirycznych Janusz Wątroba StatSoft Polska Centrum Zastosowań Matematyki -
Warsztaty Statystyka i Analiza Danych Gdańsk, 20-22 lutego 2014 Zastosowania analizy wariancji w opracowywaniu wyników badań empirycznych Janusz Wątroba StatSoft Polska Centrum Zastosowań Matematyki -
Drzewa filogenetyczne jako matematyczny model relacji pokrewieństwa. dr inż. Damian Bogdanowicz
 Drzewa filogenetyczne jako matematyczny model relacji pokrewieństwa dr inż. Damian Bogdanowicz Sprawa R. Schmidt a z Lafayette Podczas rutynowych badań u pielęgniarki Janet Allen stwierdzono obecność wirusa
Drzewa filogenetyczne jako matematyczny model relacji pokrewieństwa dr inż. Damian Bogdanowicz Sprawa R. Schmidt a z Lafayette Podczas rutynowych badań u pielęgniarki Janet Allen stwierdzono obecność wirusa
